完成したライブラリーのクオリティチェックで、予想と異なるサイズのピークが認められたことはないでしょうか。例えば、インサート配列がないようなライブラリー(Adapter Dimer)や、インサートが極端に短いライブラリーの混入があった場合、そのままシーケンスしてよいか迷われることもあるかと思います。
本ページでは、予期せぬ構造を持つライブラリーについて、アダプターダイマーを中心に、その原因やランに及ぼす影響、および対策を解説いたします。
- アダプターダイマー(Adapter Dimer)とは
- アダプターダイマーの原因
ライブラリー調製時にアダプターダイマーが生じる原因として、例えば以下のようなことが挙げられます。
- ライブラリー調製に使用するインプットDNA/RNA量が不十分
- ライブラリー調製に使用するインプットDNA/RNAのクオリティが低い
- ビーズ精製が不十分
インプットとして使用するDNAやRNAが推奨よりも少ない場合には、過剰なアダプター同士が結合し、アダプターダイマーが形成されやすくなりますので、適切なインプットDNA/RNA量を使用すること、また、そのためのDNA/RNA量の正確な定量が重要になります。例として、インプットDNA量の定量には、Qubitなどの蛍光色素を利用したdsDNA特異的な定量法で定量いただくことをお勧めしております。ライブラリー調製キットをご使用の場合は、各ワークフローで推奨される定量方法をご確認ください。
ライブラリー調製キットによっては、インプットDNA/RNAのクオリティに基準がある場合があります。分解の進んだDNA/RNAなど、検証されていない低品質のインプットDNA/RNAを使われた場合、アダプターダイマーが形成されることがあります。
ライブラリー調製の過程において磁気ビーズ等を用いたサイズセレクションのステップがある場合、多くのワークフローでは、アダプターダイマーが生成されていたとしても、この精製ステップでほとんどが除去されると考えられます。ビーズハンドリングの方法が適切でなかった場合、最終的なライブラリーに目的のサイズ以外のライブラリーやアダプターダイマーが残ってしまう可能性があります。
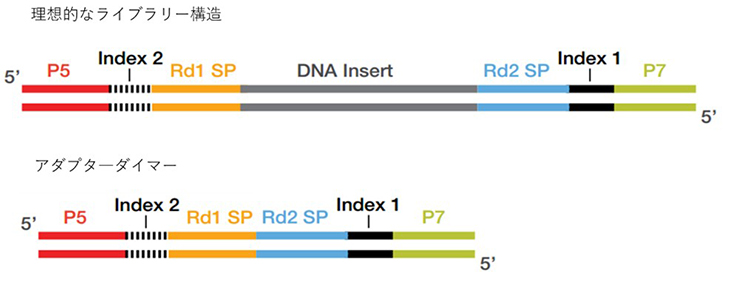
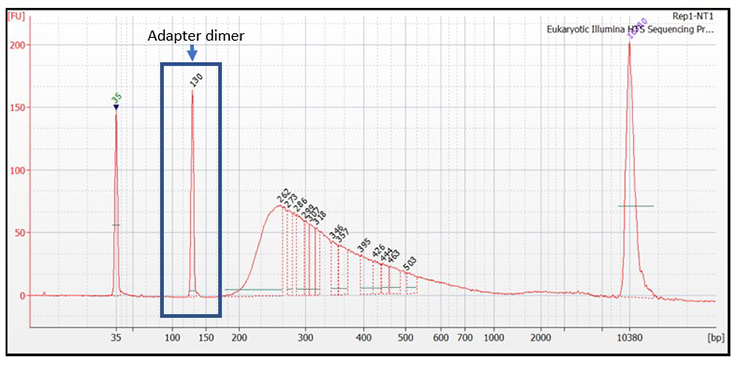
ライブラリー調製にて期待される最終的なライブラリーは、インサートDNAの両端にアダプター配列が付加した構造です。しかし、インサートDNAをもたず、アダプター配列同士が結合したライブラリーが生成されることがあり、これをアダプターダイマーと呼びます(図1)。アダプターダイマーは、BioanalyzerやFragment Analyzerなどでライブラリーのクオリティチェックをした際、120~170 bp付近にピークとして現れます(図2)。アダプターダイマーはフローセル上のオリゴに結合する配列(P5/P7)やIndex配列、シーケンスプライマーが結合する配列など、シーケンスに必要なアダプター配列の全長を持っているため、ライブラリーと同様にフローセル上でクラスターを形成します。
- アダプターダイマーを除去することの重要性
アダプターダイマーは通常のライブラリーよりもサイズが小さいため、より効率的にクラスターが形成されます。そのため、アダプターダイマー由来のリードがシーケンスデータの多くを占めてしまい、目的のライブラリー由来のデータが十分に得られない可能性があります。また、アダプターダイマーが多く含まれていると、ランの途中で塩基を読み切ってしまった場合に十分なシグナルが得られず、シーケンスクオリティが低下する可能性やフォーカスエラーによりランが途中で停止してしまう可能性がありますので、シーケンスにかける前に適切に除去することが重要です。
なお、アダプターダイマーの混入率について、iSeq™ 100, NextSeq™ 1000/2000, NovaSeq™ 6000, NovaSeq X/X Plusなどのパターン化フローセルを採用したシステムでシーケンスする場合は0.5%以下、MiSeq™, MiniSeq™, NextSeq 500/550などランダムフローセルを採用したシステムでシーケンスする場合は5%以下に抑えることを推奨しております。
- アダプターダイマーが混入した場合のラン結果
- アダプターダイマーの除去方法
- 短いインサートを持つライブラリーについて
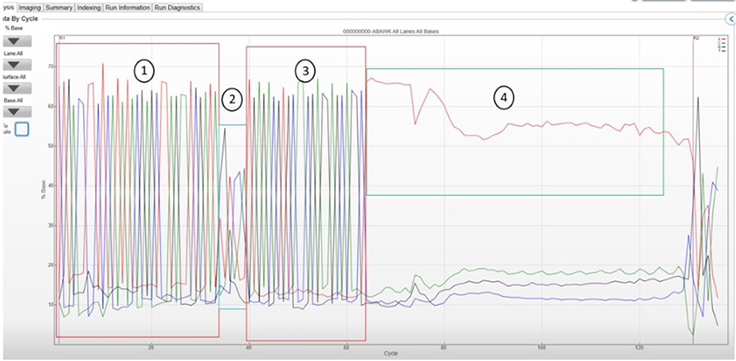
アダプターダイマーが混入しているライブラリーのシーケンス結果について、Sequencing Analysis Viewer(SAV)のData By Cycleで%Baseのプロットを表示させると図3のような特徴的な結果を示します(図3はMiSeqの例)。
調製したライブラリーにアダプターダイマーが存在する場合、追加のビーズ精製またはゲル精製を実施することで除去できます。
ビーズ精製の場合は、通常0.8x~1xのビーズ比率で、下記リンク先の「Bead handling best practices」に沿って実施することが推奨されております。なお、追加の精製を行うことで、ライブラリーの収量が減少する可能性がございますので、ご注意ください。
Bead handling best practices ≫
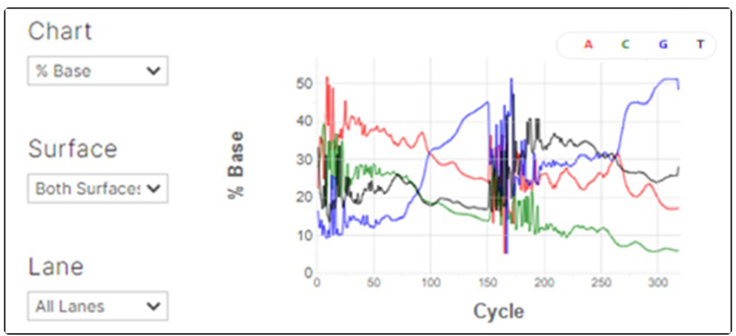
アダプターダイマー以外に、短いインサートを持つライブラリーが混入しているライブラリーのランにおいても、アダプターダイマーの場合と同様の症状が見られることがあります。想定よりも短いインサートを持つライブラリーが多く、インサートサイズよりもランで設定したリード長が長い場合、シーケンスリードの後半でライブラリーを読み切ってしまい、MiSeqの場合はAオーバーコール、その他のシーケンサーの場合はGオーバーコールが見られることがあります(図4は1色法または2色法の装置の例)。短いインサートを持つライブラリーの混入は、アダプターダイマーと同様にランのクオリティ低下やエラーによるランの中断につながる可能性がございますので、ラン前にライブラリーのサイズ分布を確認し、想定されていない短いサイズのライブラリーが含まれていないか、ご確認いただくことをお勧めいたします。
詳細は下記リンク先の「How short inserts affect sequencing performance」をご参照ください。
How short inserts affect sequencing performance ≫