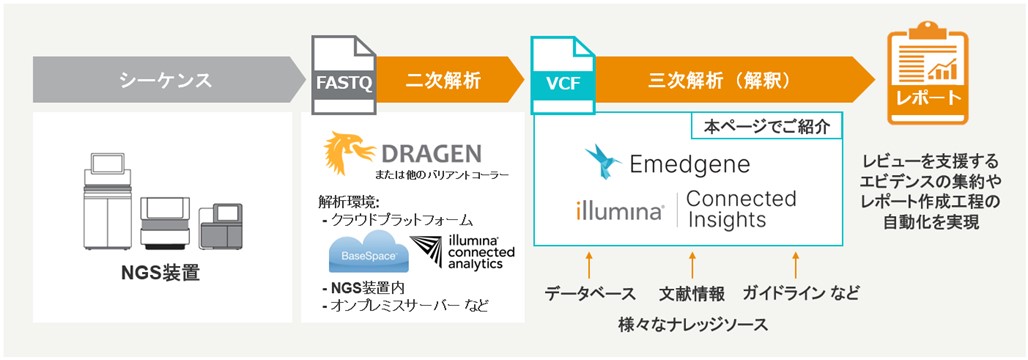
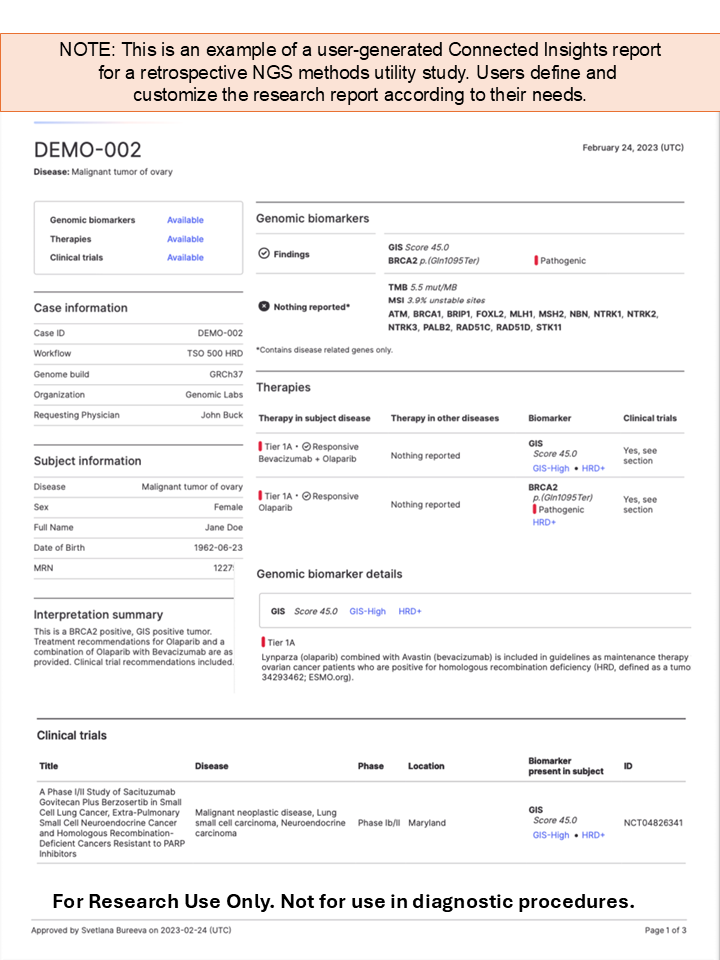
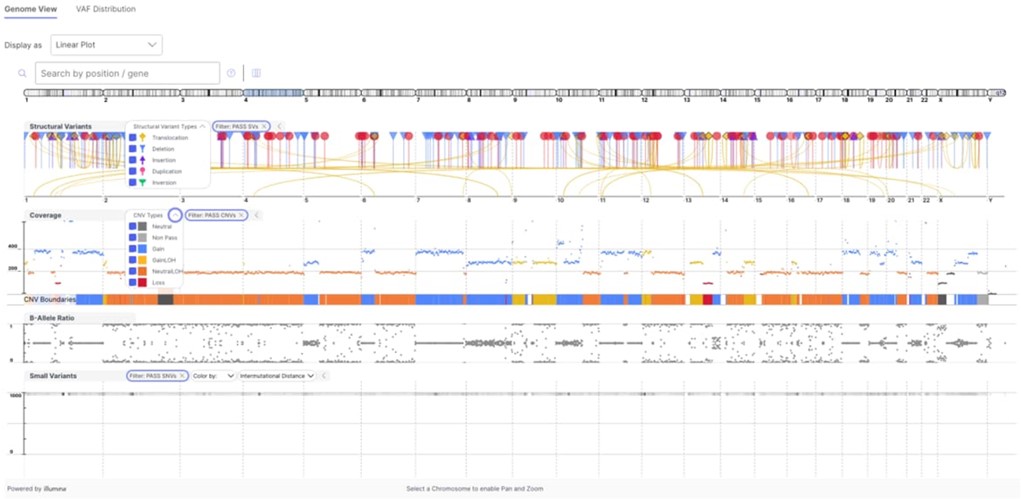
NGSを利用した遺伝子の変異解析において、イルミナDRAGENTMなどの二次解析ソフトウェアは、FASTQファイルを入力として遺伝子変異(バリアント)をコールし、VCF形式で出力します(図1)。しかし、VCFファイル内の多数のバリアントを解釈するためには、アノテーションの付与からレポート作成までに多大な労力を要し、専門的なデータ解析の技能が求められてきました。
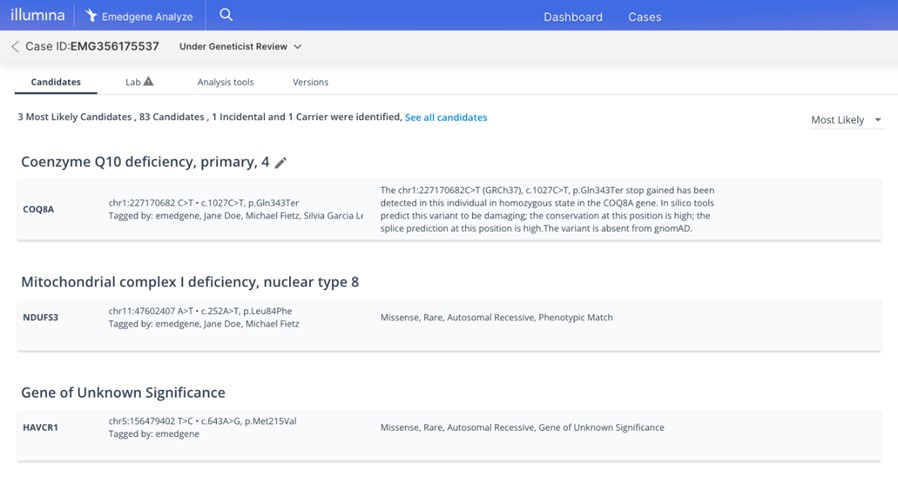
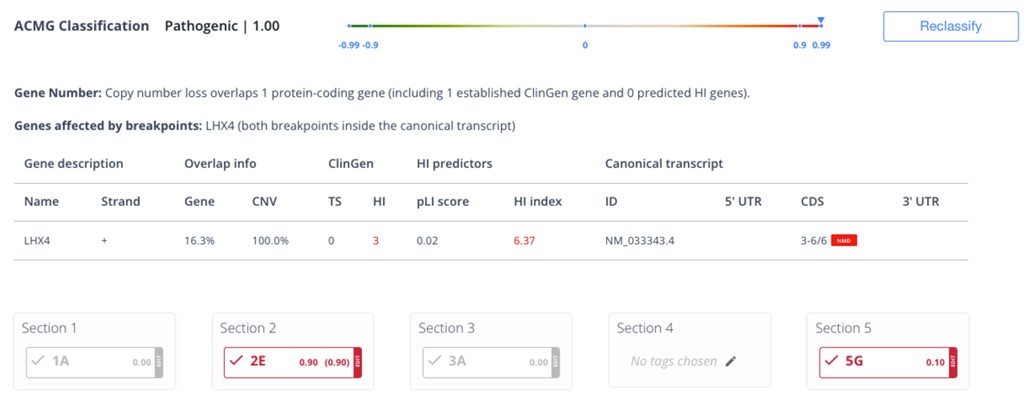
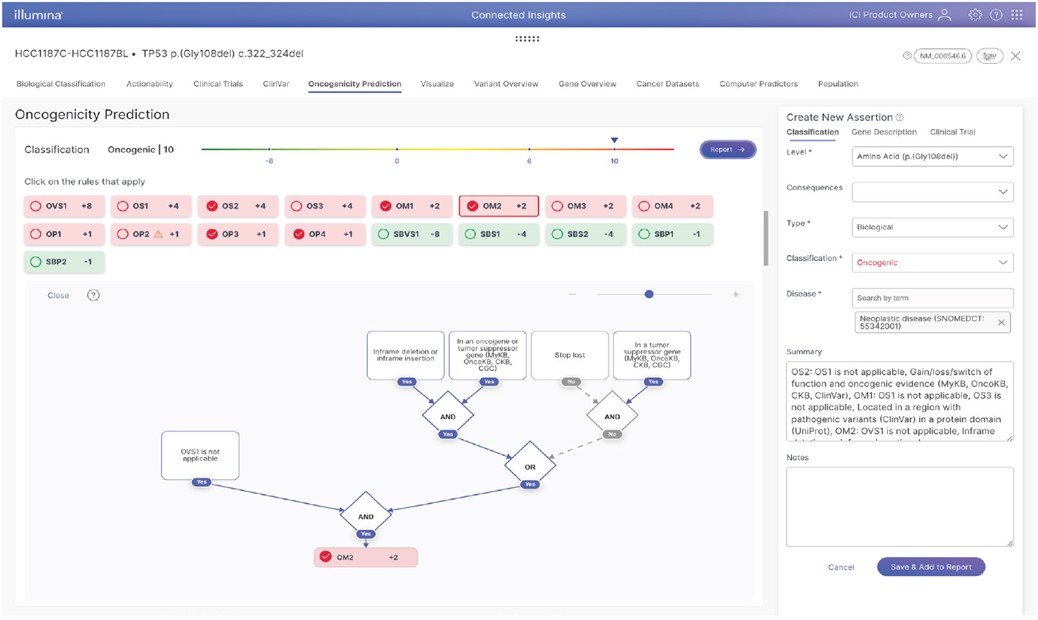
イルミナでは、バイオインフォマティクスの実務経験がない方でも、効率的かつ再現性高くヒトのバリアント解釈が行える三次解析ソフトウェアとして、「Emedgene」1,2,3 と「Illumina Connected Insights」4を提供しています(図1、表1)。これらの製品は、イルミナ発の人工知能(AI)技術(PrimateAI-3D 5およびSplice AI 6)を含む、臨床研究レベルのバリアントアノテーション(Illumina Connected Annotations)の付与から、文献情報やガイドラインをはじめとする多数のナレッジソースの集約、そしてレポート出力までの多くの工程について自動化を実現します。また、イルミナのNGS装置およびクラウドプラットフォーム(BaseSpaceTM Sequence HubやIllumina Connected Analytics)と統合することで、ヒトDNA/RNAのシーケンスから、FASTQ/VCFファイルを経由したバリアント解釈レポートの作成まで、シームレスなパイプラインとしての運用を可能にします。
本ページでは、遺伝性疾患やがん・腫瘍学(オンコロジー)分野において、臨床データの解析やゲノム医療に携わる研究者を強力に支援する、これらのソフトウェアの特長をご紹介します。
| Emedgene | Illumina Connected Insights | |
|---|---|---|
| 用途 | NGSで得られたヒトのバリアント情報(VCFファイル)のアノテーション、解釈、およびレポート作成 | |
| 分野 | 希少疾患を含む遺伝性疾患研究 | 腫瘍学(オンコロジー)研究 |
| 特長 |
|
|
表1 「Emedgene」と「Illumina Connected Insights」の特長
a. Online Mendelian Inheritance in Man(OMIM)、ClinVar、gnomADなどの国際的なデータベースに加え、日本人特有のバリアント情報を含むJapanese Multi-Omics Reference Panel(jMorp)などを参照するカスタマイズが可能。
b. 10万人を超える世界中のユーザーから信頼されているThe Jackson Laboratory Clinical Knowledgebase(JAX-CKBTM)などオンコロジーに特化したナレッジソースを含む。
各ソフトウェアの詳細・レポート例(クリックで詳細が表示されます)
参考資料
- Emedgeneパンフレット(ご利用いただいたお客様の声)
- Emedgeneデータシート
- Meng L, Attali R, Talmy T, Regev Y, Mizrahi N, et al. Evaluation of an automated genome interpretation model for rare disease routinely used in a clinical genetic laboratory. Genet Med. 2023 Jun;25(6):100830. doi: 10.1016/j.gim.2023.100830. Epub 2023 Mar 16. PMID: 36939041.
- Illumina Connected Insightsデータシート
- Gao H, Hamp T, Ede J, Schraiber JG, McRae J, et al. The landscape of tolerated genetic variation in humans and primates. Science. 2023 Jun 2;380(6648):eabn8153. doi: 10.1126/science.abn8197. Epub 2023 Jun 2. PMID: 37262156; PMCID: PMC10713091.
- Jaganathan K, Kyriazopoulou Panagiotopoulou S, McRae JF, Darbandi SF, Knowles D, et al. Predicting Splicing from Primary Sequence with Deep Learning. Cell. 2019 Jan 24;176(3):535-548.e24. doi: 10.1016/j.cell.2018.12.015. Epub 2019 Jan 17. PMID: 30661751.
略語一覧
AI: Artificial Intelligence, GIS: Genomic Instability Score, HRD: Homologous Recombination Deficiency, MSI: Microsatellite Instability, NGS: Next Generation Sequencing, VAF: Variant Allele Frequency, SOP: Standard Operating Procedures, TMB: Tumor Mutational Burden, WES: Whole Exome Sequencing, WGS: Whole Genome Sequencing, WTS: Whole Transcriptome Sequencing
上記製品の使用目的は研究に限定されます。
価格やデモンストレーションのリクエストにつきましては、下記フォームからお問い合わせください。
弊社製品について、何かご質問・ご不明な点がございましたら、テクニカルサポートまでお気軽にご相談ください。