平素はNextSeq 1000/2000をご利用いただきありがとうございます。本ページでは、NextSeq 1000/2000のシーケンスラン試薬(XLEAP-SBS)★使用上の技術的な注意点をご紹介します。
★NextSeq 1000/2000のシーケンスラン試薬(Standard-SBS)使用上の注意点については、こちらのページをご参照ください。Standard-SBSのシーケンスラン試薬は、2025年末の販売終了を予定しています。
NextSeq 1000/2000 XLEAP-SBS*1の仕様 (Specification)
Table 1: Sequencing Output Per Flow Cell
| Flow Cell Type | P1 | P2 | P3 *2 | P4 *2 |
|---|---|---|---|---|
| 1 × 50 bp | 90 Gb | |||
| 2 × 50 bp | 10 Gb | 40 Gb | 120 Gb | 180 Gb |
| 2 × 100 bp | 80 Gb | 240 Gb | 360 Gb | |
| 2 × 150 bp | 30 Gb | 120 Gb | 360 Gb | 540 Gb |
| 2 × 300 bp | 60 Gb | 240 Gb | ||
| Single-End Reads Passing Filter | 100 M | 400 M | 1.2 B | 1.8 B |
| Paired-End Reads Passing Filter | 200 M | 800 M | 2.4 B | 3.6 B |
Table 2: Quality Scores
| 1 x 50 bp | ≥ 90% of bases higher than Q30 |
|---|---|
| 2 × 50 bp | ≥ 90% of bases higher than Q30 |
| 2 × 100 bp | ≥ 90% of bases higher than Q30 |
| 2 × 150 bp | ≥ 90% of bases higher than Q30 |
| 2 × 300 bp | ≥ 85% of bases higher than Q30 |
*1: XLEAP-SBSキットのご使用には、NextSeq1000/2000 Control Software v1.7以上へのアップグレードが必要です。詳しくは本ページの「よくある質問:ソフトウェアアップデート」をご確認ください。
*2: P3およびP4キットは、NextSeq 2000のみで使用可能です。NextSeq 1000では使用できません。
*これらのNextSeq 1000/2000ランの評価には、SAV (Sequencing Analysis Viewer) v2.5.12以上が推奨です。
*オーダー番号などの情報のある、NextSeq 1000/2000用シーケンスラン試薬の製品ページはこちら(「ご注文」「XLEAP SBS reagents」と順番に選択してください)。
使用前の注意点
フローセルは2-8 ℃保存です。ランの前には、保管場所から取り出し、結露が生じないように開封せずに10-15分室温においてください。その後、室温に戻ったことを確認してから、開封してランにお使いください。
カートリッジ解凍条件(時間)はお使いのキットによって異なりますのでご注意ください。
Table 3: Summary of Thawing Methods for XLEAP-SBS kits
| Minimum Time to Complete Thaw | Do Not Exceed | Storage of Thawed Cartridge *3 | |
| 25℃ Water Bath*1 | 8 hours | 10 hours | 72 hours at 2-8°C |
| Refrigerator (2-8 ℃)*2 | 6 hours (room temp) + 16 hours (2-8 ℃) | 72 hours (2-8 ℃) | 56 hours at 2-8°C |
| Room temperature | 12 hours | 16 hours | 72 hours at 2-8°C |
*1: 解凍するカートリッジの数に関係なく、少なくとも 9.5~10 cm の水深を維持してください。完全に水中に沈める必要はありません。解凍時には、常に袋のラベルが上を向いていることを確認してください。
*2: 冷蔵庫で解凍した場合は、未開封のカートリッジを、シーケンスの前に少なくとも15分間室温で放置します。室温での放置は1時間を超えないようにしてください。
*3: “Storage of Thawed Cartridge”は、Minimum Thaw Timeの後にカートリッジを2-8℃に移動させた場合の時間です。
解凍したカートリッジは、ホイルバッグから取り出し、ゆっくり10回反転させ、中の試薬をよく混ぜます。
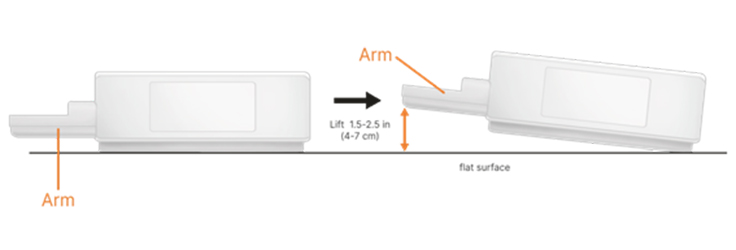
その後、カートリッジを平らな面に静置し、図のようにカートリッジのアームを平らな面から約 4~7 cm持ち上げ、ベンチに5回落として泡抜きをします。(参考動画:5秒)この操作により気泡が減少し、カートリッジのコンポーネントが安定します。カートリッジを誤って落とした場合は、このプロセスを繰り返します。
その他、カートリッジの解凍法、またフローセルの取り扱いの詳細については、NextSeq 1000/2000 Product DocumentationのXLEAP-SBS Sequencing Protocolのページをご参照ください。
よくある質問:ソフトウェアアップデート
- 各キットを使用する際に、Control Softwareのアップデートは必要ですか?
- 各キットを使用したランの解析に、Onboard DRAGEN workflowのアップデートは必要ですか?
- XLEAP-SBSキット用に、Control Softwareをv1.7、DRAGENをv4.2にアップデートした場合、Standard-SBSキットは使用できなくなりますか?
はい、XLEAP-SBSキットを使用する際にはNextSeq 1000/2000 Control Software Suite v1.7以上へのアップデートが必須になります。この時、オンライン装置についても、Auto updateを使用しないでください。必ずマニュアルで、手順書NextSeq 1000/2000 Control Software Suite v1.7.1 Customer Release Notes and Upgrade Instructionsを参照してアップデートください。
NextSeq 1000/2000 Control Software Suite v1.7は、DRAGEN v4.2とのみ互換性があるため、DRAGEN workflowのアップデートも必須です。両方のソフトウェアアップデートを同時にインストールしてください。
*さらに、DRAGEN 4.2 workflowsでの解析には、reference genomeをversion 9にアップデートする必要があります。XLEAP-SBSキットを使用する際には、一度にControl Software、Onboard DRAGEN、reference genomeをすべてアップデートすることを推奨しますが、順調にアップデートを進めても、4時間程度はかかる作業になるため、初回XLEAPラン実施の前日までに完了させておくことを強く推奨します。手順について詳しくは、手順書NextSeq 1000/2000 Control Software Suite v1.7.1 Customer Release Notes and Upgrade Instructionsをご参照ください。
いいえ。Control Software v1.7およびDRAGEN v4.2にアップデートした装置でも、Standard-SBSキットでシーケンスラン可能です。
よくある質問:XLEAP-SBSキット
- シーケンス時間はどれくらいかかりますか?
- 各キットは、インデックスを含めて最大何サイクルまでランできますか?
- P1、P2、P3、P4 キット間で、ロードするライブラリー濃度に差はありますか?
- SAV(Sequencing Analysis Viewer)を使いたいのですが、バージョンに制限はありますか?
NextSeq 1000/2000 仕様 (Specification) のページの目安をご参照ください。
全キットに、キット名のサイクル数+38サイクル分の試薬が含まれています。
キット間で最適なローディング濃度が異なる場合がありますので、ライブラリーごとにロード濃度の最適化を行うことを推奨します。一般には、P1およびP2の300 cycle以下のキットは、Standard-SBSキットと同じローディング濃度、P3およびP4、またP1およびP2の600 cycleキットについては、Standard-SBSキットのローディング濃度から25%程度低い濃度から検討をスタートできます。また%DuplicateやCoverageなどの二次解析メトリクスもご参照いただけます。詳しくはこちらのページをご参照ください。
*検討の結果、全キットで同じローディング濃度が最適となることもあります。
*上記はXLEAP-SBSキットのみに関するご案内です。Standard-SBSキットについては、こちらのページをご参照ください。
SAV version 2.5.12以上が必要です。SAV v2.5.12は、NextSeq 1000/2000装置上ではなく、別PC(Windows 10以降)での使用を想定しています。SAV v3.0は、Windows OS用、Mac OS用、Linux用をご用意していますが、現在の Linux用 SAV v3.0は Enterprise Linux バージョン (EL8+) のみと互換性があります。NextSeq 1000/2000 は現在CentOS 7(EL7)を使用しており、互換性がありません。
よくある質問:その他、XLEAP-SBSキットとStandard-SBSキットとの違い
- Reagent Cartridgeサイズが大きくなりますので、保管スペースにご注意ください。全XLEAP-SBSキットのReagent Cartridgeサイズは、Standard-SBS 600 cycle キットのReagent Cartridgeと同じサイズです。
- 変性希釈手順が一部変更されています。Denature and Dilute protocol generatorをご参照ください。
- RTAがバージョン3からバージョン4に変更され、Q-Scoreのbinningが変更されています。詳しくは、こちらのページをご参照ください。
- BlueレーザーとGreenレーザーによる2色法であることには変更ありませんが、塩基の割り当てが変更されています。XLEAP-SBSでは塩基CがBlue/Greenの両チャンネルポジティブ、塩基AがBlueチャンネルのみポジティブです。詳しくはこちらをご覧ください。
(*Standard-SBSでは塩基AがBlue/Greenの両チャンネルポジティブ、塩基CがBlueチャンネルのみポジティブでした。詳しくはこちらをご覧ください。)
- 上記の変更により、少ないサンプル数のランの際、最適なIndex組み合わせに変更があります。必ず、Adapter Pooling Guideの、[XLEAP-SBS Chemistry]と付記されている方の組み合わせをご参照ください。すべてのサイクルにおいて、Blue/Greenの両方にポジティブシグナルが存在することが理想となります。
- XLEAP-SBSカートリッジには、Illumina DNA PCR-Free Library Prep 用のシーケンスプライマーがあらかじめ含まれていますが、Standard-SBS カートリッジには含まれておりません。
- その他XLEAP-SBSキットでの変更点について詳しくは、こちらのページご参照ください。
*参考ページ (1): NextSeq 1000/2000 Documentation
*参考ページ (2): NextSeq 2000 Sequencing System specifications (XLEAP-SBS)
*参考ページ (3): [Knowledge Database] NextSeq 1000/2000