De Novoシーケンス
De Novoシーケンスとは?
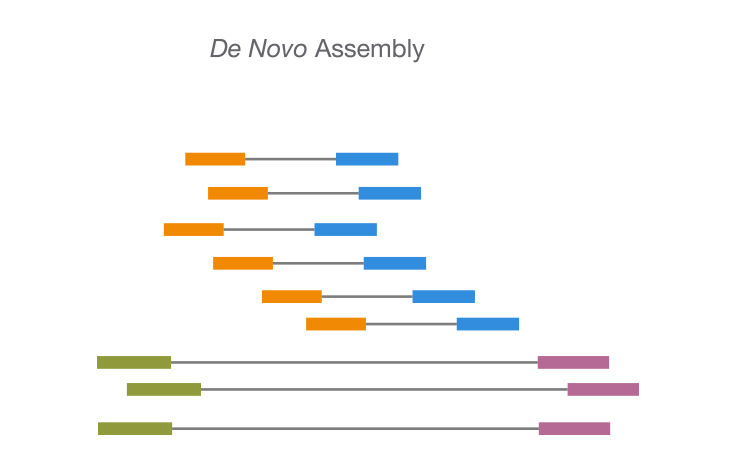
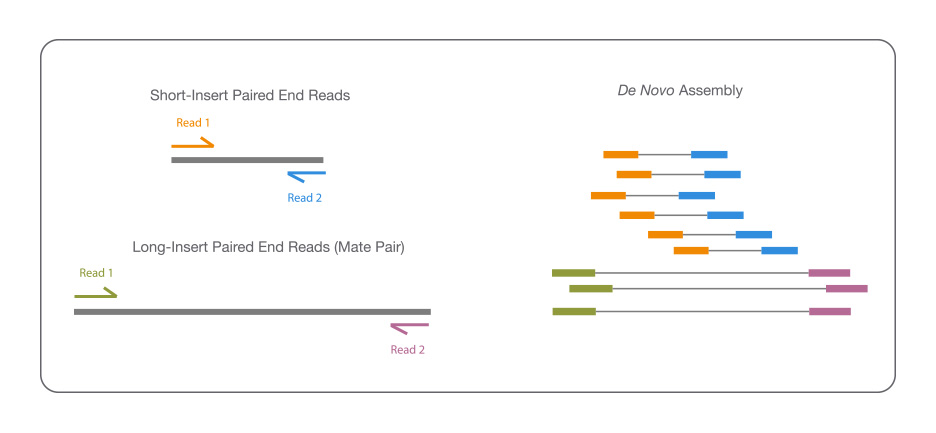
De novoシーケンスとは、アライメントに利用できるリファレンスシーケンスがない新規ゲノムのシーケンスを指します。シーケンスリードはコンティグとして組み立てられ、de novoシーケンスデータのカバレッジ品質は、コンティグのサイズと連続性(つまり、データ内のギャップ数)に依存します。
次世代シーケンサー(NGS)は、サンガーシーケンスなどの従来の手法と比較して、あらゆる種のより高速で正確な特性評価を可能にします。イルミナのNGSテクノロジーは、あらゆる種の迅速で包括的かつ正確な特性評価を提供します。
De Novoシーケンスの利点
- 複雑なゲノムや倍数体ゲノムでも正確なリファレンスシーケンスを生成
- 新規生物のゲノムマッピングや既知の生物のゲノム仕上げに有用な情報を提供します
- 非常に類似している領域や反復する領域を明確化し、正確なde novoアセンブリを実現
- 構造的バリアントや、欠失、逆位、転座などの複雑な再構成を同定

注目のDe Novoシーケンス記事
コロナウイルスの特性化と管理に役立つイルミナシーケンサー
New England Journal of Medicine誌に発表されているように、イルミナのNGSが、他のテクノロジーと組み合わせて、新型コロナウイルスゲノムのde novoシーケンスと特性評価にどのように使用されたかをご覧ください。
記事を読むCOVID-19感受性の遺伝学的特徴
イルミナは、COVID-19の重症患者と軽症患者のゲノムを比較するための、Genomics Englandが主導した全英調査に、シーケンスを提供しています。この研究は、感受性に関連する遺伝的要因を明らかにするのに役立つ可能性があります。
記事を読む
De Novo細菌シーケンスの推奨ワークフロー
包括的なDe Novoゲノムシーケンスワークフロー
イルミナのSequence by Synthesis(SBS)ケミストリーは、最も広く採用されている次世代シーケンサー(NGS)技術で、世界のシーケンスデータの約90%を生み出しています。*
イルミナは、業界をリードするデータ品質に加えて、ライブラリー調製からデータ解析まで、de novoシーケンスを簡素化する統合ワークフローを提供しています。
以下のをクリックしてワークフローの各ステップに対応する製品をご覧ください。
ヒトWGS、de novo微生物ゲノムアセンブリ、腫瘍/正常バリアントコーリングなどの高感度アプリケーション向けの優れた使いやすさ、均一なカバレッジ、高精度データ。
ロングリードテクノロジーの詳細と、de novoアセンブリサービスからゲノムフェージング、構造バリアント検出まで、幅広いパートナーシップ製品をご覧ください。
迅速で使いやすく、手頃な価格のシーケンスで、微生物種の均一なカバレッジとゲノムアセンブリを実現します。
MiSeqシステムターゲット化した小規模ゲノムシーケンスのためのスピードと簡潔性。
NovaSeq 6000システム拡張性のあるスループットと柔軟性により、ほぼすべてのゲノム、シーケンス手法、プロジェクト規模に対応します。
シーケンスプラットフォームを比較して、それぞれのラボやアプリケーションに最適なシステムを特定できます。
シーケンス試薬イルミナのシーケンスシステムに適したシーケンス試薬、フローセル、バッファーが含まれるキットを検索できます。
シングルセルおよび分離ゲノムに適したDe novoアセンブラ。
BaseSpace Velvet De Novo Assemblyアプリベルベットアセンブラを使用した細菌ゲノムのde novoアセンブリ。
統合的ゲノミクスビューア複雑なバリアント解析を実行するための複数のサンプルからのアライメントとバリアントを表示します。
NGSデータの超高速二次解析を実行します。
BaseSpace Sequence HubNGSデータ解析と管理を行うイルミナのゲノミクスコンピューティング環境。
関連ソリューション
NGS企業の選択

使いやすいバイオインフォマティクスツールと業界をリードするサポートとサービスで、クラス最高の次世代シーケンス企業を探してください。エビデンスをご覧ください。
微生物シーケンス
微生物全ゲノムシーケンスは、新規生物のゲノムマッピング、既知の生物のゲノム仕上げ、または複数のサンプルにわたるゲノムの比較のための重要なツールです。微生物WGSの詳細はこちら。
がん全ゲノムシーケンス
腫瘍サンプルの全ゲノムシーケンスは、がん組織における固有の変異を包括的に把握し、がん遺伝子、腫瘍抑制因子、その他のリスク因子の解析に情報を提供します。がんWGSの詳細はこちら。
メタゲノム
ショットガンメタゲノムシーケンスにより、微生物学者は細菌の多様性を評価し、解析が困難または不可能な培養不可能な微生物を研究することができます。メタゲノムシーケンスの詳細はこちら。
動植物シーケンス

デノボシーケンスは、植物や動物の機能や環境相互作用に関する洞察を提供します。一部の研究者は、アセンブリされたゲノムを使用してマップ位置を割り当て、その後のSNP発見のために多様な品種情報をスタックします。植物と動物のシーケンスの詳細はこちら。
Interested in receiving newsletters, case studies, and information on genomic analysis techniques? Enter your email address.
*社内計算データ Illumina, Inc., 2015