クロマチン免疫沈降シーケンス(ChIP-Seq)
ChIP-Seqとは。
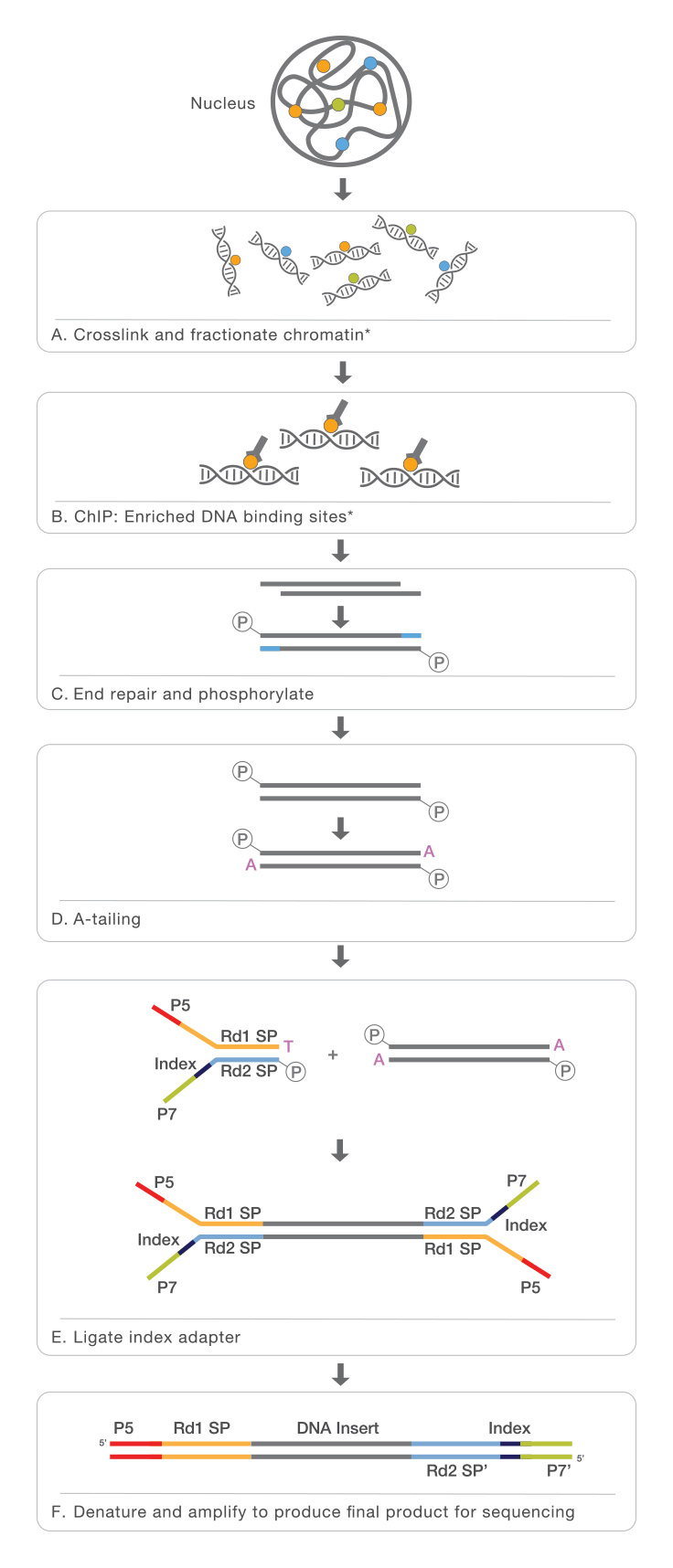
クロマチン免疫沈降(ChIP)アッセイとシーケンスを組み合わせたChIPシーケンス(ChIP-Seq)は、転写因子やその他のタンパク質のゲノム全体のDNA結合部位を同定するための強力な手法です。ChIPプロトコールに従い、DNA-結合タンパク質は特異的な抗体を用いて免疫沈降されます。その後、結合したDNAを共沈させ、精製し、シーケンスされます。
次世代シーケンサー(NGS)のChIPへの応用により、発生やがんの進行など様々な疾患や生物学的パスウェイに関与する遺伝子制御事象の洞察が得られています。ChIP-Seqにより、ゲノムワイドのタンパク質と核酸の相互作用を徹底的に調べることができます。
ChIPシーケンスのメリット
エピゲノム研究に用いられるアレイなどのようなアプローチは、既知のシーケンスを由来とするプローブを必要とするため、本質的にバイアスがかかりますが、ChIP-Seqは事前知識を必要としません。ChIP-Seqは、超並列シーケンスによる全ゲノムプロファイルを提供し、コスト効率よく、正確に、また偏りなく複数のサンプルで数百万のカウントを生成し、エピジェネティックなパターンを調査することができます。さらに、以下のようなメリットもあります。
- あらゆる生物の全ゲノムにわたり、転写因子やヒストン修飾のターゲットとなるDNAを捕捉
- 転写因子結合部位を定義
- RNAシーケンスとメチル化解析との組み合わせで、遺伝子制御ネットワークを明らかにする
- 様々な入力DNAサンプルに対応
ChIP-Seqの推奨されるワークフロー
データの解析と解釈
BaseSpace ChIPSeqアプリ
MACS2を用いて転写因子結合部位を同定し、HOMERを用いてピーク内のモチーフを発見します。
Partek Flow
ChIP-Seq解析結果をRNA-Seqデータに統合することで、ピークの検出とアノテーションを行い、サンプルグループ間の比較、濃縮された遺伝子の同定、領域からのモチーフの発見、遺伝子発現に対する転写因子の制御効果の探索を行います。
包括的ChIPシーケンスワークフロー
イルミナは、ライブラリー調製からデータ解析および解釈まで、クロマチン免疫沈降シーケンスを簡素化する統合ワークフローを提供しています。
ChIP-Seqは、高度にターゲット化された転写因子では数リード(約500~1,500万)、ヒストンマーキングプルダウンなどのユビキタスタンパク質ではそれ以上のリード(約5千万)を必要とする場合があります。
以下のをクリックしてワークフローの各ステップに対応する製品をご覧ください。
ChIP由来DNAからChIP-Seqライブラリーを調製するためのシンプルでコスト効率の高い手法です。
画期的なベンチトップシーケンサーは、現在および将来のさまざまなアプリケーションにおいて、より高い効率性と少ない制約で新しい発見を探索することを可能にします。
NovaSeq 6000システム拡張性のあるスループットと柔軟性により、ほぼすべてのゲノム、シーケンス手法、プロジェクト規模に対応します。
イルミナのシーケンスプラットフォームを比較し、ラボやアプリケーションに最適なシステムを特定します。
シーケンス試薬イルミナのシーケンスシステムに適したシーケンス試薬、フローセル、バッファーが含まれるキットを検索できます。
MACS2により転写因子結合部位を同定し、HOMERによりピーク内のモチーフを発見。
BaseSpace Sequence HubNGSデータ解析と管理を行うイルミナのゲノミクスコンピューティング環境。
疾患メカニズム、薬剤ターゲット、バイオマーカーの同定で研究者をサポートする、キュレーションされたゲノムデータの発展し続けるライブラリー。
Partek FlowChIP-Seq解析結果をRNA-Seqデータに統合することで、ピークの検出とアノテーションを行い、サンプルグループ間の比較、濃縮された遺伝子の同定、領域からのモチーフの発見、遺伝子発現に対する転写因子の制御効果の探索を行います。
関連ソリューション
がんエピジェネティクス
メチル化異常や転写因子の結合の変化など、がんにおけるエピジェネティクスの変化の研究は、重要な腫瘍パスウェイにおける洞察をもたらすことがあります。がんエピジェネティクスの詳細はこちら。
遺伝子発現解析
遺伝子発現研究は、ゲノムや環境の変化が様々な疾患にどのように寄与しているかを視覚化することができます。遺伝子発現のプロファイル方法を学ぶ。
クロマチンアクセシビリティ解析

ATAC-Seqは、ゲノム全体のクロマチンアクセシビリティを決定するための定評のある手法です。その後の実験では、ChIP-Seq、Methyl-Seq、またはHi-C-Seqなどがよく行われます。ATAC-Seqの詳細はこちら。
メチル化シーケンス

NGSを用いたメチル化シーケンスのアプローチには、一塩基レベルでのメチル化パターンのプロファイルが可能であるなど、多くの利点があります。メチル化シーケンスの詳細はこちら。
注目の論文
細胞増殖制御とリンパ腫発生における、Mycによる選択的な転写制御。
研究者らは、イルミナシーケンスを使用して、B細胞リンパ腫の発生過程における全ゲノムのクロマチン免疫沈降とRNA発現プロファイリングを行います。
論文を読むコレラ菌ヒストン様ヌクレオイド構造化タンパク質(H-NS)の、RNA-Seq解析と全ゲノムDNA-結合プロファイル。
著者たちは、イルミナシーケンスによる差動RNA-seqと、抗FLAG M2モノクローナル抗体を用いたクロマチン免疫沈降シーケンスを使用しました。
論文を読むChIP-Seqとマイクロアレイで明らかになった、Sox9が制御するがん特異的遺伝子ネットワーク
転写プロファイルと生体内ChIP-Seq研究により、Sox9が制御するがん特異的遺伝子ネットワークが、腫瘍の発生と浸潤を結びつけることを明らかにしました。
論文を読むゲノム解析技術に関する最新のニュース、事例研究、情報を提供します。ぜひご登録ください。電子メールアドレスをご入力の上、ご登録ください。
補足資料

ERα Binding部位のChIPシーケンスプロファイル
Radboud Universityの研究者らが、エストロゲン受容体αの結合部位を解析するためのChIP-Seq法について述べています。