ミドリムシ、オオムギなど、多様な生物の産業応用をゲノム解析で加速する
植物のゲノム情報解析や計算モデルを用いた表現型予測、微細藻類による物質生産に向けたゲノム編集など、さまざまな研究手法を駆使する理化学研究所の持田先生に研究の現状と展望を伺いました。
Q. まず現在の研究内容をご紹介ください。
A. ミドリムシからの燃料の生産性の向上を目指す研究と、高等植物の有用遺伝子の探索などを行っています。
理化学研究所では、2つの組織に所属して研究を行っています。1つはバイオ燃料や健康食品として有用なミドリムシなどの微細藻類の生産性を向上するための研究で、使いやすい微細藻類を育種するのが大きな目標です。もう1つは、環境資源科学研究センターにおける高等植物の研究です。こちらでは、大規模なゲノムワイド関連解析(GWAS)やトランスクリプトーム解析、計算モデルを用いた農業形質の予測方法の開発などを行い、麦類などの作物の生産性の向上に有用な遺伝子の探索を行っています。
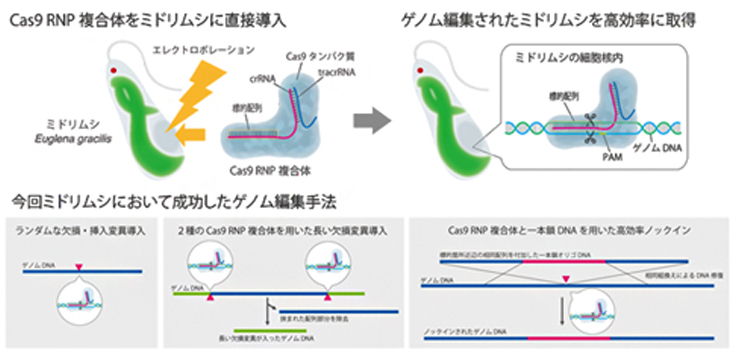
昨年、Cas9 RNP複合体を導入することによるゲノム編集に、ミドリムシでは世界で初めて成功しました。ミドリムシにはパラミロンという顆粒があり、これは好気条件下ではバイオプラスチックの材料や機能性食品となる多糖として蓄積されます。一方、嫌気条件下では、エネルギー獲得のため、パラミロンは油脂のワックスエステルに変わります。さまざまな条件を設定し、このパラミロン合成に関わる酵素の遺伝子EgGSL2(Euglena gracilis Glucan Synthase-Like 2)の機能を阻害することを試みました。エレクトロポレーション法でCas9 RNP複合体を導入した結果、最適の培養条件では導入後72時間で80〜90%という高効率でゲノム編集を行えるようになりました。さらに、2種類のCas9 RNP複合体を用いたDNAの特定領域への長い欠損変異の導入、そして、一本鎖のドナーDNA分子のノックインも可能にしました。
これまでミドリムシのゲノム編集はうまくいかないということになっていました。幸い、我々のチームには、コケ植物のゲノム編集技術を開発したことがある研究者がいたこともあり、効率の良いゲノム編集がミドリムシでもできるようになりました。
ミドリムシをはじめとする有用な微細藻類の産業利用を加速するために、弊所のバトンゾーン研究推進プログラムでは、(株)ユーグレナとの連携チームとして微細藻類生産制御技術研究チームを設置して、油脂等の生産性を向上するための研究開発を行っています。そこでは、ミドリムシを用いた物質生産に有用な遺伝子の候補について、ゲノム編集によって機能を検証し、目的とする物質の生産との関係を調べています。
ゲノム編集の結果を迅速に確認するため、イルミナのiSeq 100™を導入しました。サンガー法で個々に調べるのではなくて、ある細胞の集団をそのまま解読するハイスループットアッセイに使い勝手がいいだろうと考えたからです。コンパクトで、かつハンドリング時間が短く、結果がスピーディーに出るのが魅力で、ゲノム編集ができたかどうかすぐに知りたいというような状況でも使えます。また、誰が使っても安定した結果が出るのも助かります。
Q. 高等植物における遺伝子探索のご研究を教えてください。
A. 麦類と近縁の草本モデル植物のミナトカモジグサをつかった遺伝子探索や、オオムギ集団から農業形質に関連する遺伝子と生育条件の関係を調べています。
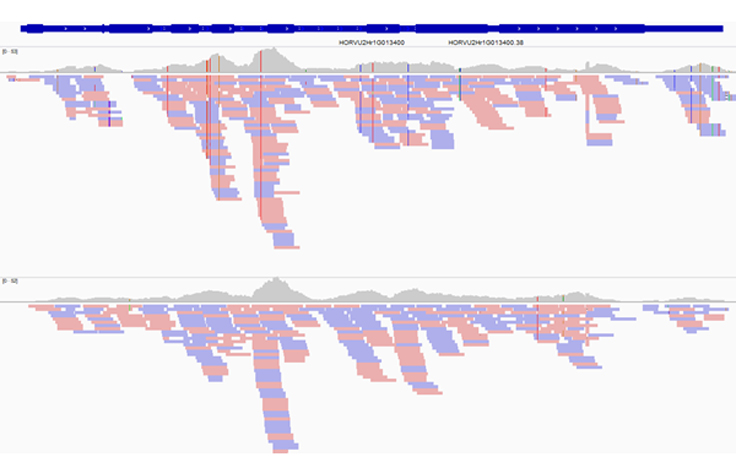
ミナトカモジグサのようなモデル実験植物やオオムギ集団を中心にゲノムワイドな多型やトランスクリプトームを解析しています。イルミナ社のNovaSeq™ 6000やHiSeq™ 4000などによる解析を受託会社に依頼しています。集団ワイドなサンプルを一度に解読することもあるので、NovaSeq 6000の使い勝手がよいという印象です。
最近、科学技術振興機構(JST)の戦略的創造研究推進事業CREST「環境変動に対する植物の頑健性の解明と応用に向けた基礎基盤の創出」研究領域の「データ科学に基づく作物設計基盤技術の構築」(研究代表者:岡山大学 資源植物科学研究所 平山隆志教授)にも注力しています。私たちの研究室は、オオムギ集団の多型解析や成長ステージごとのトランスクリプトーム解析といった多検体の解析を担当しています。
岡山大学資源植物科学研究所には、世界各地から収集されたオオムギ系統が保存されていて、出穂日などの形質データが長年にわたって集められています。私たちはそのデータを利用させていただき、ゲノムの多型と表現型の関連性や環境との相互作用の影響を調べています。
出穂日の早晩に関連する遺伝子は、オオムギでもよく調べられているものもあり、そのような遺伝子の多型と出穂形質との関連性を調べてきました。
次の研究として、まだ機能がわかっていない遺伝子もターゲットにしようということで、GWASによる遺伝子探索も進めています。
ゲノムワイドなエクソーム解析を行うときは、NovaSeq等による受託解析を利用することが多いですが、急ぎでエクソームの多型データを確認したいオオムギ2系統について、iSeq 100でシーケンシングを行ったことがあります。オオムギのような高等植物のエクソーム解析をiSeq 100で実施するには、複数のフローセルカートリッジを使う必要がありました。しかし、短期間に追試データを得る必要がある場合などは、iSeq 100が時間的コストの低減に役立つと思っています。
また、意図したとおりにライブラリーが作成できているかを小規模なサンプルセットで簡便に確認できるので、新しいライブラリー作成方法を試す場合や、サンプル全体を解析する前に作成したライブラリーを確認するのに便利だと思います。
中型機種のNextSeq™ 2000が登場したと伺っています。まずは作製したライブラリーの確認などをiSeq 100で行い、うまく行っていればNextSeqでさらに全体を解析するのは良い使い方だと思います。
Q. 計算モデルを用いた表現型の予測作成を研究に採り入れられた理由を教えてください。
A. 植物のゲノムと環境の相互作用によって形作られる農業形質を、予見性をもって改良できた方が、効率が良いと思うからです。
植物の成長に関わる遺伝要因や環境要因の中から、目的とする農業形質の改変に有用な遺伝要因を見つけ出し、環境要因との相互作用の効果や形質との関係性を計算モデルとして記述して、環境要因や遺伝要因から形質を予測することができれば、より良い遺伝要因の組み合わせを探索しやすいと思います。
また、出穂や収量といった農業形質の多くは、作物の成長の後半で顕在化するものが多いので、それまでの成長ステージにおける作物と環境の相互作用の積み重ねが、最終的な農業形質の良し悪しにどう影響しているかを理解することが重要だと考えています。近年、コンピュータビジョンやセンサーの分野も発展していて、植物の成長具合や生理状態を非侵襲かつ経時的に計測する技術の開発が進んでいます。
作物の成長過程においても、遺伝子型に応じた生理状態の多様性が計測され、表現型データとして収集されていくと考えられます。そういった表現型の背景にあるゲノムやトランスクリプトームの多様性と最終的な農業形質との関連性の知見を作物の改良に役立てるためにも、計算モデルの有用性は増していくと考えています。ゲノムやトランスクリプトームの多様性のデータを網羅的に収集するといった研究リソースの整備にはNovaSeq 6000のような大型のシーケンサーが有用だと思います。一方、目的とする農業形質の改変に有用な遺伝子のセットを決めることができれば、iSeq 100のような小回りの利くシーケンサーがアンプリコンベースの多型解析や発現解析に有用だと思います。