PCR法を超える、セルフリーの長鎖DNA増幅技術を開発
DNAの構築や増幅には、長年、大腸菌を用いたクローニング法、そしてPCR法が使われています。立教大学理学部生命理学科准教授の末次正幸先生が2017年に開発された、試験管内で環状DNAの増幅を行う方法が今、注目を集めています。この新しいDNA増幅法の特徴や応用について末次先生に聞きました。
Q. 先生が開発された新しいDNA増幅法について教えてください
A. 大腸菌がゲノムを増幅するサイクルを模したもので、試験管内で環状DNAの増幅が繰り返します
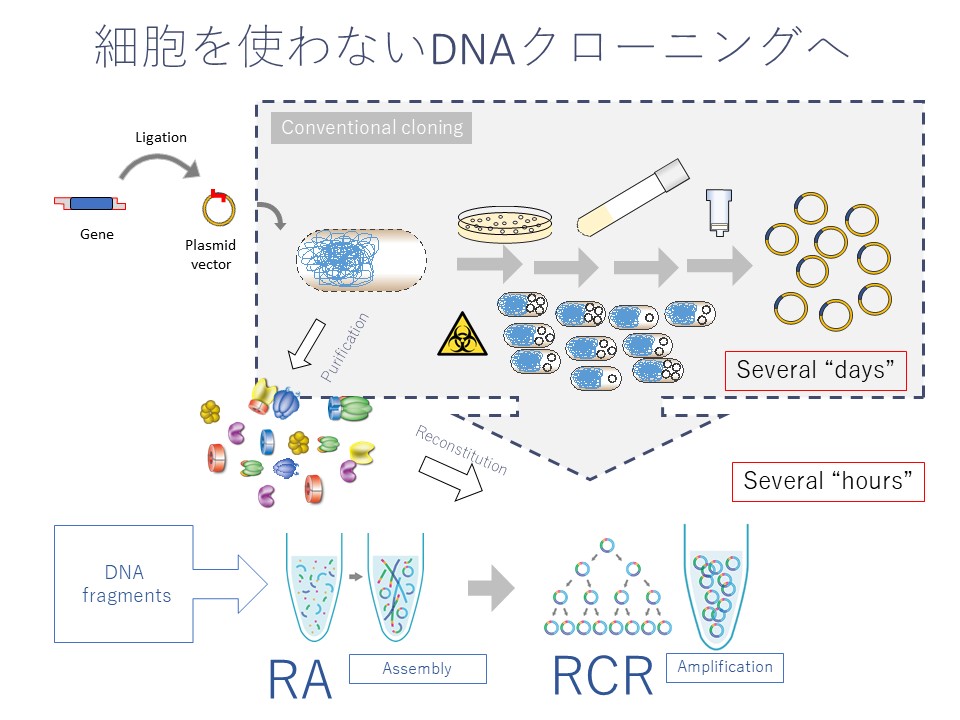
細胞を使わず、試験管内でゲノムスケールの長鎖DNAを複製でき、さらにそれを自律的に繰り返すことができるシステムを2017年に構築し、「複製サイクル試験管内再構成系(Replication Cycle Reaction :RCR)」 と名付けました(図版 1、関連論文 1)。
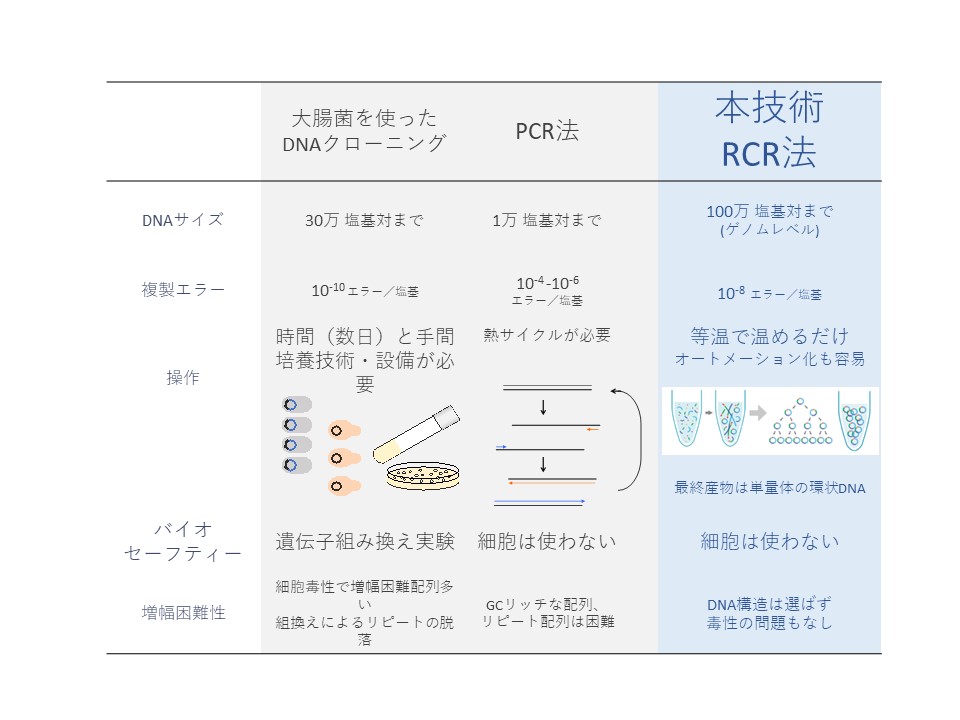
RCR法は、大腸菌の約500万塩基対のゲノムを複製するサイクルを模したもので、25 種類のタンパク質を用います。鋳型DNAの必要条件は、環状であること、また、そこにDNA複製開始起点のoriCが含まれていることです。それさえ合致すれば、配列を選ばず、20万塩基対といった、PCR法の10倍以上の、長大な環状 DNA を増幅できます。
また、RCR法の前段階でゲノムをデザインして、多断片を同時に連結する方法(Recombination Assembly:RA法)も独自に開発しました。
いずれも、長年手がけてきた大腸菌や枯草菌などの、ゲノム複製に関する基礎研究が、科学技術振興機構(JST)の革新的研究開発推進プログラム(ImPACT)野地プログラムで、新しい技術に発展したものです。
Q. RCR法を開発された理由は何でしょうか
A. 「生きている=増える」現象を自分で作りたいと思ったからです
もともと「“生きている”とはどういうことか」、「生命と非生命の違い」に興味を持っていました。大学で研究を始めるまでは、「生きている=動いていること」などと思っていましたが、研究を始めてからは、「増えるか、増えないか」が生きていることの本質と考えるようになりました。
培地の上で微生物が増えていくのを顕微鏡で観察しながら、こういう現象を自分で作りたいと思うようになりました。細胞の自己複製能を生体分子を再構成して人工的に作れれば、ある意味、生き物を作れたということであり、「生命とは何か?」という問いに答えられるのではないかと。
最初にやるべきことは、生命の設計図であるゲノムDNAの複製の再現であると目標を定めました。そして、米国のArthur Kornberg(アーサー・コーンバーグ)博士のグループが1984年に発表した、環状DNAとタンパク質を用いてゲノム複製系を試験管内で再構成する方法を参考にしました。コーンバーグはDNA合成酵素を単離し、「リボ核酸およびデオキシリボ核酸の生合成機構の発見」で1959年にノーベル生理学・医学賞を受賞しています。彼がこの再構成系を構築したのは、Kary Banks Mullis(キャリー・バンクス・マリス、1993年にノーベル化学賞を受賞)がポリメラーゼ連鎖反応(PCR)の論文を出す3年前です。このコーンバーグの系は複製反応が一部進行するだけで、未完成の系のままでした。私はこの系の完成をゲノム増幅の第一歩と考え、大腸菌をお手本に改良していきました。
Q. RCR法の開発にあたって苦労されたのはどのような点でしょうか
A. 参考にしたアーサー・コーンバーグの系の再現、さらには必要なタンパク質の組み合わせを見つけるのに時間と手間がかかりました
完成までは泥臭い仕事でしたね。まずコーンバーグの系を再現するだけでも10数種類の精製されたタンパク質が必要です。そして、精製したタンパク質を用意しても、組み合わせたときに1種類でも失活していたらDNAは複製できない。DNA分解酵素のコンタミネーションも大きなリスクです。
この系を基盤に、ゲノム複製の開始 → 伸長 → 終結 → 分離のプロセスが自律的に進行していくための必要十分条件を探しました。
立教大学でラボを立ち上げてから2年くらいは、ゲノムが増える気配がなくて焦りました。そういう時は、タンパク質をもう一度精製し直したり、精製法を変えたりし、また、タンパク質1個1個の組成、濃度、DNA分解酵素の量、試薬、反応温度を丁寧に検討しました。組み合わせが無限にあって大変でした。それで、最適と思われる25個のタンパク質の組み合わせをようやく見つけました。
そもそも、ゲノム複製のプロセスを完全に再現できたからといって、それがDNA増幅に結びつく保証はありません。増幅のためには、複製終結後のDNAがさらに次のラウンドのサイクルに進行する必要があります。つまり、複製サイクルが自律的に何度も繰り返されなければなりません。このようなサイクルの繰り返しは、転写、翻訳、さらには細胞周期機構といったイベントが必要であるというのが、以前の複製研究から考えられていたものでした。私自身、そのような繰り返しのための因子を見出すことが、学術的な新しさになると期待して始めた研究でした。ところが、実際に完成してみると、そんな因子は不要で、複製サイクルに直接機能する酵素反応のみで勝手に複製を繰り返すことがわかって驚きました。生命の原始的な状態では細胞周期はなくて、勝手に増えていく。生命って案外適当なんだな、と感じたのを覚えています。
Q. RA法+RCR法によるゲノム合成には、どんな特徴がありますか
A. 長いDNA鎖をデザインして合成できること、作業が簡便で速いことがメリットです
このシステムのポイントは、DNA増幅法として優れている点です(図版 2)。
ゲノムスケールの長鎖DNAを人工合成するためには、大腸菌や酵母などの生き物を使って、莫大な数の短いDNA断片をつなぎ合わせていくステップが必要です。
1973年に登場した大腸菌にDNAを入れて増やすDNAクローニング法は、大腸菌にとって毒になるものは増やせません。また、手間と数日の時間がかかり、自動化が難しい。
長いDNAを得たいときには酵母が使われます。ただ、やはり酵母も生き物ですので、毒性のある遺伝子は導入しにくく、また、リピート配列は抜けることがあります。そのため、酵母がゲノムの人工合成の標準になるかといえば、そうでもないと考えています。
PCR法には増幅できるDNAの長さが限られる、特別な装置が必要といった課題があります。GC含量が高い、あるいは繰り返し配列が多い遺伝子の増幅は困難ですし、増幅エラーが起きやすい点もネックです。
一方で、RA法+RCR法はセルフリーで、手間がかからず、数時間で長鎖DNA合成まで至ります。GC量が多いもの、リピート配列があるもの、毒性があるものも増やせるし、細胞を使わないので、安心して使えます。最終的にはデザインされた目的を持った、いろいろな種類のゲノムを合成することが可能となります。ゲノム合成が手軽になる分、バイオセキュリティーやバイオセーフティーといった倫理面での議論を深めていくことが今後求められます。
自動化も想定されます。DNAとタンパク質を連結反応液に入れて温めて、そこから一部を採取して増幅反応液に入れ、再度温めるだけの操作です。なので、分注機と保温機のようなシンプルな構成で済みます。
シングルセルからとってきたゲノムをRCR法で増やせば、ロングリードシーケンサーの前処理としても使えるのではないでしょうか。
Q. RA法とRCR法を普及させるために、どのような展開をお考えでしょうか
A. RA法、RCR法ともキット化して年内をめどに販売する予定です
RA法とRCR法のそれぞれの系について、反応液や25種類のタンパク質をセットにしてキット化し、そこにDNAを入れて温めればよいようにしました。このキット化はもともと研究室に入ったばかりの学生でも再現性よく実験を進めてもらうための工夫です。分注保存してありますので、それを取り出してDNAを入れて温めるという簡単な操作です。おかげで学生たちはたくさん実験を進めてくれます。
内閣府ImPACTのプロジェクトでこのキットを希望者に無償配布していたのですが、プロジェクトが終了したので、配布はいったん停止しています。今、会社を立ち上げていて、今年中には販売できるようにする予定です。社会実装のための第1歩です。
Q. RA法+RCR法で、産業ではどのような発展があるでしょうか
A. 有用な物質を産生する人工的な微生物の構築、遺伝子治療用のDNA製造などができそうです
デザインした長鎖DNAを手軽に構築できるようになるので、天然微生物にはないような有用な機能を持った人工微生物の構築が期待できます。いわゆる合成生物学産業です。
ほかにも、例えば医療分野では、遺伝子治療の分野で役立てるかもしれません。遺伝子治療用の製剤としてはウィルスやプラスミドDNAなどが使われていますが、これらの製造には大腸菌を使ったクローニングが必要とされます。ただし、大腸菌を使うとなると、時間と手間がかかるだけでなく、大腸菌由来の毒性成分の懸念も残ります。それと比べると、RCR法でセルフリー製造するDNAは医療用途に向いているといえるでしょう。
大腸菌を使ったクローニングは、未だ研究室で一般的な手法です。将来的には、RA-RCR法のキットを使う無細胞クローニングが大腸菌に取って代わる可能性もあると思っています。形質転換や、菌培養、プラスミド抽出といった古典的な操作から、解放されたいと思っている研究者は多いのではないでしょうか。
変わったところだと、コンピューターのデジタル情報をDNAのAGCTコードに直して、高密度に蓄積するというDNAストレージの分野が注目を浴びつつあります。この分野でも、長いDNA鎖を合成したり増幅したりする簡単な方法として利用できそうです。
DNAシーケンスの分野では、NGSという革新的技術によって、学術だけでなく産業面の発展も導かれました。一方、DNA合成の技術は徐々にコストダウンしているものの、まだNGSのような革新は起こっておらず、これから起こる可能性は十分あると予想しています。ここ数年の精力的なNGS解析により、あらゆる生命のゲノムの情報が蓄積し、その情報に基づく仮説が産生されてきていますが、そのような仮説を実験的に検証する有効なすべを我々はまだ持ちません。DNA合成の技術革新が起これば、コンピューター上でデザインされたゲノム丸ごとの人工合成が一般的なものとなり、その情報のアウトプットとしての生命機能を検証するアプローチが普及するでしょう。既存の生命科学の研究アプローチ自体が大きく変わる可能性があります。
Q. 今後はどのような研究を進めていく予定でしょうか
A. 複製系に転写や翻訳系を融合し、試験管内に「進化」を作りだしたいと考えています
複製系で増幅するDNAは、そもそも情報分子です。そして、その情報は転写翻訳系によって引き出すことができます。例えば、複製系を構成するタンパク質を全て遺伝子の情報に還元してDNAに載せてしまう。そして、転写翻訳反応を掛け合わせるとどうなるか。系の中に、自発的に複製系が発現し、それによって自身の情報をコードするDNAの複製が導かれます。このような自己複製系を構築しようとしています。
また、この自己複製系に進化能を持たせることにも挑戦しています。というのもDNA複製エラーを人為的に誘発することで、自己複製の過程において、遺伝情報の異なる多様性なDNA分子種が生み出されます。その種のうち、自己複製に優れたものが、淘汰され、優位に増えてくるようになるのではないでしょうか。ダーウィンの唱えた進化の仕組みの分子版です。ここまでを試験管内でできるようにしたいです。
Q. ベンチトップ型の次世代シーケンサーを導入予定と伺っています
A. 一回の反応で長鎖DNAの配列を確認できるように、iSeq 100 システムを導入する予定です
現在、RA-RCR法で構築したDNAの配列確認には、サンガー法を用いています。ただ、サンガー法では、1サンプルあたり500塩基対くらいしか読めません。研究の進展に伴って、構築するDNAサイズはどんどん大きくなり、1万〜100万塩基対のDNAを読む実験が多くなってきました。サンガー法でできなくはないですが、数十から数百本のプライマーを設計合成して、シーケンスサンプルを調整するのが手間です。一方で、NGSであれば、一回の反応で長鎖DNAの配列を確認することができます。これまでNGSは外注していましたが、時間がかかるのがネックです。高度なデータ量を要求しないので、手軽なスペックの機器ということで、近々イルミナ社の iSeq 100 システムを導入予定です。研究室が狭いので、iSeq 100システムのコンパクトさも、我々にとってメリットですね。
関連論文
1. Exponential propagation of large circular DNA by reconstitution of a chromosome-replication cycle
Masayuki Su’etsugu, Hiraku Takada, Tsutomu Katayama, Hiroko Tsujimoto
Nucleic Acids Research
参考
JST戦略的創造研究推進事業CREST
・[ゲノム合成]ゲノムスケールのDNA設計・合成による細胞制御技術の創出
・人工ゲノムのセルフリーOn chip合成とその起動
革新的研究開発推進プログラム(ImPACT)野地プログラム
・「豊かで安全な社会と新しいバイオものづくりを実現する人工細胞リアクタ」