少量の水から魚類の分布や環境が読める環境DNAで生物システムを網羅的に解析
神戸大学大学院人間発達環境学研究科の源 利文先生は、世界でも早い時期に環境DNA分析を用いる研究に着手し、学会設立や水を使う環境DNA分析のマニュアル作成にも尽力されています。今後多方面での応用が期待される環境DNA分析の方法論やその意義について、源先生に伺います。
Q. まず「環境DNA」「環境DNA分析」とは何かを教えてください。
A.「環境中に放出された生物由来のDNA」を「特異的に、あるいは網羅的に分析する手法」です。
「環境DNA」の定義は、国際的にもまだきちんと整理されていません。微生物から大型生物まで、また生きている個体やその分泌物、死骸などすべてのDNAを指す場合もあれば、生体内のDNAは除き、生物の体外に出たDNAだけを指す場合など、まちまちです。私自身は「環境中に放出された生物由来のDNAの総称」と捉えています。
研究としては、大別して2つの方向性があります。1つは魚などの特定の種類が環境中にいるかどうか、またその数が多いか少ないかを調べる種特異的な環境DNA分析(環境DNAバーコーディング)、もう1つは、魚なら魚について、その種類を網羅的に調べる、いわゆる網羅的環境DNA分析(環境DNAメタバーコーディング)です。
種特異的な環境DNA分析は、主にリアルタイムPCRで行います。これには、対象種のDNAを特異的に検出するプライマーの設計やその検証が不可欠です。一方、網羅的な環境DNA分析は、魚類の場合ユニバーサルプライマーを用いた次世代シーケンシング(NGS)によって、分析することができます。ただ、種特異的解析に比べると解析あたりのコストが大きいので、サンプルが十分数溜まってから実施するなどの工夫は必要ですね。
テーマによっては、種特異分析と網羅的分析の両方を実施します。
例えば、私たちが研究しているタイ肝吸虫は、まず貝に寄生し、その貝を食べた主にコイ科の魚、そしてその魚を食べたヒトに寄生します。感染ルートを追うには、寄生虫、貝、魚の3つの情報が必要です。寄生虫と貝は種特異分析、魚はいろんな種類の魚に寄生するため、網羅的分析を行い、それらの情報を重ね合わせることで、ヒトへの感染リスクのある場所を特定することを考えています。
東北4県の10か所ほどにしかいないとされる絶滅危惧種のゼニタナゴの調査では、本来の生息地であった秋田県の雄物川の99か所の水を採取し、NGSによる網羅的分析とPCRによる種特異的分析を両方行ったところ、種特異的分析のみで検出され、実際、陽性反応が出た地点にゼニタナゴがいたという経験があります。このように、希少な生物を対象にするならば種特異的分析を基本とし、他の生物とどういう関係にあるかを知りたい場合は網羅的解析で、といった使い分けが有効だと思っています。
Q. 環境DNA学会で「環境DNA調査・実験マニュアル」を作成された経緯を教えてください。
A. 将来的な知見の蓄積や応用を見据え、学会を設立し、マニュアルを作成しました。
5~6年前から、環境DNA分析はさまざまな研究分野に使えるだろうと多くの研究者が気づき始めていました。ただ、もしみんながバラバラに研究すると手法もバラバラになって、知見の蓄積や応用が難しくなるのではないかという懸念がありました。少なくとも海外ではすでにそういう事態になっていましたから。当時、日本では私たちの研究室が当初開発した手法やそこから派生した手法を使っている研究者が多く、今なら統一が可能だとして、環境DNA学会の創設とマニュアル作りが始まりました。学会の初代そして現会長の近藤倫生先生(東北大学 大学院 生命科学研究科)が旗振り役をしてくださったのは大きかったですね。主に学会の環境DNA技術標準化委員会のメンバーが分担してマニュアルを執筆し、初版は2019年4月に公開しました。
魚類の網羅的環境DNA解析の手法は、国際的にさまざまなチームが作っていたのですが、千葉県立中央博物館の宮正樹先生が作成され、私たちも開発に携わった、MiFish(マイフィッシュ)プライマーを使う方法が標準的な手法になりつつあります。MiFishプライマーを使う手法とヨーロッパのチームや米国などの他国の作成したプライマー間の比較がいくつかの論文で行われていて、いずれもMiFishプライマーを用いる検出法が一番よい、あるいは他と同等以上であるという結論になっています。「環境DNA調査・実験マニュアル」でもMiFishプライマーを使う方法を掲載しています。
2020年4月には改訂版を公開しました(https://ednasociety.org/wp/wp-content/uploads/2020/09/eDNA_manual_ver2_2_1.pdf)。改訂に向け、2020年初頭にマニュアルに関する意見を募集したところ、多数の要望がありました。私たちがそれほど重要ではないと考えていた部分、自由度を残しておく方がよいと判断した部分、まだ固まっていない部分も詳細を書いてほしいという要望が多数よせられました。改訂版ではすべてを反映することはできませんでしたが、採用できなかった意見は次回以降に入れていきたいと思っています。マイナーなアップデートはできれば毎年、メジャーなアップデートは2~3年に一度を目指しています。マニュアルに関係する動画も学会から公開の予定です。今後、研究者や研究対象が増えてくると、改訂などの作業はもっと大変になりそうですが、この手法の普及のために必要なことだと思います。
Q. 「環境DNA調査・実験マニュアル」で最もポイントとなるのはどの部分でしょうか。
A. ラボ内でのコンタミネーションを防ぐ方法を丁寧に説明しています。
「コンタミネーションの防止」ですね。サンプル中に数コピーしかないようなごく希薄なDNAを拾おうとするには、コンタミネーションが最大の敵です。マニュアルができる以前は「論文に書いてある通りに分析したのに、うまくいかない」という声が届いていました。論文にはノウハウの細かい記載がないため、直接問い合わせがあった方にはお伝えしてきましたが、通常の方法では、特にラボ内でPCR産物がサンプルに混入する、いわゆるキャリーオーバーコンタミネーションが起こりやすいことがわかってきたので、マニュアルではそのあたりも説明しています。ただ、当研究室でもやはりコンタミネーションをゼロにするのは難しく、環境の整備やルールづくりの他、データ処理の工夫など、様々な面からの対策が必要ですね。
Q. NGSによって出てきたデータの解析で難しいのはどんなところでしょうか。
A. DNA配列だけでは近縁種との区別がつかないケースがあることです。
NGS分析で出てきた配列から種が決まらないケースがあります。例えば、MiFishで増幅される遺伝子領域におけるクロダイの配列は南方に棲むミナミクロダイと同じです。私たちが舞鶴湾で調べた配列はクロダイだと考えられますが、厳密にはミナミクロダイとの区別はできません。「ミナミクロダイは舞鶴湾に居ないはず」という既存の知見を加えて、クロダイだと判定していますが、途中にこのような推察のステップが入ってしまうのです。もっと確実に判定するためには、MiFishとは異なるマーカー領域も見るべきですが、そうすると努力量が増え、環境DNA分析の良さが失われる恐れもあります。
また、遺伝子データベースに登録された種名が間違っているケースが意外に多いのも困るところです。私たちが利用している国際的な遺伝子データベースは、世界中のあらゆる研究者が自分で登録します。各国の担当機関が簡単な審査をしますが、内容には立ち入らないため、間違った情報を登録してもチェックできません。登録者に修正を求めるのも簡単ではなく、ほぼお手上げといった状態です。そのため、現在、環境省がデータを精査して安全なデータベースを作ろうとしています。こうした作業はいずれ学会が引き継がなければならないのではないかと思います。
なお、日本の淡水魚に関しては、9割以上がデータベースに配列が載っているので、ごくマイナーなもの以外は淡水魚に関してはほぼ特定できます。日本沿岸の海水魚は3~4割といったところでしょうか。そのためにデータベースや特異的に検出するプライマーの設計が重要になるということですね。
Q. 環境DNA分析や先生ご自身の研究はどのように始まったのでしょうか
A. 2000年代にウイルスの環境DNA分析から始まりました。
環境DNA分析は、まず微生物の分析が1990年代から2000年代初頭に盛んになりました。
目に見える大きさの生物(マクロ生物)の最初の研究例は、2008年、フランスのチームが行った、池の水の分析によるウシガエルの分布の報告です。その後、米国・五大湖のコイの外来種の調査など、世界中の何か所かで同時多発的に種特異的な環境DNAの研究が進みました。
私自身の行ったウイルスの環境DNA分析は、2007年ごろにスタートしました。総合地球環境学研究所(地球研)でコイに感染するコイヘルペスウイルスとコイ、人間の生活との関連を調べるうち、琵琶湖でのコイヘルペスウイルスの季節変動や地域差をつかむことができました。そして、宿主であるコイの量や感染の状態を知るには1匹のコイが出すウイルス量がわかれば概算できると思いつき、予備的な検討を行いました。すると、水中のDNA総量が非常に多く、コイ自身が糞や粘液などいろんな形でDNAを放出していることがわかりました。共同研究者である魚類生態学者の山中裕樹先生(龍谷大学 先端理工学部)とそんな話をしていたら、水から魚類の分布がわかるかもしれないというアイデアにいたり、以来、本格的に取り組みました。そして、琵琶湖の内湖のひとつである伊庭内湖(滋賀県東近江市)の水から、タナゴの仲間のカネヒラのDNAを検出したのが野生の魚の環境DNAを検出できた最初の成功例です。
2009年から今でいう環境DNAメタバーコーディングに取り組み始め、水槽に入れた5種の魚のDNAをランダムクローニングの後、サンガー法でシーケンスする形でコツコツと調べました。当時はNGS分析を行っていなかったので、この作業は大変でした。この研究の論文の電子出版が出たのが2011年で、これがマクロ生物の網羅的な環境DNA分析の最初の論文です(Surveillance of fish species composition using environmental DNA, Toshifumi Minamoto, Hiroki Yamanaka, et al., Limnology volume 13, pages193–197(2012)、https://link.springer.com/article/10.1007/s10201-011-0362-4)。1か月差でヨーロッパのチームがNGS分析を用いた論文を出してきたのには肝を冷やしましたね。
Q. 先生の最近のご研究についてご紹介ください。
A. 種特異的環境DNA分析と網羅的な環境DNA分析を組み合わせて研究しています。
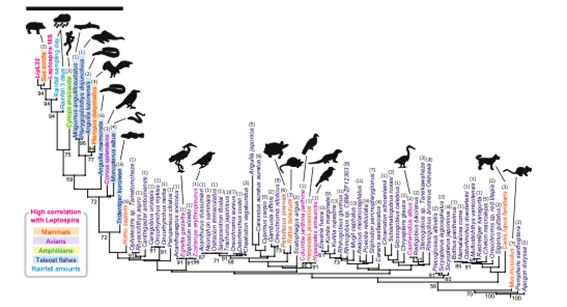
レプトスピラ症という人畜共通感染症について研究しました1。レプトスピラは野生動物や家畜、ペットなどの腎臓に保菌され、尿中に排菌されます。ヒトはこの尿と直接、あるいは尿が含まれた水や土壌などを介して感染します。軽い風邪のような軽症例から、黄疸や腎障害を伴う重症例までがみられます。国全体で見ると患者数は多くはないのですが、特に沖縄県では水辺のレジャーなどで感染する例があり、宿主がわかりませんでした。そこで、沖縄県内の2つの川で水を汲み、水中のレプトスピラの特異的分析と脊椎動物の網羅的分析を繰り返して、イノシシかブタのDNAがレプトスピラのDNAと共に出てくることを明らかにしました。恐らく野生のイノシシが保菌していると推測されます。感染症の病原体の特異的分析と自然宿主候補の網羅的分析を同時に実施すると、こういうことがわかります。
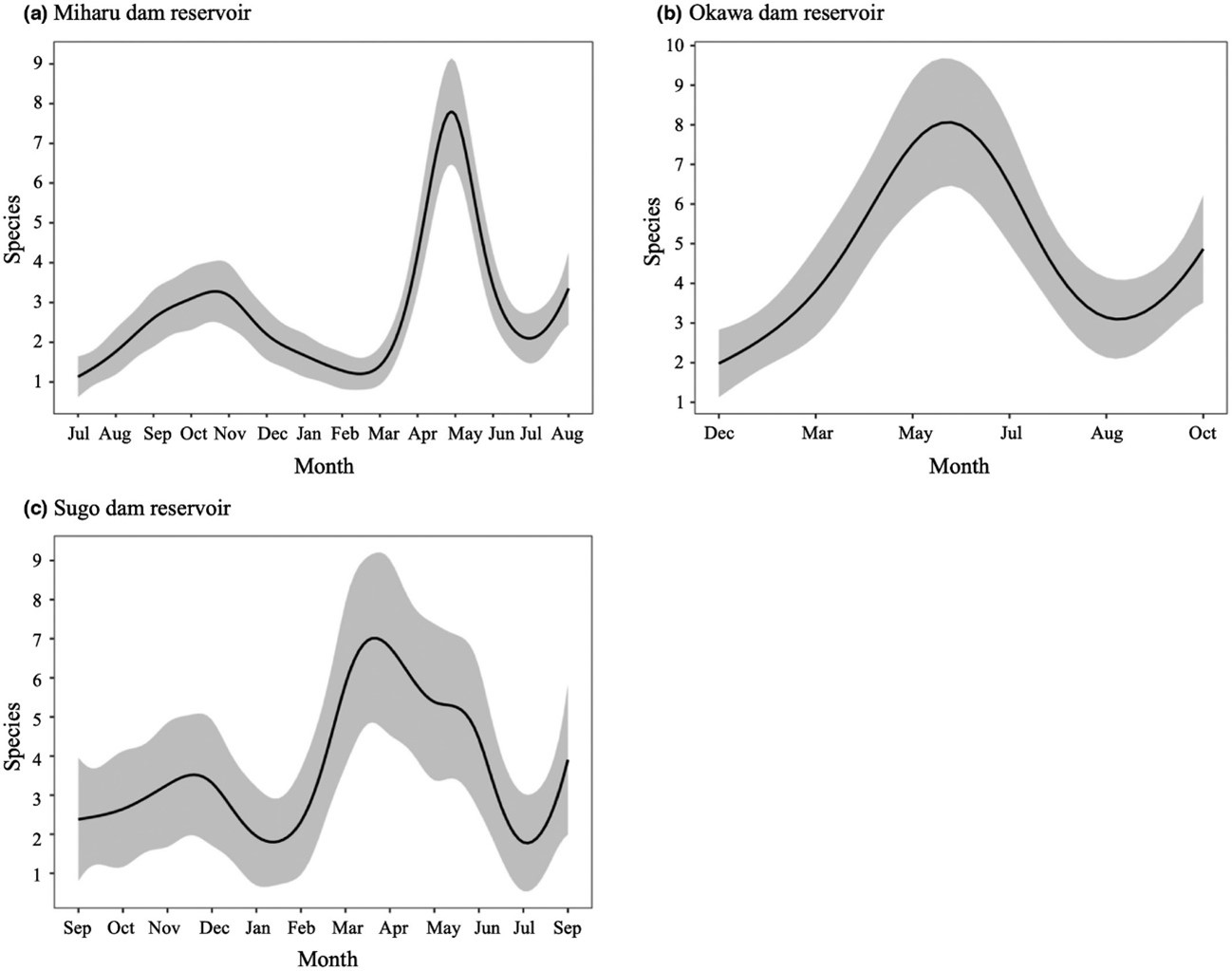
また、水中での特異的な環境DNAの検出にはいつの季節がよいかに関しては、論争があります。夏場は生物の活動が活発になってDNAがより放出されると思われる一方で、水温が高く、微生物活性が上がるので、放出されたDNAの分解も促進されます。逆に冬場はDNA放出量は少ないけれど、分解も遅いだろうと。ですから、どの季節が一番良いのかは調べてみないとわからないわけです。私たちは3つのダム湖で1年を通して水を採取し、春が最も多くの魚種が検出されることを明らかにしました2。これは、春に産卵する種が多く、産卵期は個体の動きが活発であること、精子や卵の放出があること、また仔稚魚は代謝が高いことなどが原因で、環境DNAが増えるようです。
Q. 今後はどのような研究を計画していらっしゃいますか。
A. 環境DNA分析をさらに発展させるような研究をしたいと考えています。
基礎研究としては、そこにいる生き物のストレスのような生理状態や繁殖行動をキャッチしてみたいと思います。DNAが難しければRNAを使えるかもしれません。
応用的な研究としては、魚類の分布の情報を水産業に利用する、あるいは寄生虫や病原体の分布の情報をヒトやその他の動物の健康に生かせないかと考えています。
過去復元の研究にも可能性を感じています。海洋のイワシやアジなどは堆積物のDNA量と水揚げ量を比較する研究が行われています。同じように湖底の堆積物をスライスして上から順番に網羅的な解析を加えれば、その場所の魚類相の変遷がわかるかもしれません。今のところ、少なくとも300年分くらいの変化であればつかめそうだということがわかってきました。水や温度、水質といった環境情報も組み込むことができれば、人間活動の影響も推定できるかもしれません。
環境DNA分析の面白いところは、サンプルさえ採ってしまえば、後からいろいろな目的でそのサンプルを再利用できることです。微生物からマクロ生物まで、環境DNA分析という技術で串刺しすることで、新たな世界が見えてきます。

岡山県でのフィールド研究の様子。「水を求めて、各地に行けるのがこの研究の楽しいところです」
参考文献
1. 1. Environmental DNA metabarcoding to detect pathogenic Leptospira and associated organisms in leptospirosis-endemic areas of Japan Y Sato, et al., Scientific Reports, (2019) 9:6575 https://doi.org/10.1038/s41598-019-42978-1 Hayami K, Sakata MK, Inagawa T, et al.
2. Effects of sampling seasons and locations on fish environmental DNA metabarcoding in dam reservoirs. Ecol Evol. 2020;10:5354–5367.
https://doi.org/10.1002/ece3.6279