がん研究における全エクソーム解析の意義およびワークフローの検証
はじめに
がん研究では、がん細胞のゲノム異常やがんゲノムの進化を網羅的に解析する手法として全エクソーム解析が広く利用されています。大阪大学大学院医学系研究科の谷内田真一教授、高田創先生、金井悠先生にご協力いただき、イルミナの新しい酵素による断片化法を用いたライブラリー調製キットIllumina® DNA Prep with Enrichment, (S) Tagmentation (旧名:Nextera Flex for Enrichment) とTwist Bioscience社のエクソームパネルによるホルマリン固定パラフィン包埋(FFPE)サンプルを対象とした全エクソーム解析を実施いただきました。更に全エクソーム解析の意義などについて谷内田先生からお話いただきました。
がん研究における全エクソーム解析の意義
谷内田先生が全エクソーム解析に携わったのは、2007年Johns Hopkins大学に留学した頃に遡ります。今では考えられない力業ですが、「サンガー法」で全エクソーム・シークエンスを行っていたことを身近で見ることができ、多くの発見がありました。先生のNatureで発表された論文もサンガー法での全エクソーム解析です。
その後は次世代シークエンサーの登場により、サンガー法はValidationのみに使うことになりましたが、今は次世代シークエンサーの技術的な信頼性も上がり、サンガー法でのValidationも行う必要性はほとんど無くなりました。
谷内田先生の研究テーマのひとつは難治がんや希少がんの網羅的ゲノム解析です。希少がんは症例数が少なく、新鮮凍結検体の入手が困難であるため、FFPEサンプルが解析対象となります。そのため全エクソーム解析が効果的な解析手法となるそうです。全エクソーム解析の利点として、先生より以下4つをあげていただきました。
- 解析パイプラインが安定している。全エクソーム解析の歴史は長く、効率良くがんにおける体細胞変異を探索できることから、網羅的がんゲノム解析に広く用いられている。
- セルフリーDNAにも応用可能である。血中腫瘍DNAは極微量のため、精度良く変異を検出するためにはディープシークエンスが必要となる。
- コストが安い。希少がんのFFPEサンプルには、腫瘍率の低いサンプルも多数存在し、深く読む必要がある。全ゲノムよりコストを抑えて、ディープシークエンスでの網羅的ゲノム解析ができる全エクソーム解析により、多くのデータを得ることができる。
- 探索研究目的にはパネルより向いている。希少がんや特殊な疾患の新規がん遺伝子やがん抑制遺伝子の探索が目的であるため、網羅的ゲノム解析ができる全ゲノムや全エクソーム解析が適している。希少がんの検体はFFPEサンプルが多いため、全エクソーム解析が適している。
また、全エクソーム解析で行いたい研究はまだまだ多く、例えばがんゲノムの進化を解明するために、がんにおける微小環境を対象とした全エクソーム解析を行いたいとのことでした。そのためには、各サンプルの正確な位置関係情報に紐づいた全エクソーム解析手法が重要になるので、技術機器の開発にも期待したいと語って頂きました。
全エクソーム解析のワークフロー検証
今回、イルミナのBead-linked transposome(BLT)テクノロジーを用いた酵素による断片化法と、大幅な改良を行った濃縮試薬を含むライブラリー調製キットIllumina DNA Prep with Enrichment, (S) TagmentationとTwist Bioscience社のエクソームパネルを使って、希少がんFFPEサンプルの全エクソーム解析を行いました。Illumina DNA Prep with Enrichment, (S) Tagmentation の大きな特徴の一つは、ワークフローの簡便さと操作時間の短さです。DNAの断片化に超音波破砕装置など特別な装置を必要とせず、合計のハンズオン時間は約2時間になります。ワークフロー全体にかかる時間も6時間半と短縮されます。 その操作の簡便さについて、高田先生と金井先生から以下のコメントを頂きました。
高田先生(写真左):「以前使用したTruSeq Exome Kitより操作時間がだいぶ短くなったので、検体数が多いときなど、半分の時間で処理できるので、とても便利と感じました。また必要なデータもとれているため、いいキットだと思います。」
金井先生(写真右):「今回初めてのシーケンスライブラリー調整だったのですが、プロトコルが簡便で効率よく作業をすすめることができました。得られたライブラリーの品質も安定しており、どなたにも使いやすい製品だと思います。」

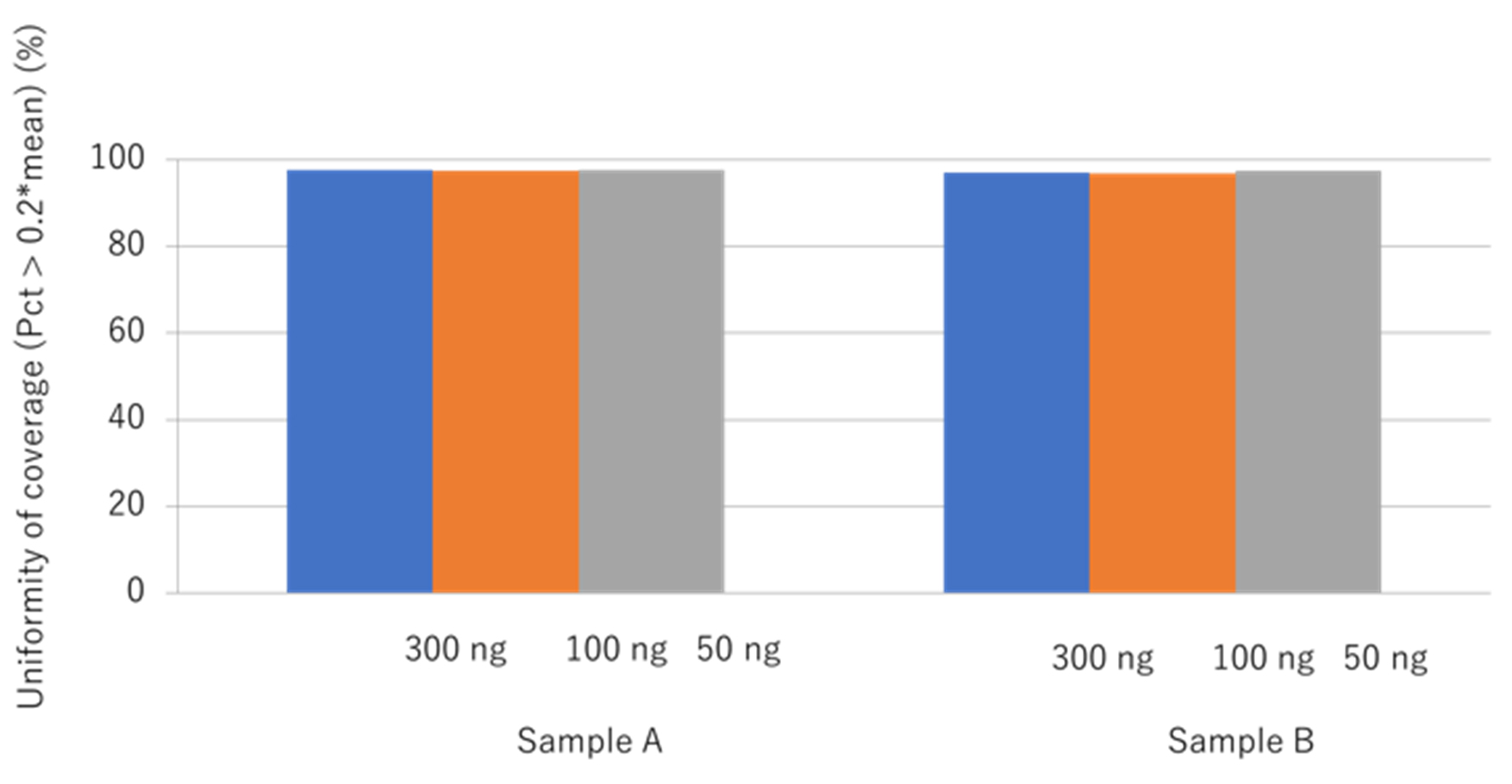
Illumina DNA Prep with Enrichment, (S) Tagmentationキットは、FFPEサンプルからのDNAインプット量は50ng-1000ngと幅広く対応しています。DNAインプット量のカバレージ均一性への影響を検証するために、今回2つのFFPEサンプルのインプット量を50ng、100ng、300ngに分けて行った実験結果を図1に示しました。どちらのサンプルでもDNAインプット量の多少に関わらず高い均一性が得られました(図1参照)。そのため本キットを使用すれば、FFPEサンプルから抽出された品質の低いDNAであっても、効果的なエクソーム解析が可能であることが示されました。
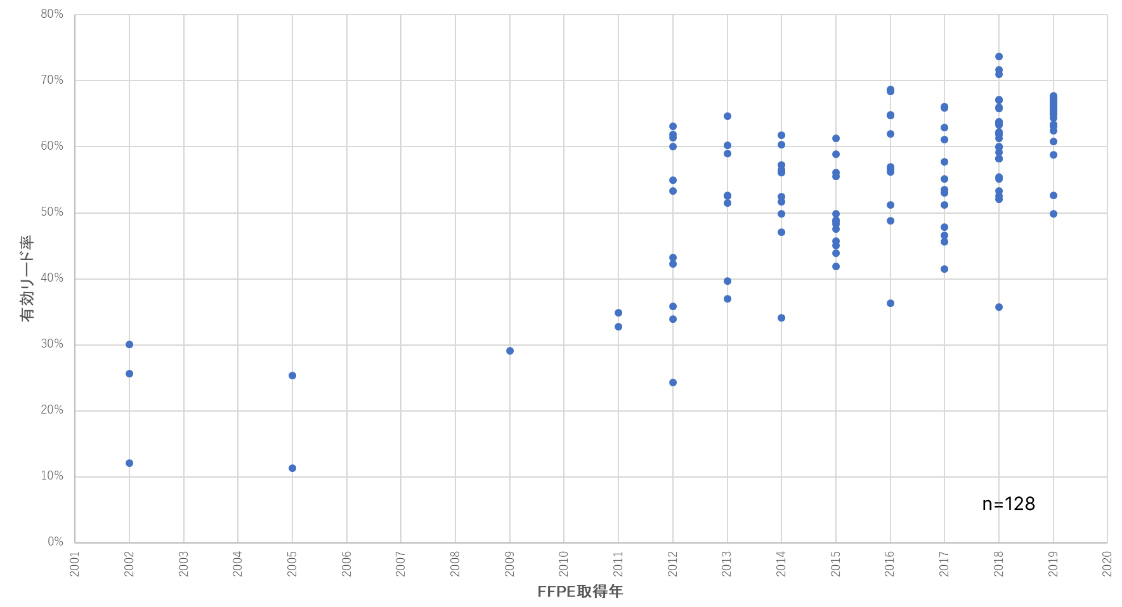
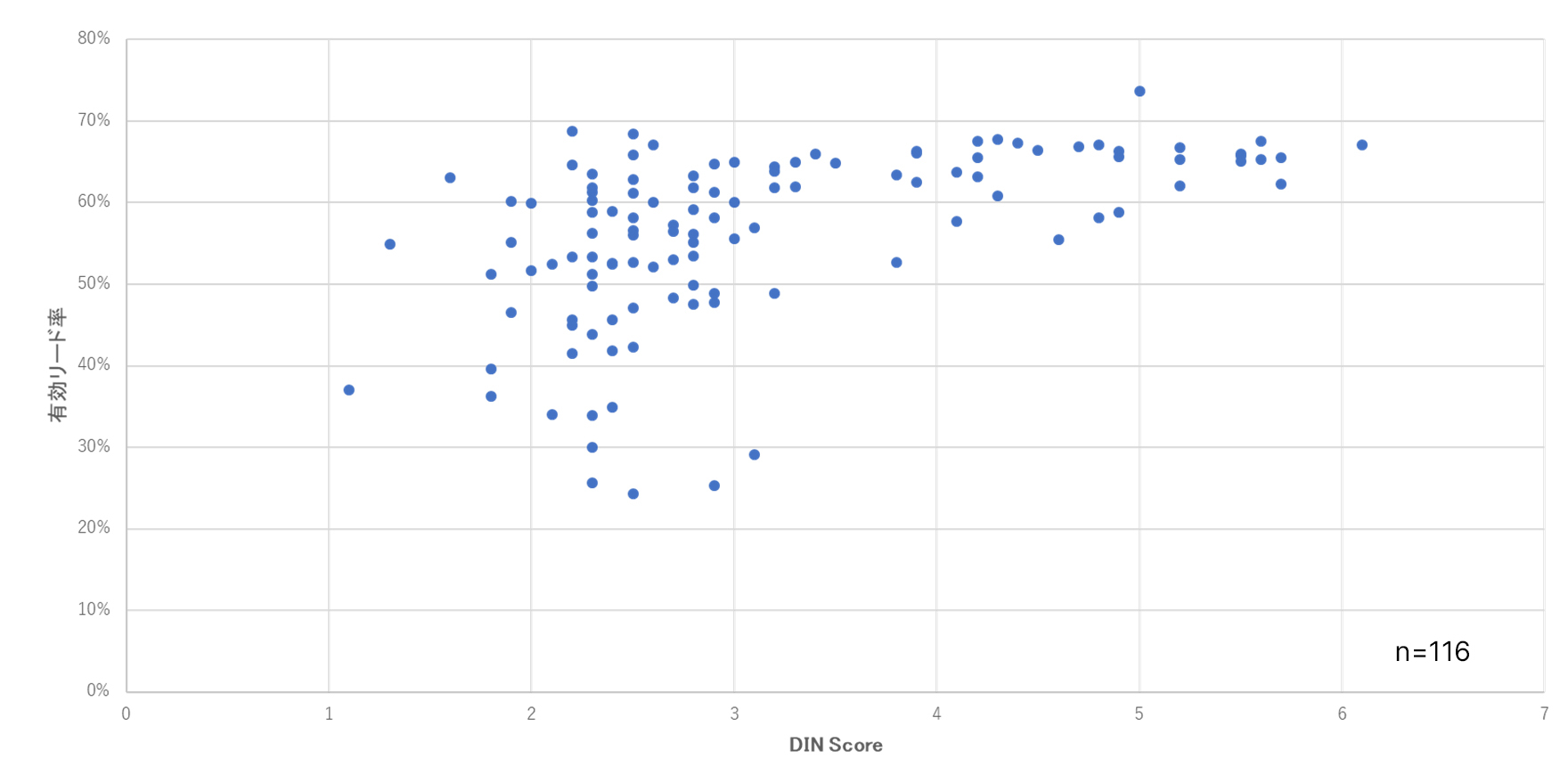
また、今回の評価に用いたFFPEサンプルは、2012年取得など比較的古いものもあったため、得られるDNAの品質低下、それに伴うシーケンス結果への影響が懸念されました。そこでFFPE取得年と有効リード率*1の関連性、サンプルのDIN値*2と有効リード率の関連性を検証しました(図2、3参照)。一般的に古いFFPEサンプルは有効リード率が低くなりますが、2012年のサンプルであっても著しい有効リード率の低下は観察されず、比較的安定した結果が得られました(図2)。 一方で、DIN値と有効リード率のプロットにおいてはDIN値3以上であれば安定した有効リード率が得られており、良いシーケンス結果を得るための指標の一つとしてDIN値の有効性が示されました。
*1 有効リード率:全リードからduplicate readなどを除去した後、最終的に解析に利用できるリードの割合
*2 DIN (DNA Integrity Number) は、アジレント社のTapeStation システムで得られたプロファイルからゲノムDNAの分解度を客観的に評価する指標です。ゲノムDNAの分解度を1~10のDIN値で表示します (1 = 完全に分解された状態、 10 = ほとんど分解していない状態)。
FFPEサンプルを用いた全エクソームのディープシーケンスは、がんゲノムの研究を進める上で今後も必要性の高いアプリケーションであるとイルミナは考えております。谷内田真一教授、高田創先生、金井悠先生にいただいたご意見、ご提供いただきましたデータは、これから実験を行う研究者の皆様の参考になると考えております。貴重なデータをご提供いただき大変ありがとうございました。