Abstract
マッピングとバリアントコーリングのためのヒトリファレンスゲノムの選択は、精度に直接影響を与えます。この選択は、GRCh37/hg19またはGRCh38/hg38かという単純な決定にとどまりません。各リファレンスには複数のバージョンが存在し、代替コンティグやデコイコンティグを含むものもあれば、含まれていないものもあります。また、特定のバージョンには、他のバージョンではカバーされていないゲノムの一部が含まれていることもあります。最終的な選択は、バリアントをコールする能力に影響を与える可能性があります。これは、CBS、CRYAA、KCNE1遺伝子に関する最新情報で説明できます。GRCh37/hg19ではこれらの遺伝子が1コピーのみ存在しますが、GRCh38/hg38では誤った重複が含まれているので、GRCh38/hg38においてのみマッピングの曖昧さが生じることが確認されました。2021年、Genome in a Bottle(GIAB)は、重複領域のマスキングを使用して、関連する遺伝子セットの偽重複を効果的に除去することで、バリアントコールの精度を向上させることを提案しました[1]。
2020年、DRAGENチームは、精度を向上させるための別の補完的なアプローチを採用しました。DRAGENは、ゲノムの「マッピング困難領域」におけるイルミナリードのマッピング精度を向上させるために、グラフベースのゲノムを開発しました。この改善はバリアントコーリングレベルで確認され、PrecisionFDA Truth Challenge v2の「マッピング困難領域」および「全ベンチマーク領域」で最高の精度を示しました[2]。グラフゲノムには、GRCh38/hg38リファレンスに追加され、フェージング済みの集団ハプロタイプに由来する数十万の短い代替コンティグが含まれています。これにより、GRCh38/hg38ヒトリファレンスの新たなバージョンが事実上作成されることになります。
リファレンスを強化することで、下流のアノテーションに影響を与えずにバリアントコールをさらに改善することには多くの利点があります。特に、医療的に重要な遺伝子を含む領域のリファレンスを改善する際に重要となります。しかし、新たに導入されるリファレンスのバリエーションごとに、ユーザーの混乱を招く可能性があることも認識しています。なぜなら、「GRCh38/hg38」という用語だけでは、解析に使用されるリファレンスを完全に表現できなくなっているためです。本稿では、GRCh38/hg38リファレンスのさまざまな種類を明確にし、DRAGENで使用する現在の推奨リファレンスについて説明し、さらに計画されている改善点について議論します。
推奨GRCh38/hg38リファレンスバージョン
現在、DRAGEN 3.9で使用することを推奨しているGRCh38/hg38のハイレベルな2つのバージョンは、hg38-alt-masked(非グラフ)およびhg38-alt-masked-graphです。どちらもダウンロード可能です[3]。これらのリファレンスはいずれも、DRAGEN v3.9で導入されたalt-masked機能を含んでいます。これについては、以下に詳しく説明します。この新しいalt-masked機能は、これまで推奨されていたリフトオーバーを基にしたALT認識リファレンス(hg38-alt-aware(非グラフ)およびhg38-alt-aware-graph)と比較して、わずかに精度が向上しています(表1参照)。

表1:DRAGENで使用するための推奨GRCh38/hg38リファレンスバージョン(グラフなしとグラフラン用)。これらすべてのリファレンスのベースラインアセンブルは[4]からダウンロードされたことに注意してください。
hg38-alt-maskedリファレンスをダウンロードできます[3]。ハッシュテーブルは、次のコマンドを使用して構築することもできます。
dragen --ht-reference hg38.fa --ht-alt-aware-validate=true --ht-num-threads=40 --build-hash-table=true --ht-build-rna-hashtable=true --enable-cnv=true --ht-mask-bed /opt/edico/fasta_mask/hg38_alt_mask.bed --output-directory hg38_alt_masked
alt-maskedハッシュテーブルの作成はDRAGEN 3.9以降でサポートされていますが、3.9 alt-maskedハッシュテーブルは旧バージョンのDRAGENでも使用できます。
hg38-alt-masked-graphゲノムハッシュテーブルをダウンロードできます[3]。hg38-alt-masked-graphハッシュテーブルは、DRAGENの3.9以前のバージョンと互換性があります。DRAGENは、ユーザーが独自のカスタムグラフゲノムを構築することに対応していません。これは、新しいハプロタイプがゲノムのその他の領域と競合する場合、集団のハプロタイプを変更すると精度の低下を引き起こす可能性があるためです。グラフゲノムの構築には細心の注意が必要です。そのため、現在、DRAGENでは完全に自動化されたアプローチをサポートしていません。
以前に構築されたBAMと新しいバリアントコーリングランの互換性に関する注意事項:マッピング/アライメント段階とバリアントコーリング段階で同じリファレンスを使用することが重要です。したがって、BAMが“*alt-aware”でマッピングされている場合、バリアントコールもalt-awareを使用する必要があり、同様にalt-maskedを使用する場合も一貫して適用する必要があります。ユーザーがalt-maskedで結果を更新したい場合、alt-maskedでバリアントコールを行う前に、alt-maskedでBAMを再マッピングすることをお勧めします。
GRCh38/hg38のすべてのバージョンのベースラインとしての共通リファレンスアセンブル
DRAGENでの使用が推奨されるGRCh38/hg38リファレンスの全バージョン(表1参照)では、[4]からダウンロードした共通のFASTAアセンブルをベースラインとして使用しています。次に、FASTAアセンブリが一連の集団SNPおよび/またはフェージングされたバリアントの一連の集団由来ハプロタイプによって拡張されているかどうかに応じてバージョンが区別され、線形リファレンスに対する代替パスが提供されます。これにより、グラフベースのバージョンが生成されます。詳細については、[2]を参照してください。さらに、ベースラインアセンブルに含まれるネイティブのGRCh38/hg38 ALTコンティグは、DRAGENマッパーの“alt awareness”機能(alt-awareバージョン)または新しいalt-masked機能(alt-maskedバージョン)のいずれかで処理できます。
コマンドFASTAアセンブリには、以下の表2に説明されている3366種類のコンティグが含まれます。

表2:[4]からダウンロードした一般的なFASTAアセンブリのコンポーネント
ネイティブGRCh38/hg38 ALTコンティグの取り扱い:ALT-awareとALT-maskingの比較
バージョン3.9のリリース以前、DRAGENは‘ALT-aware'アラインメントのリフトオーバープロセスを採用し、一部のhg19およびhg38 FASTAファイルに含まれる代替(ALT)コンティグを活用していました。この方法では、一次染色体コンティグに対するALTのアライメントを含むリフトオーバーSAMファイルを使用します。これらのアライメントはGenome Reference Consortiumによって作成されました。ALTコンティグ領域に最も適しており、有効なリフトオーバーのあるリードは、対応する染色体の位置までリフトされます。有効なリフトオーバーのないALT領域に最も適したリードは、ALTコンティグにマッピングされたままになります。
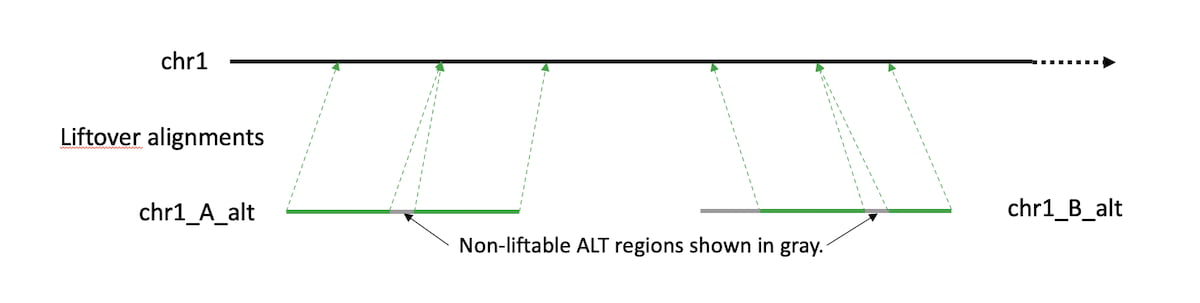
図1:代替領域とメインリファレンス間の正しいリフトオーバーの例。
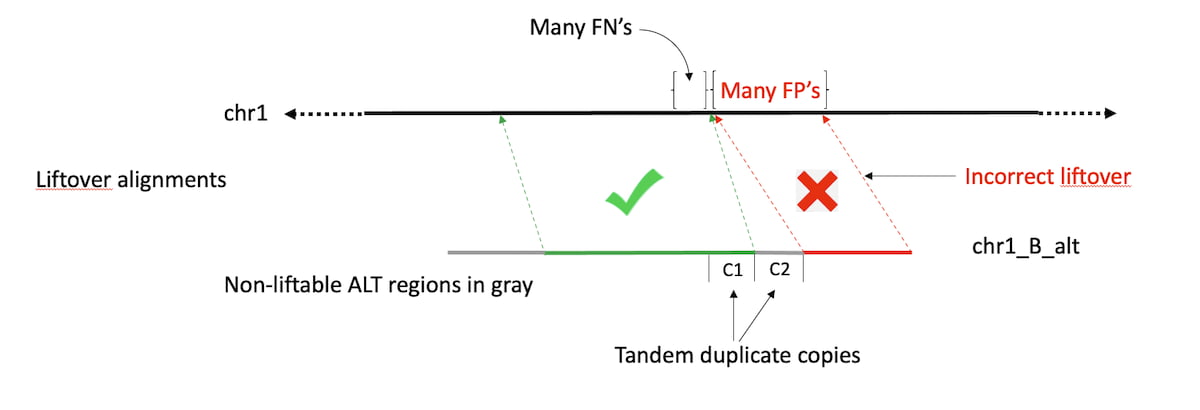
図2:不適切なリフトオーバーアライメントは、余分なFPを生成する可能性があります。
DRAGENには、ネイティブリファレンスのALTコンティグを処理するための新しいアプローチが導入されました。このアプローチでは、ALTコンティグの戦略的位置がマスキングされ、精度が向上します。一次アセンブルと類似したセグメントはマスキングされるため、競合してアライメントを奪ったり、MAPQ(マッピングクオリティ)を低下させたりすることはありません。非常に異なるセグメントはマスクされずに残り、基本的にデコイシーケンスとして機能します。複数の境界領域のマスキング状態は、マッピング精度およびバリアントコールへの実証的な影響に基づいて割り当てられます。結果セクションの図3に示すように、DRAGENのALTマスクリファレンスは、リフトオーバーベースのALT認識法と比較して、バリアントコールの精度を向上させます。塩基マスキングアプローチには、マイナスの結果をもたらすことなく、ALTコンティグを使用できる利点があります。また、定義、維持、改善も容易です。今後もマスクの改良は続きますが、すでにリフトオーバーベースの性能を上回っています。
Broad(https://github.com/Illumina/DRAGMAP)とのコラボレーションによりリリースされたDRAGENマッパーのオープンソース版であるDRAGMAPは、リフトオーバーベースのALT認識をサポートしていません[5]。代わりに、DRAGMAPは、DRAGENの最新バージョンで使用されているのと同じ推奨alt-maskedアプローチを使用します。これは、従来のリフトオーバーアプローチと比較してマスキングアプローチの精度が向上しているため、DRAGMAPではリフトオーバーをサポートする必要がないことを意味します。
グラフベースのリファレンス
前述のように、DRAGENは、ゲノムのマッピング困難領域におけるイルミナのリードのマッピング精度を向上させるグラフベースのリファレンスもサポートしています。グラフ機能は、GRCh38/hg38のネイティブなALTコンティグを使用せず、慎重に選ばれた集団ハプロタイプセグメントを利用します。このため、相同領域の識別が可能になり、集団において既知の代替パスが線形リファレンスに提供されます。
領域(MHCなど)が高度に多型的である場合、サンプルリードがリファレンスゲノムと大きく異なるため、マッパーは適切な一致を見つけたり認識したりすることができず、マッピングが困難になる可能性があります。しかし、より一般的には、サンプルのリードがある領域に適切に一致するものの、その他の領域にもほぼ同程度または完全に一致してしまう場合に、マッピングが困難になる可能性があります。これは、リファレンスゲノムの複数の場所に類似のコピーが存在する場合(セグメントの重複)、または一般的な非常に反復性の高いシーケンスの場合に起こります。
多くの場合、このようなマッピングの難しさは、単一のリファレンスゲノムだけでなく、集団内の既知のバリエーションパターンを利用することで克服できます。ショートリード(またはリードペア)が、AとBの2つの領域に同等レベルで一致しているが、それぞれの一致が不完全であり、各領域においてリファレンスゲノムと2つのヌクレオチドの違いがあると仮定します。リファレンスのみに基づく場合、マッパーはAまたはBをランダムに選択してその領域にアライメントし、MAPQは0となります。しかし、リードと領域Aとの2つの違いは集団で一般的に発生するのに対し、領域Bとの2つの違いは集団では観察されていないことが分かっていると仮定します。この知識をガイドとして使用して、リードを領域Aに合理的に高い信頼度でマッピングすることができます。
ゲノムの困難領域におけるマッピング精度を向上させる鍵となるのは、集団ハプロタイプセグメントの選択にあります。数と集団の多様性を増やすと、精度がさらに向上しますが、ハプロタイプが互いに競合しあいまいなリードマッピングにつながった場合、マイナスの影響が生じる可能性があります。マスクベースのhg38 ALT認識は、グラフリファレンスとの相性も良く、干渉を避けつつグラフパスのリフトオーバーがマッピングを適切に誘導できるようにします。
グラフリファレンスは現在、DRAGENのハードウェアアクセラレーションのバージョンのみでサポートされているため、DRAGMAPではサポートされません。
リファレンスの性能の測定
alt-maskedアプローチとリフトオーバーを基にしたALT認識の影響を比較した結果は、NIST v.4.2.1の真理値セット[6]を使用し、グラフリファレンスおよび非グラフリファレンスの両バージョンについて図3に示しています。alt-maskingアプローチにより、SNPおよびIndelの両方において、グラフリファレンスと非グラフリファレンスの両方でFP(偽陽性)およびFN(偽陰性)の数が減少します。
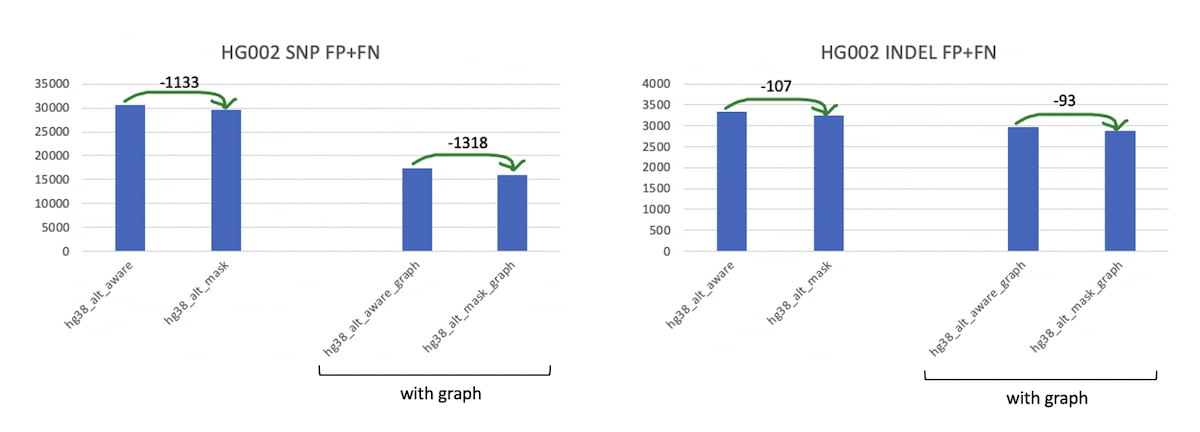
図3:NIST 4.2.1の真理値セットを使用した、alt-maskedとalt-awareの精度の比較
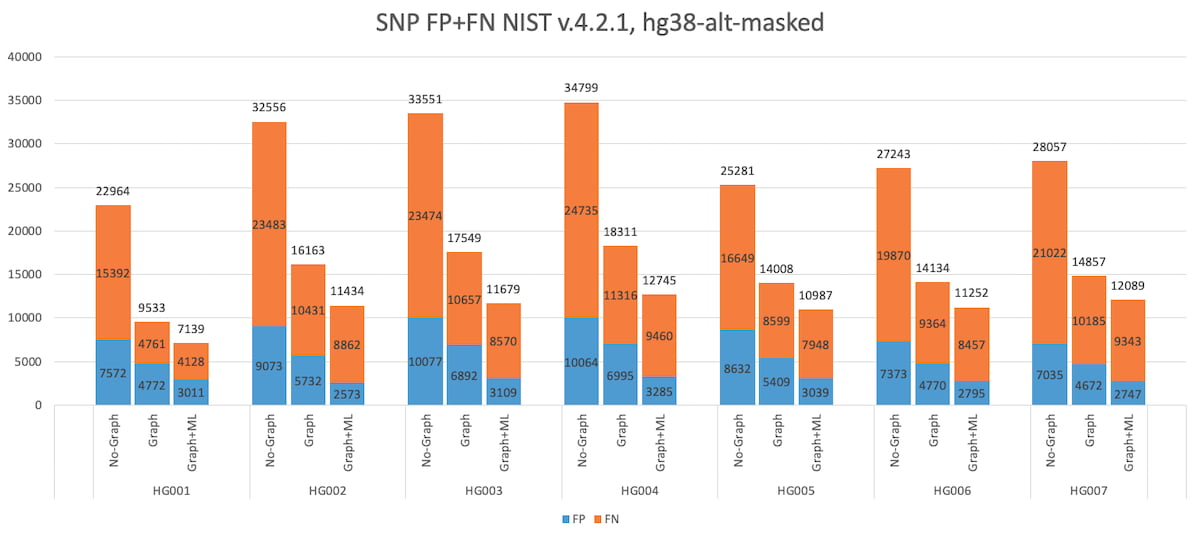
図4:拡張真理値セット(v4.2.1 VCFおよびBED)におけるHG001~HG007のSNP精度結果
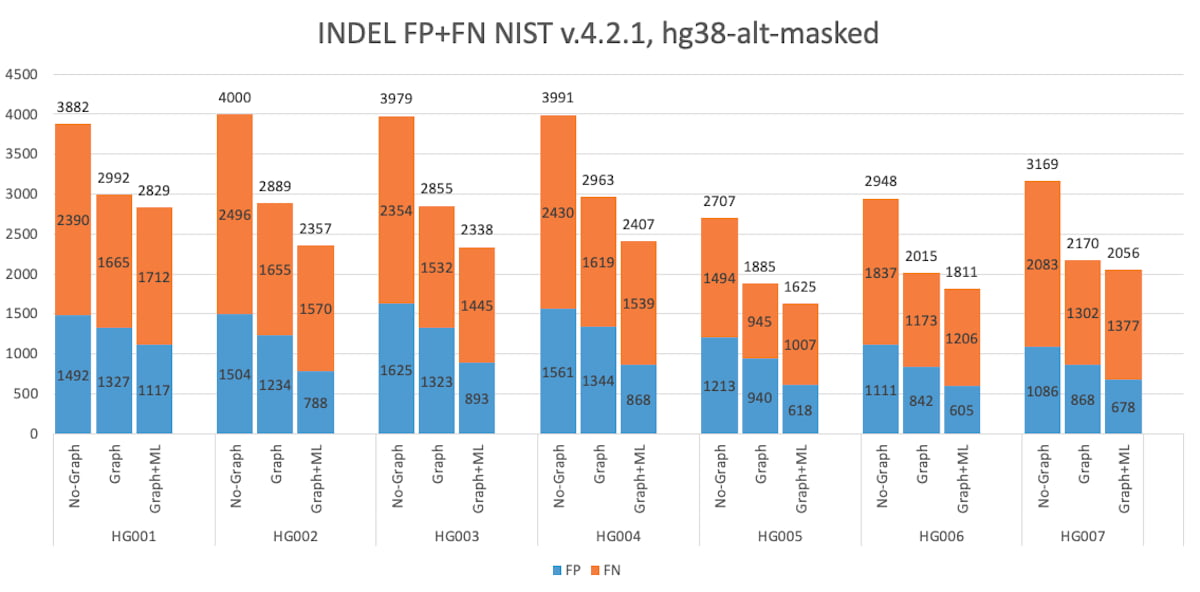
図5:拡張真理値セット(v4.2.1 VCFおよびBED)におけるHG001~HG007のIndel精度結果
グラフリファレンス(またはその他のリファレンス改善)の影響を測定する際には、最新かつ最も包括的な真理値セットを使用することが重要です。例えば、DRAGENグラフからの総エラーの50%減少は、拡張真理値セットv4.2で測定可能です。この性能の向上は、旧版のv3.3.2の真理値セットに対するベンチマークでは明らかとはなりません。これは、v3.3.2の真理値セットにはマッピング困難領域が含まれていないことと、v4.2の真理値セットがv3.3.2に存在するエラーを修正しているためです。実際、古い真理値セットv3.3.2を使用した場合、DRAGENグラフは従来のDRAGENと比較して、追加のSNPおよびIndel偽陽性(FP)を生じる傾向があります。しかし、追加されるFPコールの大部分は、v3.3.2の真理値VCFが不完全であることが原因です。v4.2の真理値VCFでは、同じFPバリアントが真陽性とマークされています。
GRCh38/hg38リファレンスのさらなる改善(3.9リリース以降のDRAGENで採用)
ゲノムマスキングとグラフの両方の潜在能力は、精度の点でまだ十分に引き出されていません。Genome in a Bottle(GIAB)コンソーシアム、Genome Reference Consortium(GRC)、およびTelomere-to-Telomere(T2T)コンソーシアムもGRCh38/hg38リファレンスゲノムの改善に貢献し[1]、1)一次アセンブルにおける誤った重複を除去するための塩基マスキング、2)新たなデコイコンティグの追加という2つのタイプの改善を行いました。現在、DRAGENチームはこれらの変更を評価し、最新のリファレンスバージョンに取り入れているところです。表3に、今後のDRAGENリリースでリファレンスアップデートをリリースする計画のロードマップを示します。新たにマスクされた塩基が最初に組み込まれ、アップデートされたリファレンスは*alt-masked-V2*という名称になります。新しいデコイはまだ完全には完成しておらず、今後のリリースに組み込まれる予定です。

表3:DRAGENで使用するための推奨GRCh38/hg38リファレンスバージョン(グラフなしとグラフ実行用)。これらすべてのリファレンスのベースラインアセンブリは[4]からダウンロードされたことに注意してください。
新たにマスクされた塩基の詳細:alt-masked-V2に向けて
最近、GIABコンソーシアムはGRCh38/hg38の偽重複をマスクする新しいリファレンスを発表しました[1]。GIABはGRCと協力し、GRCh38において座標を変更することなく、またバリアントコールに悪影響を与えることなくマスキング可能な領域のリストを作成しました。これらの領域は、誤って重複したシーケンスやコンタミネーションによるものでした。これらの重複領域はT2Tによって同定されました[7]。新たにマスクされた塩基には21番染色体の一部が含まれており、いくつかの主要な医療遺伝子CBS、CRYAA、KCNE1のマッピングが改善されました。
以下の図6は、[1]の補足資料から抜粋したものです。GRCh37には偽重複が含まれていないため、GRCh37リファレンスではテクノロジー間のバリアントコールがどのように一貫しているかを示しています。GRCh38には遺伝子の一部の偽重複が含まれているため、多くのリードが遺伝子の偽コピーであるKCNE1Bに誤ってマッピングされています。

図6:GRCh37およびGRCh38(新たにマスクされた塩基なし)によるさまざまなテクノロジーのKCNEカバレッジ[1]。
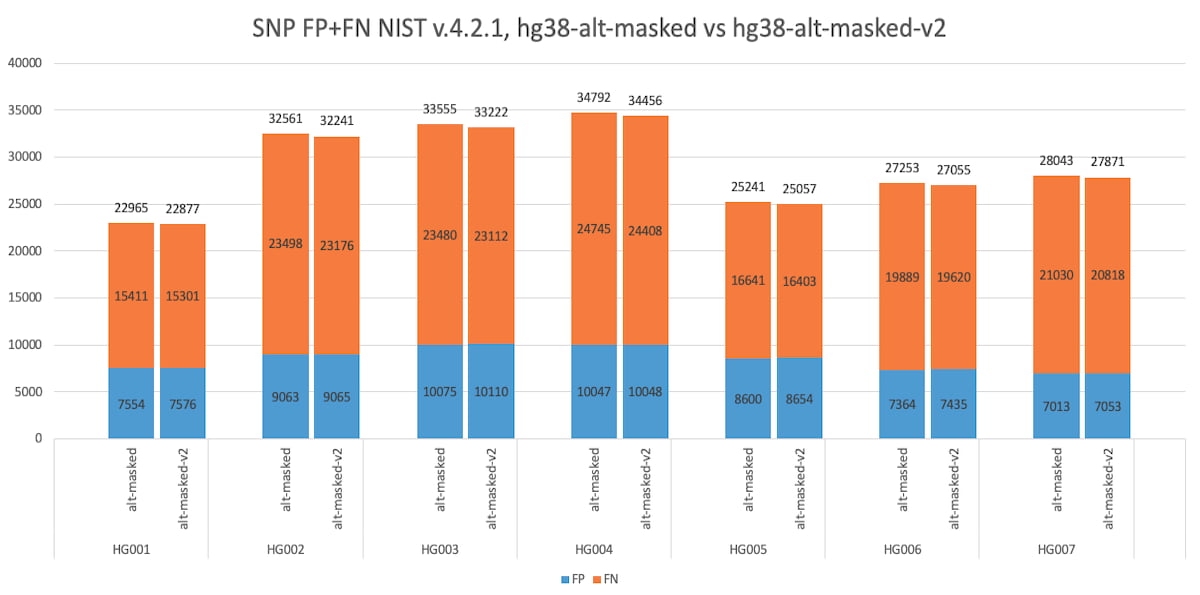
図7:V.4.2.1 NIST真理値セットを使用して全ゲノムで評価したGIABマスクリファレンスによる精度改善。SNPについて、hg38-alt-maskedとhg38-alt-masked-V2を比較
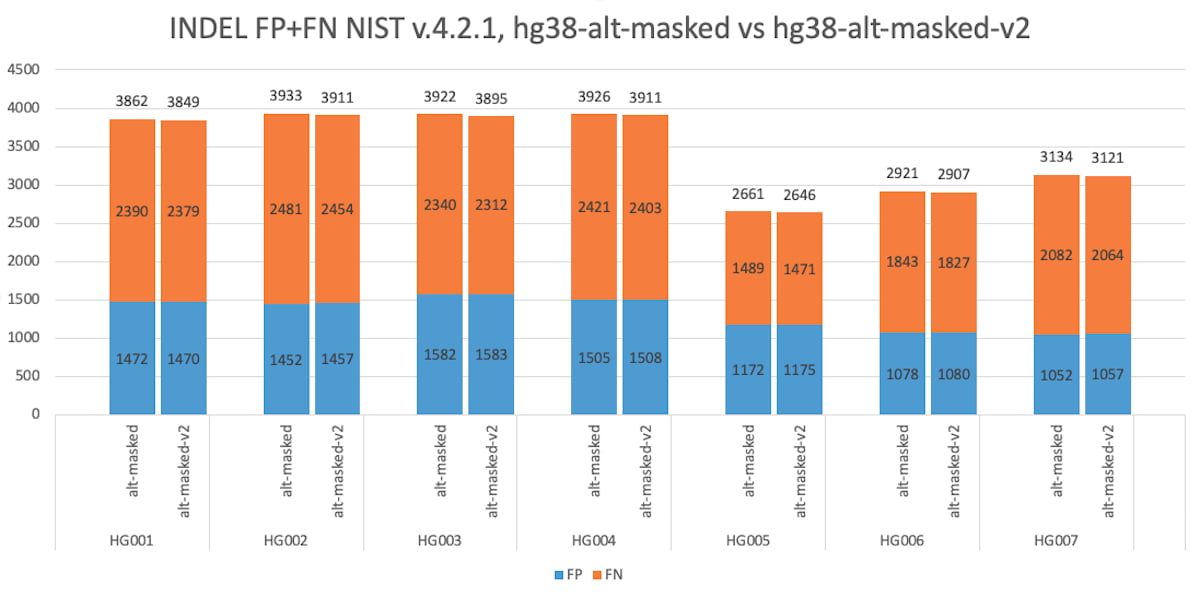
図8:V.4.2.1 NIST真理値セットを使用して全ゲノムで評価したGIABマスクリファレンスによる精度改善。Indelについて、hg38-alt-maskedとhg38-alt-masked-V2を比較
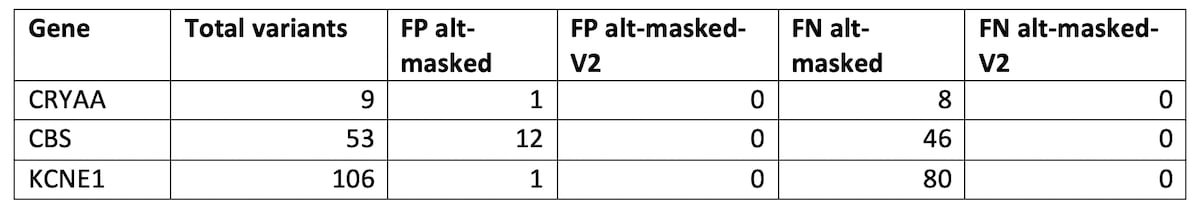
表4:CMRG v.1.0.0の真理値セットを用いて、GIABのマスキングによって影響を受ける遺伝子におけるHG002のSNP精度向上を評価し、hg38-alt-maskedとhg38-alt-masked-V2を比較した。
今後の新しいデコイコンティグの詳細:alt-masked-V3に向けて
マッピングとバリアントコーリングの精度を向上させることができる2つ目のアプローチは、デコイシーケンスの使用です。GIABとBaylor College of Medicine [8]の共同研究により、GRCh38/hg38リファレンスに新たなデコイを追加することで、誤って統合された領域の修正が進められています。例えば、本来は相同な領域が2つ存在すべきにもかかわらず、リファレンスには1つのコピーしか含まれていないケースが該当します。類似しているが同一ではないデコイは、リードを誤ったシーケンスコピーにマッピングする代わりに代替マッピングロケーションを提供することで、偽陽性バリアントを削除します。
リファレンスに対する3番目の改善計画は、集団ハプロタイプの拡張セットに基づく新しいグラフALTコンティグの導入です。現在、DRAGENが使用しているグラフゲノムは、既に精度を向上させる優れた能力を示しています。集団ベースのハプロタイプのリストをさらに拡大し、より多様なゲノム領域や民族を網羅する機会があります。目標は、すべての集団で精度を最大化できる単一のグラフゲノムを構築することです。これにより、個人に適さないグラフを使用したり、集団固有のグラフリファレンスを生成する必要がなくなります。
カスタムマスクリファレンスの作成
hg38-alt-maskedリファレンスをダウンロードできます[3]。ハッシュテーブルは、次のコマンドを使用して構築することもできます。
dragen --ht-reference hg38.fa --ht-alt-aware-validate=true --ht-num-threads=40 --build-hash-table=true --ht-build-rna-hashtable=true --enable-cnv=true --ht-mask-bed /opt/edico/fasta_mask/hg38_alt_mask.bed --output-directory hg38_alt_masked
DRAGEN 3.9以降では、HTビルドのコマンドラインでリフトオーバーやマスクベッドが指定されていない場合、初期設定の動作として自動的にalt-maskedベッドが適用され、標準でhg38-alt-masked(またはhg19-alt-masked)リファレンスが生成されます。alt-maskingはGRCH37またはhs37d5には適用されません。これらのリファレンスにはALTコンティグがネイティブに含まれないためです。
DRAGENは、DRAGENにパッケージ化されたhg38_alt_mask.bedファイルを修正するか、ユーザーが作成することで、カスタムマスクゲノムを作成する機能を提供します。DRAGENは、ベッドファイルに含まれる位置をマッピングの目的でFASTQ内でN(不明な塩基)として処理することにより、ハッシュテーブルを作成します。これにより、リファレンスを直接変更するのと同じハッシュテーブルが作成されます。FASTAにないコンティグがベッドファイル内に存在する場合、DRAGEN 3.9は停止します。今後のバージョンでは、DRAGENは停止せず、存在する領域のみをマスクします。
hg19に関する注記:
hg19には一連のネイティブALTコンティグも含まれているため、alt-awareとalt-maskedの比較は、上述のhg38についてと同じ内容がhg19に適用されます。
結果の概要と謝辞
さまざまなリードテクノロジーのマッピングと小規模バリアントコールの精度の評価がゲノムのより複雑な領域にまで及ぶため、発見や改善に遅れないように、新しい改良版を用いてリファレンスゲノムを維持および更新することが重要です。本稿では、過去のDRAGENリリース(3.9以前)の一部として既にリリースされているリファレンスゲノムのさまざまな改善について説明し、今後のDRAGENリリースに含まれる今後のさらなる改善について明らかにしました。
Genome in a Bottle(GIAB)コンソーシアム、Genome Reference Consortium(GRC)、およびTelomere-to-Telomere(T2T)コンソーシアムの皆様の、リファレンスゲノムの改良に向けた継続的な貢献に深く感謝申し上げます。これらの取り組みにより、マッピング精度およびバリアントコールの精度が向上しました。これは、バリアントの探索に役立ち、ヒトの健康を向上させるための医学的判断をサポートするため、困難な医学的に重要な遺伝子において特に重要です。
学術用途向けの詳細情報またはDRAGEN試用版ライセンスについては、dragen-info@illumina.comまでお問い合わせください。
注釈
- Wagner et al. Towards a Comprehensive Variation Benchmark for Challenging Medically-Relevant Autosomal Genes. BioRxiv (2021) doi:2021:09.07
- DRAGEN Wins at PrecisionFDA Truth Challenge V2 Showcase Accuracy Gains from Alt-aware Mapping and Graph Reference Genomes
- https://support.illumina.com/sequencing/sequencing_software/dragen-bio-it-platform/product_files.html
- http://ftp.1000genomes.ebi.ac.uk/vol1/ftp/technical/reference/GRCh38_reference_genome/GRCh38_full_analysis_set_plus_decoy_hla.fa
- https://github.com/Illumina/DRAGMAP/
- Wagner et al. Benchmarking challenging small variants with linked and long reads. BioRxiv (2021) doi:2020:07.24
- Nurk et al. A complete reference genome improves analysis of human genetic variation. BioRxiv(2021年)doi:2021.05.26.445798
- Publication pending.