はじめに
ショートタンデムリピート(STR)は、ゲノム全体に存在し、ほとんど(92%)のヒト遺伝子のエクソンに存在します1。ほとんどのSTRの生物学的機能は不明ですが2、リピート伸長(RE)疾患と総称される遺伝性疾患クラスにおいて、一部のSTRをヒトの疾患1に結びつけるエビデンスがあります。これまでに、ハンチントン病、筋萎縮性側索硬化症(ALS)、前頭側頭型認知症(FTD)、脆弱X症候群など、40を超える神経遺伝疾患がRE疾患として同定されています。3 ゲノム全体で臨床的に重要なREを同定する能力は、RE疾患を診断し、医学的に関連するゲノム領域の包括的な評価を提供するために不可欠です。RE評価の現在の対策には、PCRベースまたはサザンブロットアッセイなどのターゲット化された方法6がありますが、これらは費用と時間を要する可能性があります。
PCRフリー全ゲノムシーケンス(WGS)は、ほぼ偏りのないゲノム全体のサーベイを提供します。多くの臨床検査室では、一塩基多型(SNP)や挿入/欠失(Indel)、コピー数バリアント(CNV)などの小規模バリアントを同定するための診断ツールとしてWGSをすでに導入していますが、STR拡張などのより複雑な構造多型(SV)には導入していません。反復配列の評価においては、拡張されたSTRバリアントを正確にコールするためのツールの不足や、ツールの性能に関する分析的妥当性の証拠の不足が、臨床応用の主な制約となっています。
ExpansionHunterソフトウェアパッケージなどのSTRのジェノタイピングに特化したアルゴリズムの最近の進歩7、8により、WGSは、高感度かつ高い特異度でゲノム全体のREを確実に検出できることが示されています9。WGSとExpansionHunterの組み合わせにより、これまで診断されていなかった神経性RE疾患が明らかになりました。これにはRE疾患の家族歴がない、またはその疑いがない患者が含まれます9。Association for Medical Pathology(AMP)および米国臨床病理医協会(CAP)の推奨に従い10、トレーニングを受けた専門家がリードレベルの証拠を解析する目視点検ステップを追加することで、ジェノタイプコールの正確性をさらに裏付けることができます。小規模バリアントの場合、一般的にはIntegrated Genome Viewer(IGV)11を使用してアライメントを解析することで目視点検が行われますが、REなどのリファレンスゲノムと大きく異なる複雑なバリアントには、これらのバリアント用に特別に設計された視覚化ツールが必要です。本稿では、病原性REを検出するためのWGSの検証と実装について説明し、ソフトウェア対応の目視点検ステップを臨床パイプラインに追加することで、偽陽性結果の数が大幅に減少することを示します。
ExpansionHunterによるREのコール
ExpansionHunterは、動的グラフリファレンスゲノムを作成し、その中でリピートをループ構造として表現することで、WGSデータから特定の座位におけるリピートサイズを推定します。シーケンスリードはこのダイナミックグラフにアライメントされます8。また、リピートの長さがシーケンスデータのリード長(例:150 bp)よりも大幅に短い場合、スパニングリードが存在するため、それを利用してリピートサイズを正確に同定することが可能です。 リピートの長さがリード長よりも長い場合、完全にリピート内に入るリード(インリピートリード、またはIRR)を特定し、カウントしてリピートのサイズを確率的に推定することができます。
REViewerによるデータの可視化
ラボの慣行には、データの目視点検のためのIGVなどのツールの使用が含まれます。しかし、これらのツールは、データを標準のリファレンスゲノムにアライメントすることに依存しており、これは大量の追加シーケンスの挿入を含む可能性のあるより大きなREにとって問題となる可能性があります。IGVソフトウェアとRE調査のギャップに対処するため、イルミナはRepeat Expansion Viewer(REViewer)を開発しました。これは、ExpansionHunterによって特定されたリピートを含むWGSリードの静的視覚化を行うソフトウェアツールです12。REViewer内で、リードはExpansionHunterで特定された遺伝型によって表される2つのハプロタイプにアライメントされ、リードのパイルアップは静止画像としてプロットされます。得られたプロットにより、ハプロタイプと、対応するExpansionHunter遺伝型のリードパイルアップを直接可視化できます(図1)。この視覚化により、ユーザーは領域にアライメントされたシーケンスリードを同定して調査できるため、IGVを使用して小規模バリアントコールを視覚的に確認する方法と同様に、ExpansionHunterコールの追加評価が可能になります。この視覚化に基づいて、ExpansionHunterが行ったジェノタイプコールを評価して、偽陽性の可能性や保護的な中断(つまり、FMR1におけるAGG中断)を特定することができます。これにより、検査の適応に応じて、研究室がバリアントの直交的な検証を実施できる可能性があります。
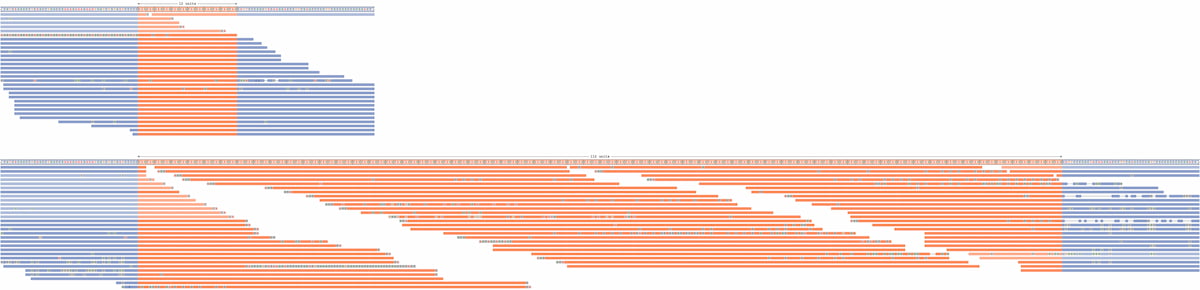
図1:ExpansionHunterを使用して検出されたREの視覚化:DMPKリピートにおける一方のアリルでのリピート拡張を示すリードパイルアップ。REViewerはリピート全体にリードを分散させ、ハプロタイプ全体で同様のカバレッジを実現し、拡張リピートの存在を裏付けます。リピート内のリードのアライメント位置はランダムに選択されます。淡い色で描かれたアライメントは、いずれかの対立遺伝子に割り当てることができるリードに対応しています。
WGSパイプラインによりERの同定が増加
ExpansionHunterとREViewerを使用してWGSデータでREを同定できることを検証するため、Genomics England ProjectとIllumina Clinical Services Laboratory(ICSL)のサイエンティストはWGSワークフローを使用して、以前に検査された793件のサンプルで最も一般的な遺伝性神経性RE疾患のうち13を遡及的に評価しました9。ExpansionHunterの出力をベンチマークデータセットと比較した結果、221の拡張アリルのうち215、および1321の正常アリルのうち1316が正しく分類されていることが確認されました。これにより、全体の感度は97.3%(95%信頼区間: 94.2%~99%)、特異度は 99.6%(95%信頼区間: 99.1%~99.9%)であることが示されました。すべてのコールは目視点検され、各コールをサポートするリードの品質に基づいて必要に応じて再分類されました[*]。目視点検および補正[PD1]後、感度は99.1%(95%信頼区間:96.7%~99.9%)、特異度は100%(95% 信頼区間:99.7%~100%)でした(表1)。伸長したコールの視覚化により、偽陽性が検出可能となり、両方のアリルが伸長しているサンプルで1つのアリルのみが伸長したと分類された遺伝子の1つですべての偽陰性アリルを再分類することができました。これらの結果は、臨床検査室で「正常」または「伸長」に分類される特定の座位におけるREの同定に関して、このWGSパイプラインを証明する裏付けを提供します。
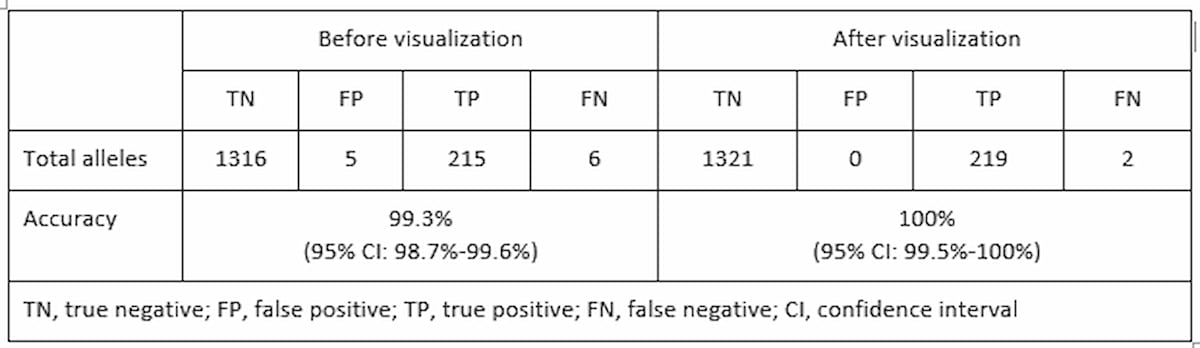
表1:目視点検による精度の向上:目視点検後に検査されたすべての遺伝子座位における正常アリルと伸長アリルの総数に基づく性能9。
RE診断のためのWGSパイプラインの使用例
イルミナのクリニカルサービスラボ(ICSL)は、ExpansionHunterソフトウェアの臨床バリデーションを実施し、希少および未診断の遺伝性疾患患者のWGS臨床検査の一環として、2019年12月にExpansionHunterと目視点検ワークフロー(図2)を実施しました。ICSL検査の定義には、イルミナの慈善活動であるイルミナiHopeプログラムの一環として行われた、21の臨床的に重要なRE疾患の評価が含まれます。このプログラムは、分子検査へのアクセスが限られた個人に臨床WGSを提供することを目指しています。ICSLはこれまでに、筋強直性ジストロフィー(DMPK)、脊髄小脳失調症7型(ATXN7)、脊髄小脳失調症8型(ATXN8OS)、フリードライヒ失調症(FXNの両アリル伸長)など、臨床的に重要なRE疾患に関する7つの臨床報告を発表しています。
ある症例では、キンシャサ大学との協力により行われたコンゴ民主共和国のiHopeプログラムを通じて、ICSLは、脊髄小脳失調、橋小脳萎縮、骨格筋萎縮、錐体路徴候異常、錐体外路筋硬直、筋緊張亢進、構音障害、部分失明を患う3人の成人姉妹からサンプルを受け取りました。さらに、父方家系に類似の症状を持つ家族歴が確認されました。臨床WGS解析とExpansionHunterにより、脊髄小脳失調症7型に関連するATXN7遺伝子におけるCAGトリヌクレオチドリピートの伸長が同定されました。脊髄小脳失調症7型の表現型スペクトルには、青年期または成人期発症の進行性小脳失調および視覚症状が含まれ、3人の姉妹に見られる臨床的特徴と一致しています。これらの所見を記載した臨床報告書が、家族の医師に対し発行されました。
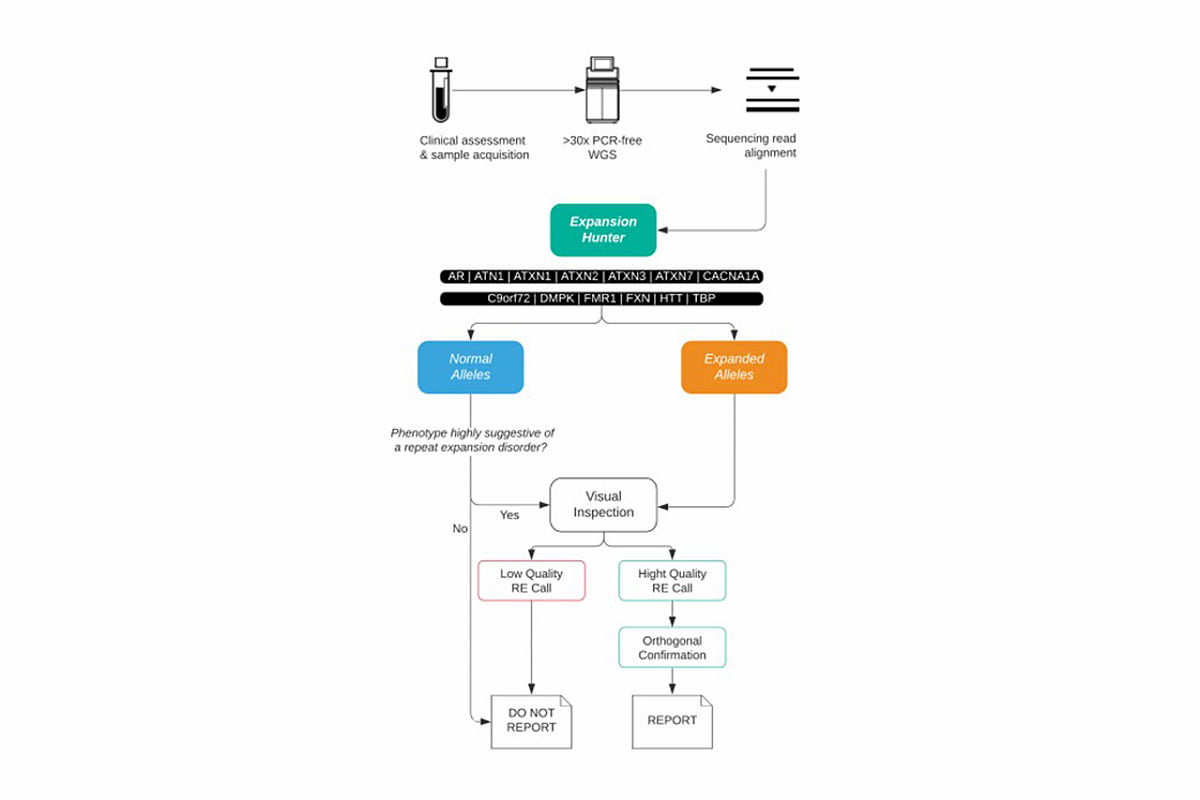
図2:REの診断ツールとしてWGSを使用するための推奨臨床ワークフロー:最初の臨床評価後、WGSデータはExpansionHunterを使用して解析されます。伸長がコールされた場合、または表現型がRE疾患に関連している可能性がある場合は、ExpansionHunterが検討するリードの目視点検が推奨されます。リードの品質が高いと思われる場合、サンプルは直交検査に送られ、リピートの長さを確認し、特性評価を実施します。伸長のコールが十分ではない場合、表現型の重複が強い患者のみを直交確認と特性評価の両方に送ってください。
ディスカッション
ここでは、WGS、ExpansionHunter、およびREViewerを使用して、臨床的に関心のあるRE疾患を同定し、目視点検する臨床パイプラインの有用性について説明しました。現在、このパイプラインは複数の臨床検査室で使用されており、ICSLチームはこのパイプラインを使用して、検証済みのREが同定された臨床報告書を発行しています。ExpansionHunterとREViewerは、それぞれgithub.com/illumina/ExpansionHunterとgithub.com/illumina/REViewerから無料でダウンロードできます。
詳細はこちら
論文「英国における神経性リピート伸長疾患の診断のための全ゲノムシーケンス:遡及的診断の精度と予防的臨床検証研究」を参照してください。
学術用途向けの詳細情報またはDRAGEN試用版ライセンスについては、dragen-info@illumina.comまでお問い合わせください。
関連コンテンツ
希少疾患ゲノミクス:illumina.com/areas-of-interest/genetic-disease/rare-disease-genomics
遺伝性疾患検査:illumina.com/areas-of-interest/genetic-disease
全ゲノムシーケンス:illumina.com/techniques/sequencing/dna-sequencing/whole-genome-sequencing
注釈
- Madsen BE, Villesen P, Wiuf C. Short tandem repeats in human exons: a target for disease mutations. BMC Genomics. 2008;9:410. Published 2008 Sep 12. doi:10.1186/1471-2164-9-410
- Fan H, Chu JY. A brief review of short tandem repeat mutation. 2007;5(1):7-14. doi:10.1016/S1672-0229(07)60009-6
- Paulson HL. Chapter 9-Repeat expansion diseases. In: Geschwind DH, Paulson HL, Klein C, editors. Handbook of Clinical Neurology (Neurogenetics, Part I; vol. 147) [Internet]. Elsevier; 2018:105–123. Available at: http://www.sciencedirect.com/science/article/pii/B9780444632333000099. Accessed October 14, 2020.
- Gossye H, Engelborghs S, Van Broeckhoven C, van der Zee J. C9orf72 Frontotemporal Dementia and/or Amyotrophic Lateral Sclerosis. In: Adam MP, Ardinger HH, Pagon RA, et al., eds. GeneReviews®. Seattle (WA): University of Washington, Seattle; January 8, 2015. Accessed October 14, 2020. Clinical neurogenetics: autosomal dominant spinocerebellar ataxia.
- Shakkottai VG, Fogel BL. Clinical neurogenetics: autosomal dominant spinocerebellar ataxia. Neurol Clin. 2013;31(4):987-1007. doi:10.1016/j.ncl.2013.04.006
- Bird TD. Myotonic Dystrophy Type 1. In: Adam MP, Ardinger HH, Pagon RA, et al, editors. GeneReviews® [Internet]. Seattle (WA): University of Washington, Seattle; 1993. Available at: http://www.ncbi.nlm.nih.gov/books/NBK1165/. Accessed October 14, 2020.
- Dolzhenko E, Bennett MF, Richmond PA, et al. ExpansionHunter Denovo: a computational method for locating known and novel repeat expansions in short-read sequencing data. Genome Biol. 2020;21(1):102. Published 2020 Apr 28. doi:10.1186/s13059-020-02017-z
- Dolzhenko E, Deshpande V, Schlesinger F, et al. ExpansionHunter: a sequence-graph-based tool to analyze variation in short tandem repeat regions. Bioinformatics. 2019;35(22):4754-4756. doi:10.1093/bioinformatics/btz431
- Ibanez K, Polke J, Hagelstrom T, et al. Whole genome sequencing for diagnosis of neurological repeat expansion disorders. bioRxiv 2020.11.06.371716; doi: https://doi.org/10.1101/2020.11.06.371716
- Roy S, Coldren C, Karunamurthy A, et al. Standards and Guidelines for Validating Next-Generation Sequencing Bioinformatics Pipelines: A Joint Recommendation of the Association for Molecular Pathology and the College of American Pathologists. J Mol Diagn. 2018;20(1):4-27. doi:10.1016/j.jmoldx.2017.11.003
- Robinson JT, Thorvaldsdóttir H, Wenger AM, Zehir A, Mesirov JP. Variant Review with the Integrative Genomics Viewer. Cancer Res. 2017;77(21):e31-e34. doi:10.1158/0008-5472.CAN-17-0337
- Dolzhenko E, Weisburd B, Ibanez Garikano K, et al. REViewer: Haplotype-resolved visualization of read alignments in and around tandem repeats. bioRxiv 2021.10.20.465046; doi: https://doi.org/10.1101/2021.10.20.465046