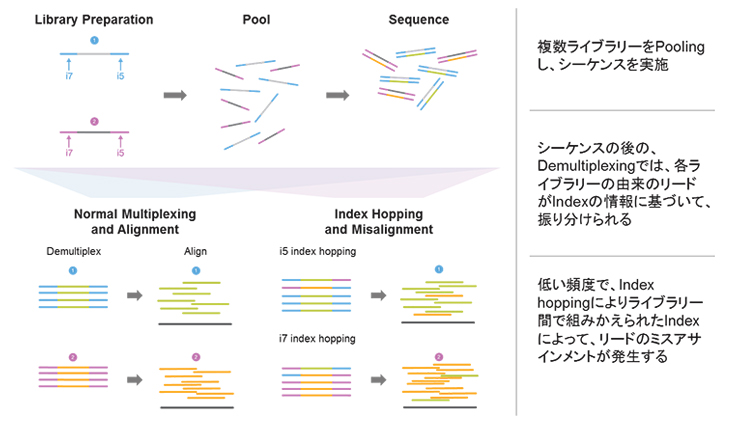
Index hoppingとは
- 複数ライブラリーをプーリングし、シーケンスをした際にライブラリー間でIndex の組み換えが起こり、Demultiplexing 後のライブラリーリードに、別のライブラリー由来のリードが含まれてしまう現象
- 多検体のシーケンス ( Multiplexed Sequence ) をした際に、低頻度ながらライブラリー分子間での組み換えが生じ、結果として本来と異なる誤った Index を持つ配列が生じてしまう現象
結果として、得られたサンプルの配列情報には、他サンプル由来の配列が混入してしまいます。
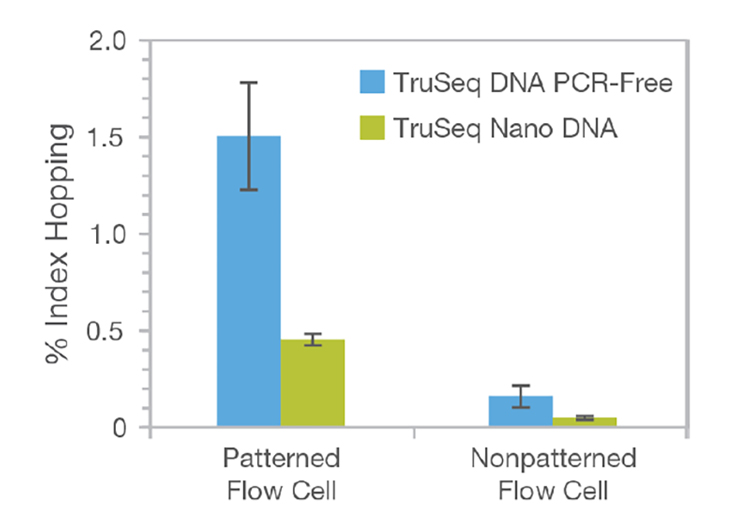
Index hoppingの生じる傾向と頻度例
Index hoppingは、iSeq™ 100、MiSeq™ i100、NextSeq™ 1000/2000、NovaSeq™ 6000、およびNovaSeq X/X plusの、Patterned Flow Cell を採用しているシステムで、比較した場合に高い割合で発生し、MiSeq、MiniSeq™やNextSeq 500/550などの従来型のNon-patterned Flow Cellを用いたシステムでは、非常に低い割合でのみ発生することがわかっています。
また一般に、TruSeq™ DNA PCR-Free Library Prep Kitのようなライゲーションのみを含むライブラリー調製法では、TruSeq Nano DNA Library Prep KitのようなPCR増幅ステップを組み込んだライブラリー調製法と比較した場合に、インデックスホッピングの発生率が高いライブラリーが生成される傾向があります。
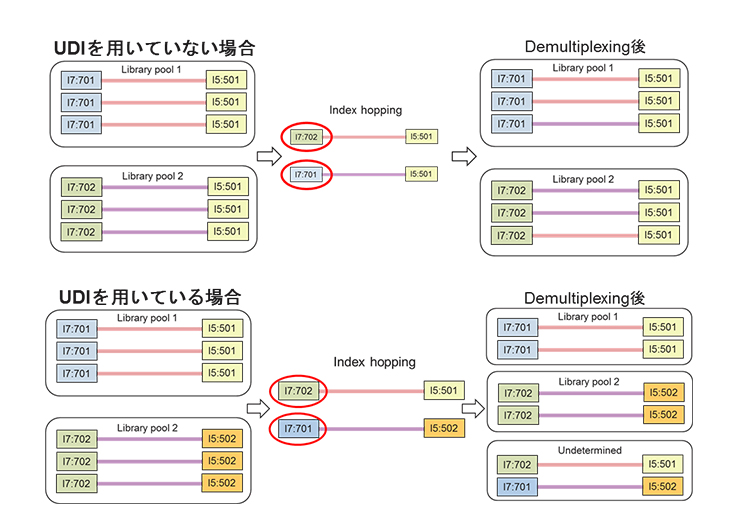
Unique Dual Index (UD Index, UDI)の使用でIndex hoppingを検出
このIndex hoppingの問題は、2か所にIndex配列を持つ(Dual index)ライブラリー調製の際に、2か所の全長にわたってユニークな配列のインデックス=Unique Dual Index (UDI)を採用することにより、大幅に影響を低減することが可能です。
UDIを使用したライブラリーでは、Index hoppingが起きた分子のシーケンスリードは、Demultiplexing時に“undetermined” (どのサンプルライブラリーにも使用していない Index 配列を持つリード)として振り分けられ、各サンプルライブラリーのリードに混入することはありません。
UDIの製品詳細に関しましては、下記のサポートページをご参照ください。
Index hopping の詳細情報に関しましては、下記のサポートページをご参照ください。