はじめに
何百万ものヒトゲノムとエクソームがシーケンスされていますが、疾患の原因となる変異を良性の遺伝子変異と区別することが難しいため、その臨床応用は依然として限られています1,2。その健康に対する有害な影響により、臨床的に重要な遺伝的バリアントは集団では極めてまれである傾向があります3。したがって、集団における高頻度のバリアントの観察は、良性の結果に有利な強力なエビデンスであり2,4、これにより、根絶プロセスによって病原性変異を系統的に同定することができます。多様なヒト集団にわたる共通のバリエーションをアッセイすることは、良性バリアントをカタログ化するための効果的な戦略ですが5、現在のヒトにおける共通のバリエーションの総量は限られています。リファレンスゲノムの7,000万を超える潜在的なミスセンスバリアントのうち、全体のアリル頻度が0.1%を超えるのは1,000分の1程度です5,6。
現代の人類の集団以外では、チンパンジーは次に近い存在種で構成され、99.4%のアミノ酸配列同一性を共有しています7。ヒトとチンパンジーにおけるタンパク質コード配列のほぼ同一性は、チンパンジータンパク質コードバリアントで動作する自然選択も、ヒトの同一変異の適合性に対する結果をモデル化する可能性があることを示唆しています。州ごとに同一の多型が2つの種の適合性に同様に影響を与える場合、チンパンジー集団における高いアリル頻度でのバリアントの存在は、ヒトにおける良性の結果を示し、既知の良性バリアントのカタログを大幅に拡大させる必要があります。これにより、チンパンジーバリアントで検証する必要がある仮説が策定されます。
ヒト集団では一般的な霊長類バリアントが良性である傾向があることを実証しました。トレーニングデータとして、ヒト以外の霊長類6種の集団シーケンスから何十万もの一般的なバリアントを使用して、病原性変異を高精度に予測するディープニューラルネットワークであるPrimateAIを開発しました。
他の霊長類によく見られるバリアントは、ヒトではおおむね良性です
エクソーム凝集コンソーシアム(ExAC)およびゲノム凝集データベース(gnomAD)で収集された123,136人のヒトを含む集合エクソームデータが最近利用可能になったことで、アリル頻度スペクトル5におけるミスセンスおよび同義変異に対する自然選択の影響を測定することができます。シングルトンバリアント(コホートで1回のみ観察)は、交絡因子で調整した後のde novo変異によって予測される予測2.2:1のミスセンス:同義比と密接に一致しています(図1a)8。しかし、アリル頻度が高いほど、自然選択による有害な変異のパージにより、観察されたミスセンスバリアントの数が減少します。
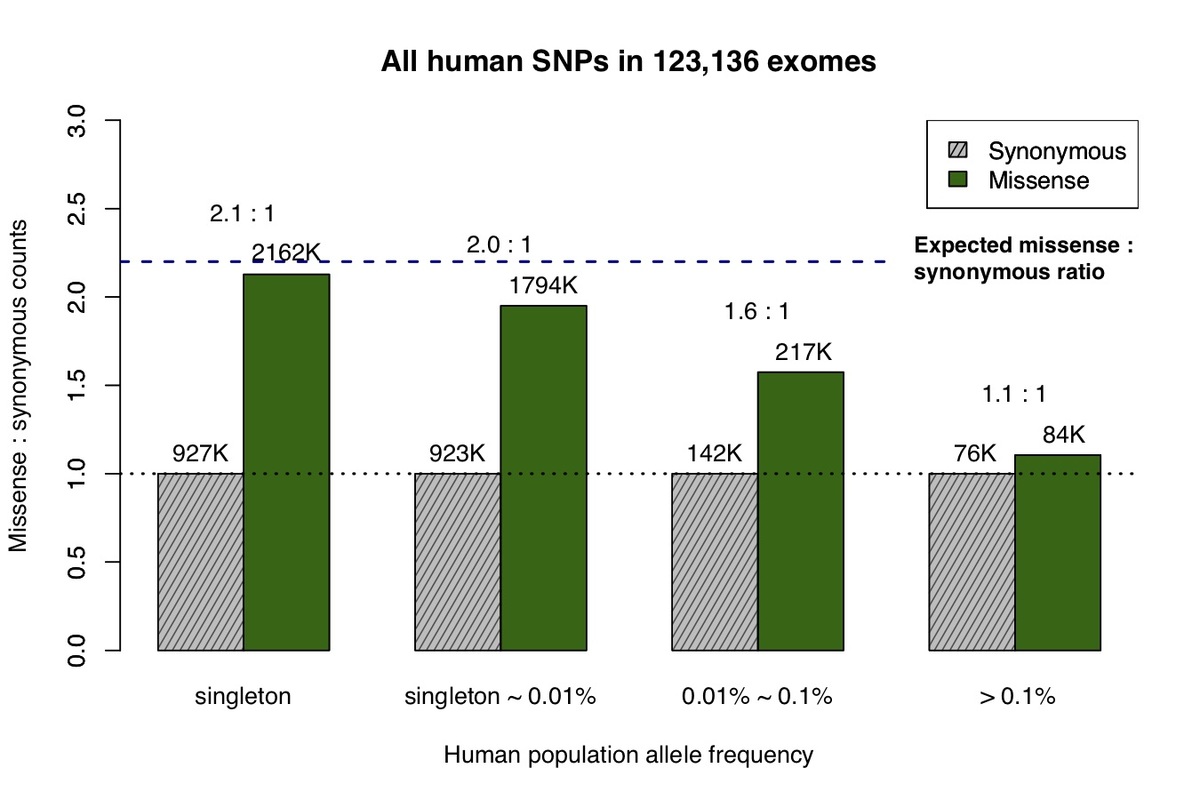
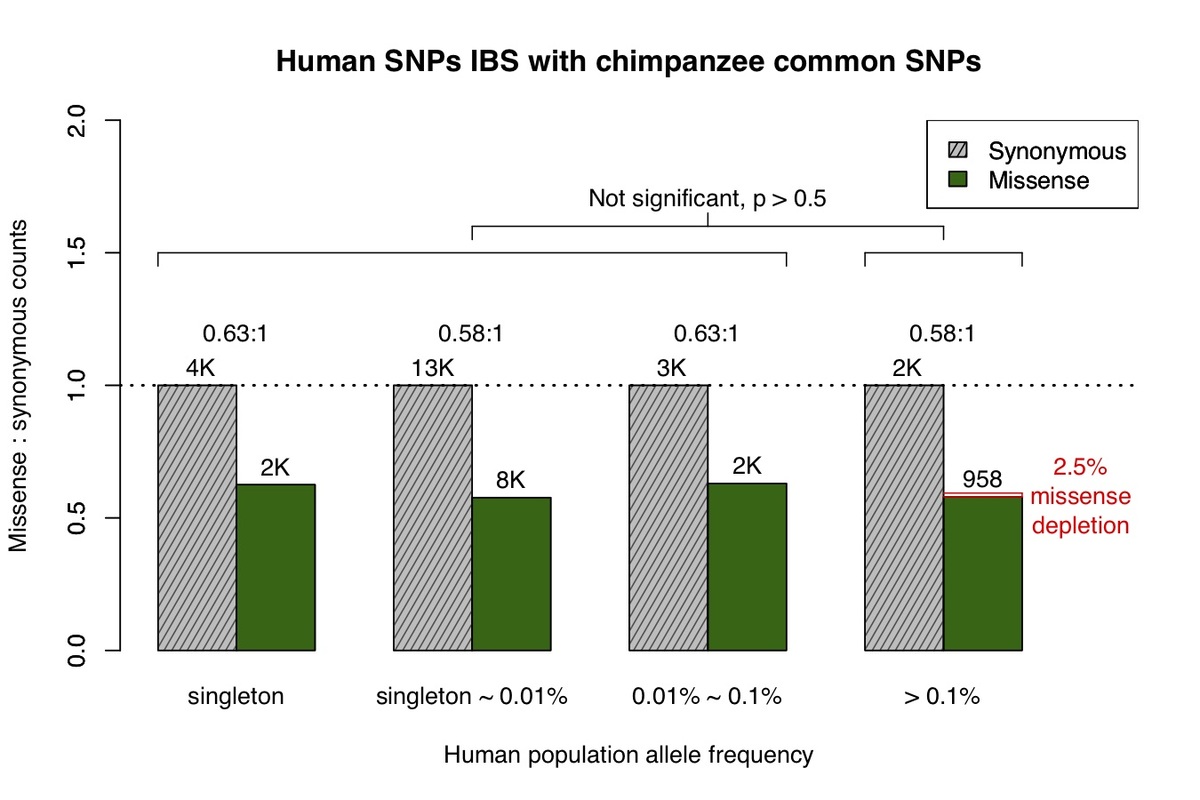
a 、ExAC/gnomADデータベースで観察されたすべてのミスセンスおよび同義バリアントは、アリル頻度によって4つのカテゴリーに分けられました。灰色の網掛けバーは各カテゴリーの同義バリアントの数を表し、濃い緑色のバーはミスセンスバリアントを表します。各バーの高さは、各アリル頻度カテゴリーの同義バリアント数に合わせてスケーリングされます。 b , ヒトミスセンスのアリル頻度スペクトルおよびチンパンジー共通バリアントと同一状態別(IBS)の同義バリアント。
原発バリアントは、gat apeゲノムシーケンスプロジェクトおよびdbSNP 9,10から取得されました。まず、ヒトバリアントと状態が同一である一般的なチンパンジーバリアントを調べ(図1b)、ミスセンス:同義体比がヒトアリル周波数スペクトル全体でほぼ一定であることを発見しました。これは、ヒト集団における一般的なチンパンジーバリアントに対するネガティブな選択がないことと一致しています。一般的なチンパンジーバリアントと状態が同一であるヒトバリアントで観察される低いミスセンス:同義語比は、チンパンジーにおけるより大きな有効集団サイズと一致しており、軽度の有害性変動をより効率的にフィルタリングすることができます11,12。
次に、非ヒト霊長類6種のうち少なくとも1種で変異が観察され、状態ごとに同一であるヒトバリアントを特定しました。6種の各種の変動は、シーケンスされた個体数が限られており、各種で観察されたミスセンス:同義比が低いことに基づいて、主に一般的なバリアントを表しています。チンパンジーと同様に、6種の非ヒト霊長類由来のバリアントのミスセンス:同義体比は、ヒトのアリル頻度スペクトル全体でほぼ等しいことがわかりました。ただし、一般的なアリル頻度でミスセンスバリエーションが軽度に枯渇したこと(図2)は例外で、これは少数の希少バリアントが含まれていることから予想されます。
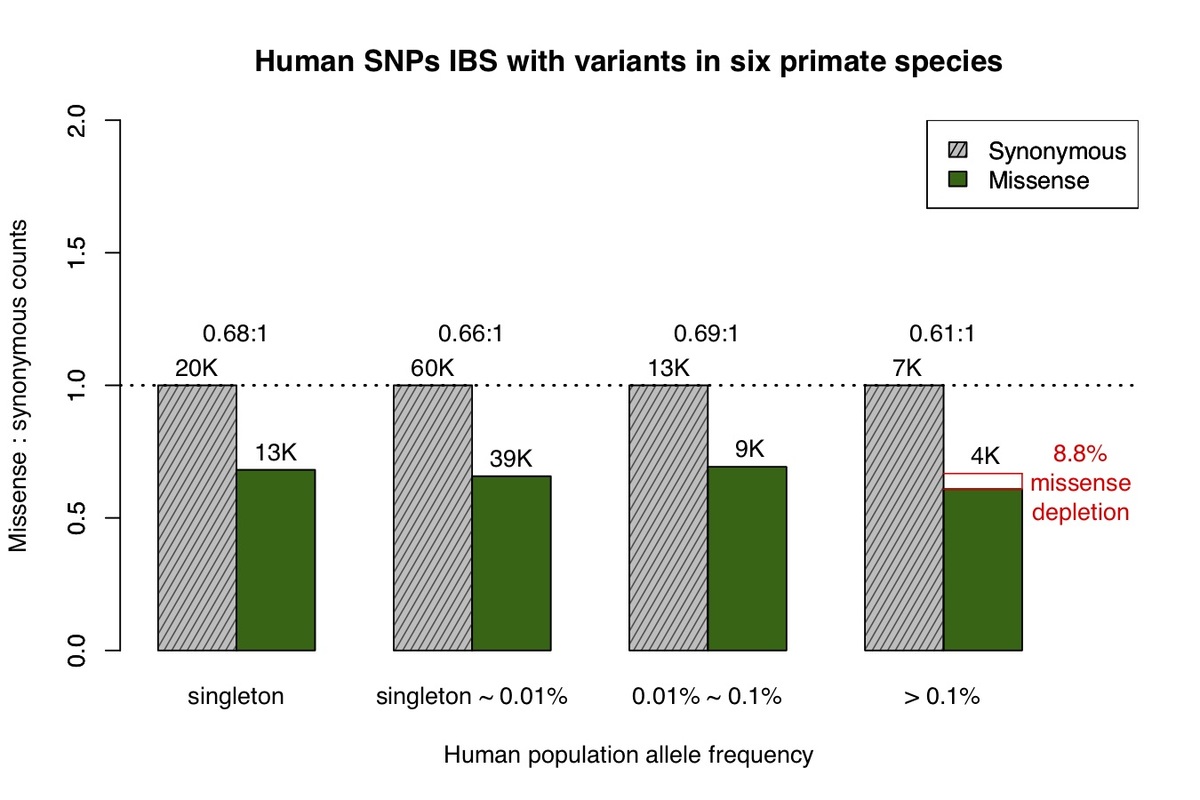
図2。図1と同様に、非ヒト霊長類種の少なくとも1つで観察されるヒトミスセンスおよび同義バリアントのアリル周波数スペクトル。
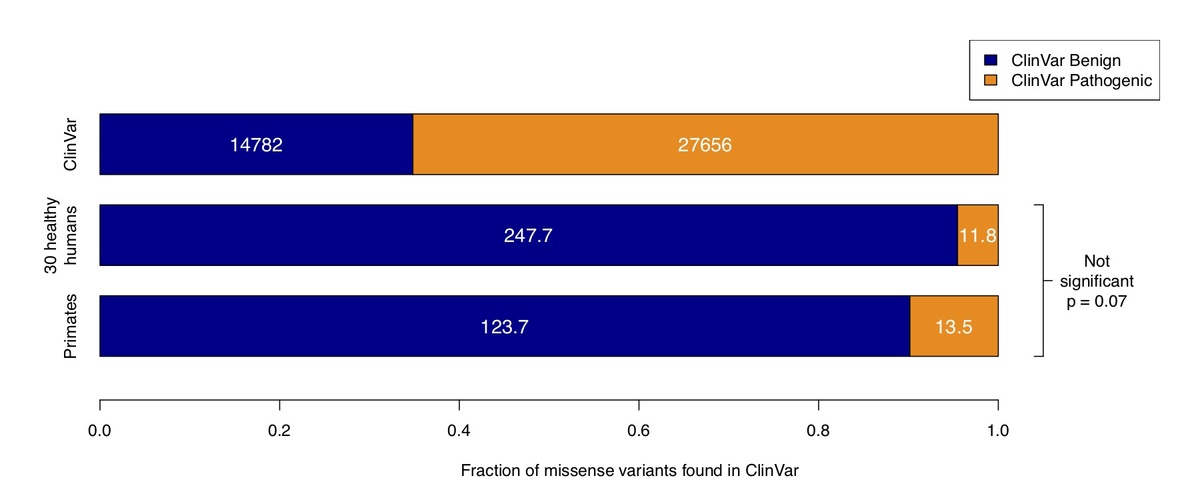
図3。ExAC/gnomADアリル頻度(中央の列)からサンプリングしてシミュレーションしたヒト30例のコホートにおけるClinVarバリアントと比較した、ClinVarデータベース全体(最上列)における良性および病原性のミスセンスバリアントのカウント、霊長類で観察されたバリアント(最下列)との比較。矛盾する良性および病原性のアサーションと、重要性が不明な場合にのみアノテーションされたバリアントは除外された。
バリアント病原性分類のためのディープラーニングネットワーク
臨床アプリケーションにおけるバリアント分類の重要性は、問題に対処するために教師あり機械学習を使用する多くの試みにインスピレーションを与えてきましたが、これらの取り組みは、トレーニング14~24のために自信を持ってラベル付けされた良性および病原性バリアントを含む適切なサイズの真実データセットがないことによって妨げられました。ヒト専門家がキュレーションしたバリアントの既存のデータベースはゲノムのごく一部をカバーしており、ClinVarデータベースのバリアントの約50%はわずか200の遺伝子(ヒトタンパク質コード遺伝子の約1%)から来ています。さらに、系統的研究では、多くのヒト専門家のアノテーションが疑わしい裏付けとなる証拠があることが示されており5,25、これは、1人の患者のみで観察される可能性のある希少バリアントの解釈の難しさを浮き彫りにしています。ヒトの解釈バイアスを減らすために、最近の分類子は、一般的なヒト多型またはヒトとチンパンジーの固定置換についてトレーニングされています26~29。しかし、これらの分類子は、ヒトがキュレーションしたデータベースでトレーニングされた以前の分類子の予測スコアの入力にも使用します。これらのさまざまな方法の性能の客観的ベンチマーキングは、独立したバイアスのない真実のデータセットがない場合には困難でした30。
ヒト以外の霊長類6種(チンパンジー、ボノボ、ゴリラ、オランウータン、アカゲザル、マーモセット)からの変動は、一般的なヒトの変異と重複しない300,000を超える固有のミスセンス変異に寄与し、主に、選別を精製するふるいを通して良性の結果の一般的な変異を表し、機械学習のアプローチに利用できるトレーニングデータセットを大幅に拡大しています。各霊長類種は平均で50Kバリアントに相当する量を占め、ClinVarデータベース全体の現在の合計数よりも多くのバリアントが存在します。さらに、このコンテンツには人間の解釈におけるバイアスはありません。
一般的なヒトバリアントと霊長類のバリエーションからなるデータセットを使用して、新しいディープ残差ネットワークであるPrimateAI(https://github.com/Illumina/PrimateAI)をトレーニングしました。これは、目的のバリアントに隣接するアミノ酸配列と、他の種におけるオルソロガスな配列アライメントを入力として取得します(図4a)31。人間が設計した機能を使用する既存の分類器とは異なり、当社のディープラーニングネットワークは一次シーケンスから直接機能を抽出する方法を学習します。タンパク質構造に関する情報を組み込むために、シーケンスのみから二次構造と溶媒アクセシビリティを予測するために別のネットワークをトレーニングし、32,33、これらをサブネットワークとしてフルモデルに含めました(図4b)。結晶化に成功したヒトタンパク質の数が少ないことを考えると、一次配列から構造を推測することは、タンパク質構造が不完全であることや機能的ドメインアノテーションによるバイアスを回避するという利点があります。タンパク質構造を含むネットワークの総深度は、約400,000のトレーニング可能なパラメーターで構成される36層の畳み込み層でした。
良性ラベルのバリアントのみを使用して分類器をトレーニングするために、特定の変異が集団の一般的なバリアントとして観察される可能性が高いかどうかとして予測問題をフレーム化しました。いくつかの要因が、高いアリル頻度でバリアントを観察する確率に影響を与えます。このうち、有害性にのみ関心があります。良性トレーニングセットの各バリアントをラベルなしミスセンス変異と一致させ、交絡因子をコントロールし、良性バリアントとマッチさせたコントロールを区別するためにディープラーニングネットワークをトレーニングしました8。ラベル付けされていないバリアントの数がラベル付けされた良性トレーニングデータセットのサイズを大きく上回るため、コンセンサス予測を得るために、それぞれが良性トレーニングデータセットに一致する異なるラベル付けされていないバリアントのセットを使用して、8つのネットワークを並行してトレーニングしました。
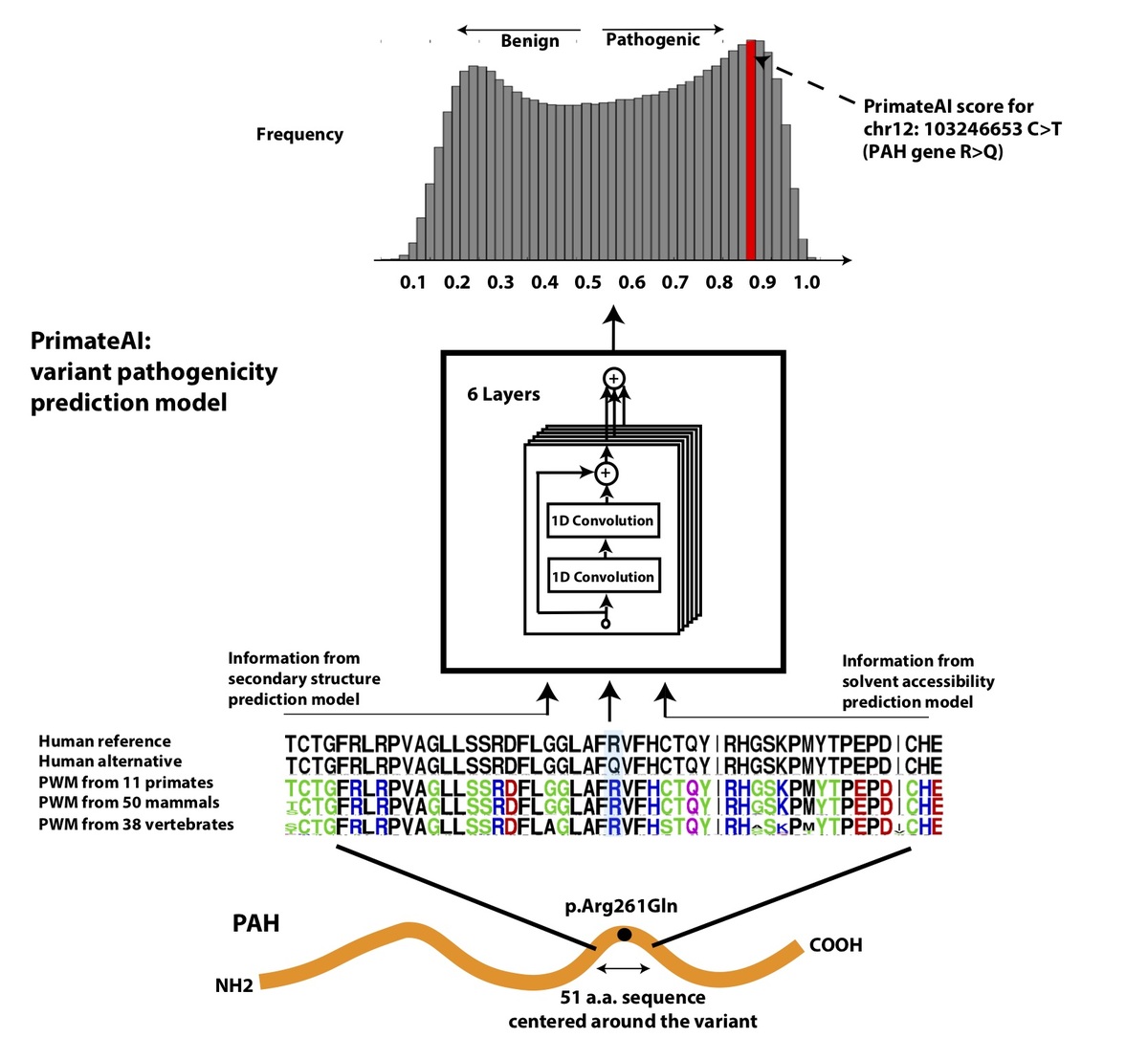
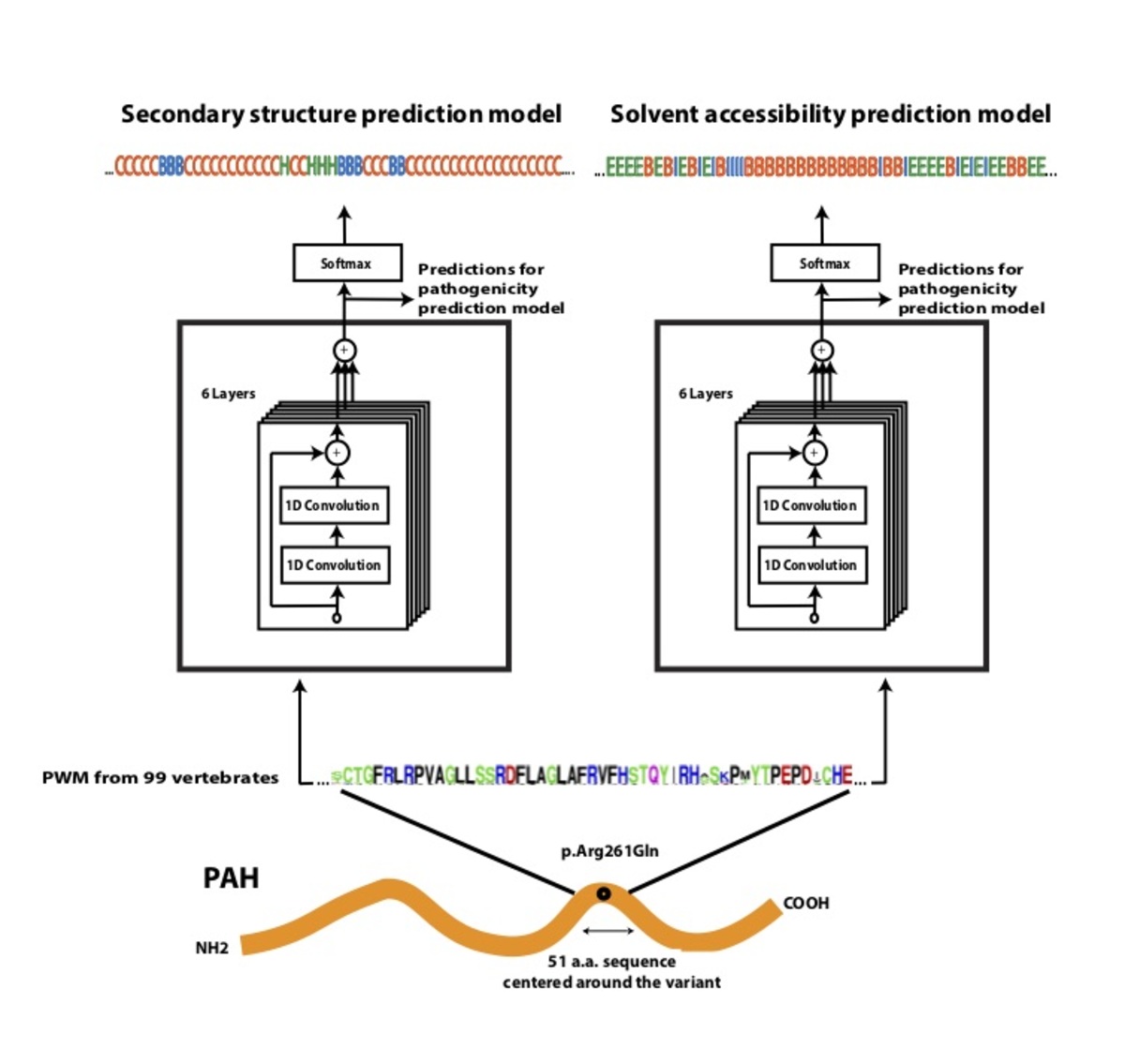
図4。病原性予測のためのディープラーニングネットワークであるPrimateAIのアーキテクチャ。 a、病原性予測のためのディープラーニングネットワークのアーキテクチャであるPrimateAI。PrimateAIスコアとして示される予測病原性は、0(良性)から1(病原性)までのスケールです。ネットワークは、バリアントを中心とするヒトアミノ酸(AA)リファレンスおよび代替シーケンス(51 AA)の入力、99の脊椎動物種から計算された位置重み行列(PWM)保存プロファイル、b 、二次構造および溶媒アクセシビリティ予測ディープラーニングネットワークの出力として、3状態タンパク質二次構造(ヘリックス-H、ベータシート-B、コイル-C)および3状態溶媒アクセシビリティ(埋設-B、中間-I、露出-E)を予測します。
病原性予測の例
ディープラーニングネットワークでは、一次アミノ酸配列のみをインプットとして用い、てんかん、自閉症、知的障害の主要な疾患遺伝子である電位依存性ナトリウムチャネルSCN2A(図5)に示すように、重要なタンパク質機能ドメインの残基に高い病原性スコアを正確に割り当てます。SCN2Aの構造は4つの相同リピートで構成されており、それぞれ6つの膜貫通型ヘリックス(S1~S6)を含んでいます34,35。膜が脱分極すると、正に荷電したS4膜貫通型ヘリックスが膜の細胞外側に向かって移動し、S5/S6細孔形成ドメインがS4-S5リンカーを介して開かれます。S4、 S4-S5リンカー、およびS5ドメインの変異は、早期発症てんかん性脳症と臨床的に関連しており36、ネットワークにより、遺伝子の病原性スコアが最も高く、健康な集団のバリアントでは枯渇すると予測されています。
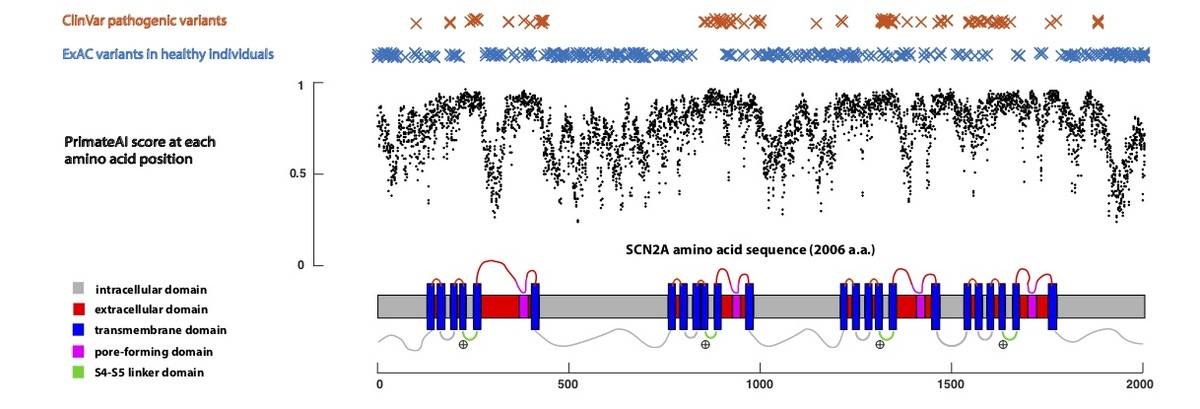
図5 SCN2A遺伝子の各アミノ酸位置における予測病原性スコア。主要な機能ドメインに注釈が付けられています。遺伝子に沿ってプロットされたのは、各アミノ酸位置におけるミスセンス置換の平均PrimateAIスコアです。
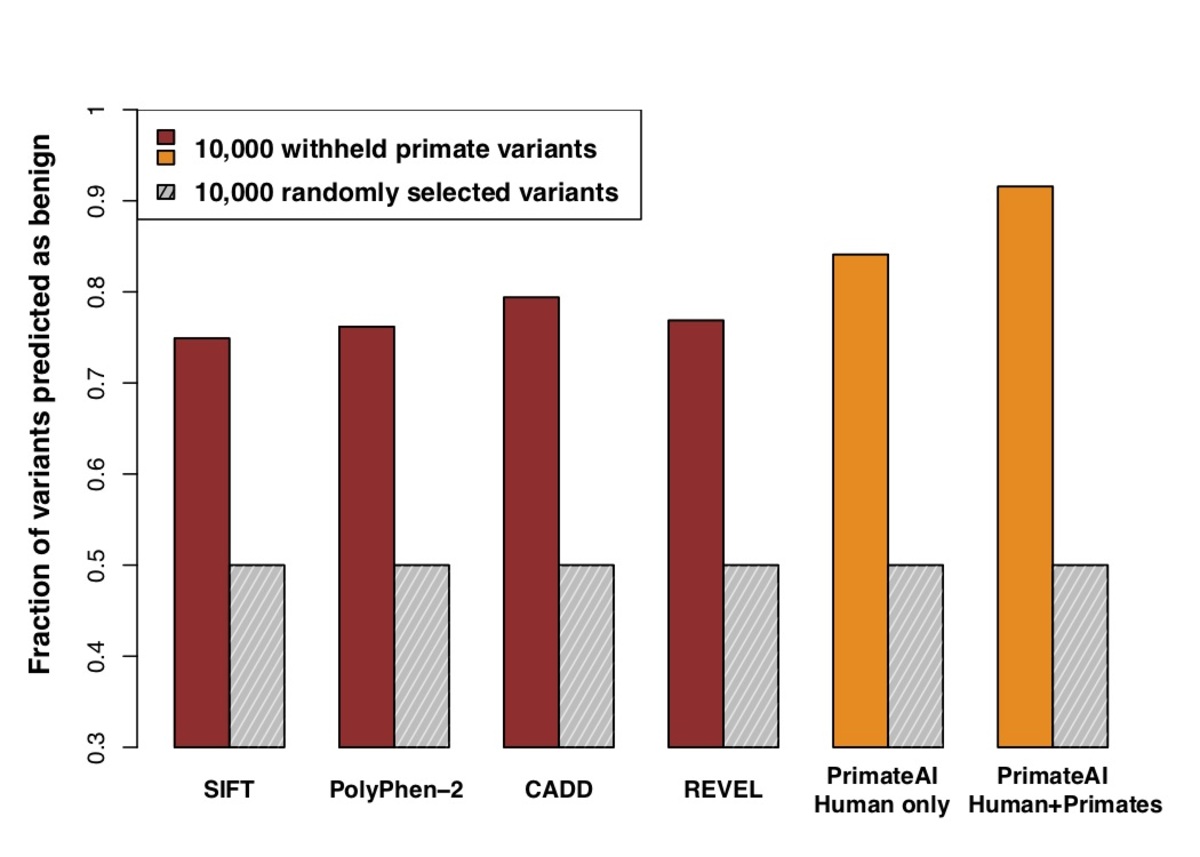
図6。 トレーニングを差し控えた10,000種の一般的な霊長類バリアントのテストセットの良性の結果を予測する際の分類子の比較。y軸は、変異率を一致させた10,000のランダムバリアントのセットで各分類子の閾値をその50パーセンタイルスコアに正規化した後、良性として正しく分類された霊長類バリアントの割合を表します。
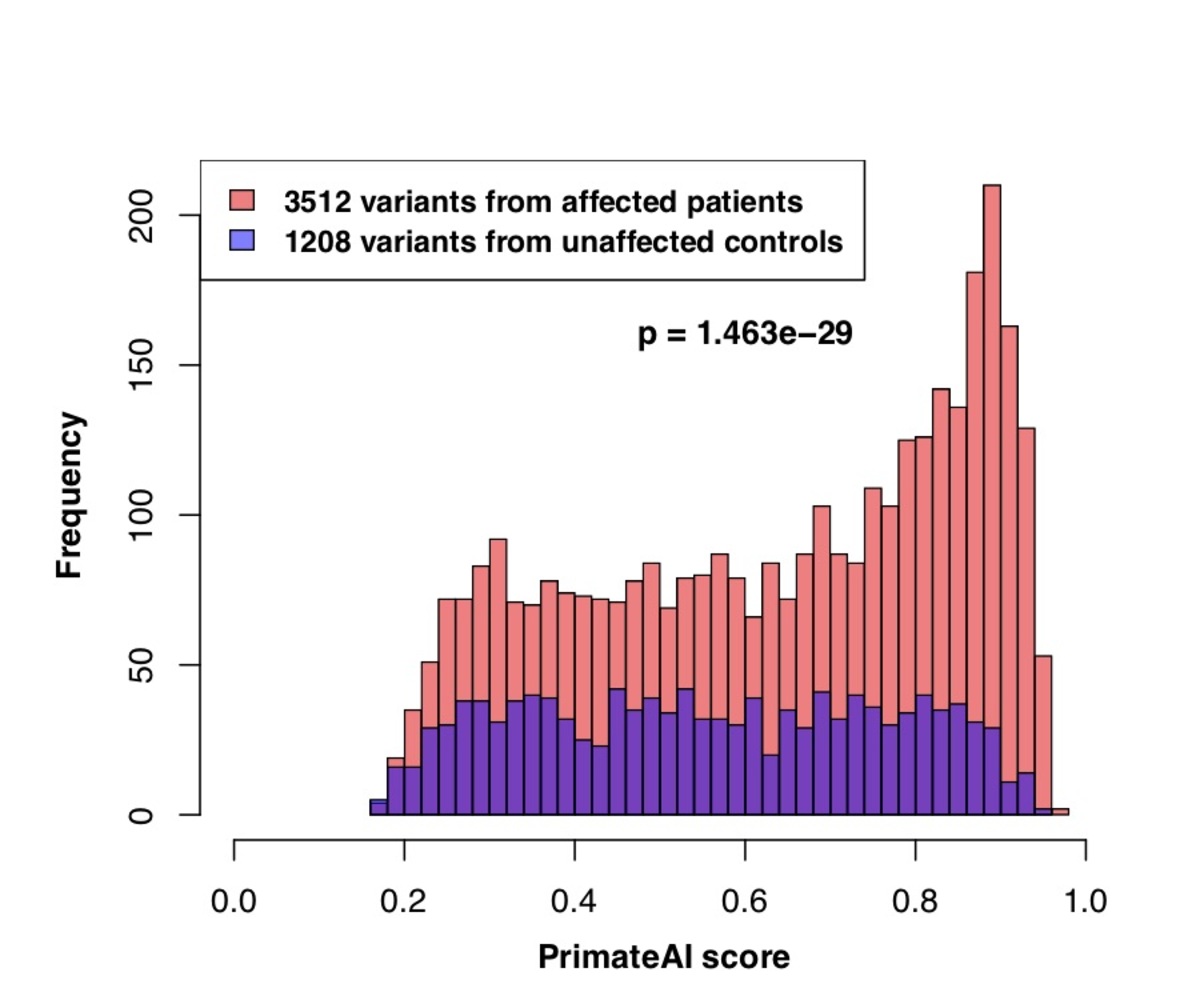
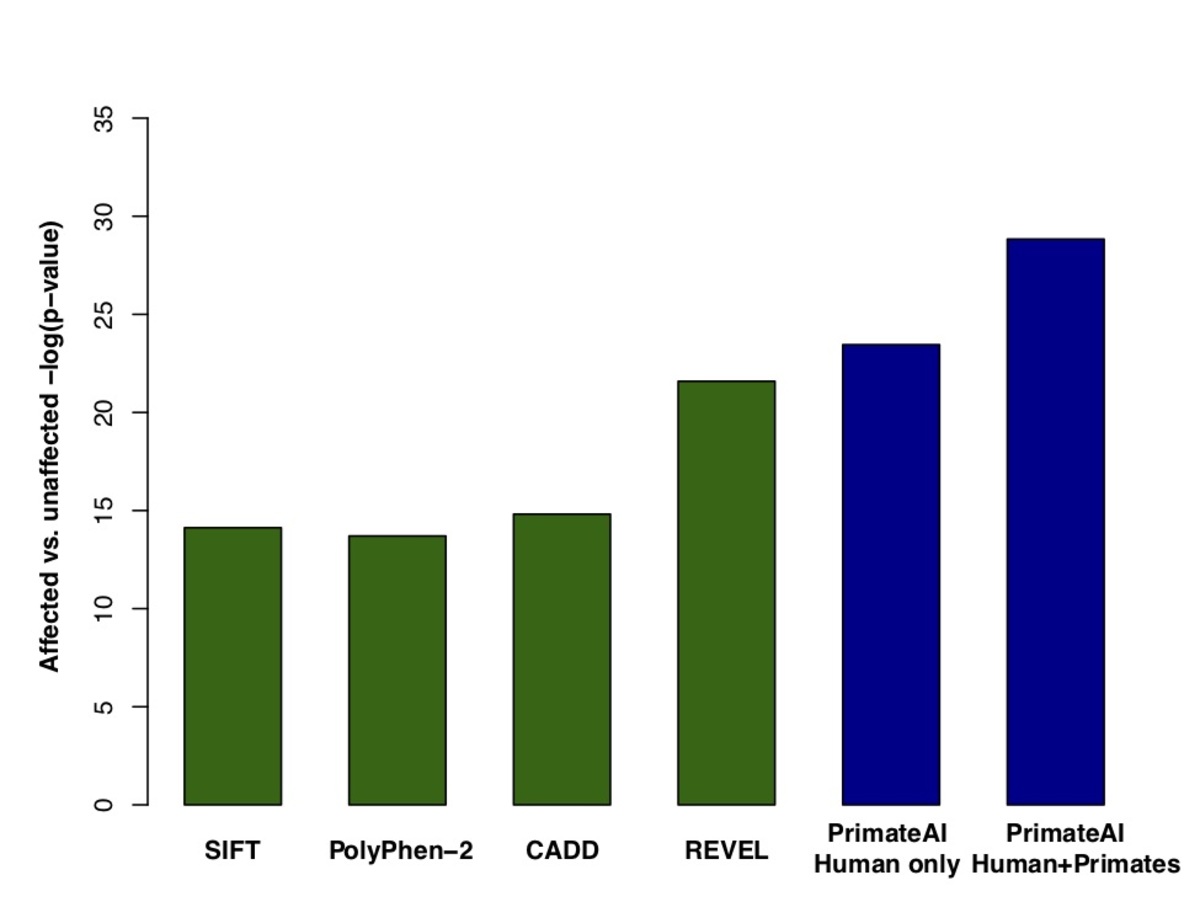
図7. a:DDD患者で発生するde novoミスセンスバリアントと、影響を受けていない兄弟姉妹を比較したPrimateAI予測スコアの分布。対応するWilcoxon順位和p値。 b:DDD症例と対照症例におけるde novoミスセンスバリアントの分離における分類子の比較。Wilcoxon順位和検定のp値を各分類子について示しています。
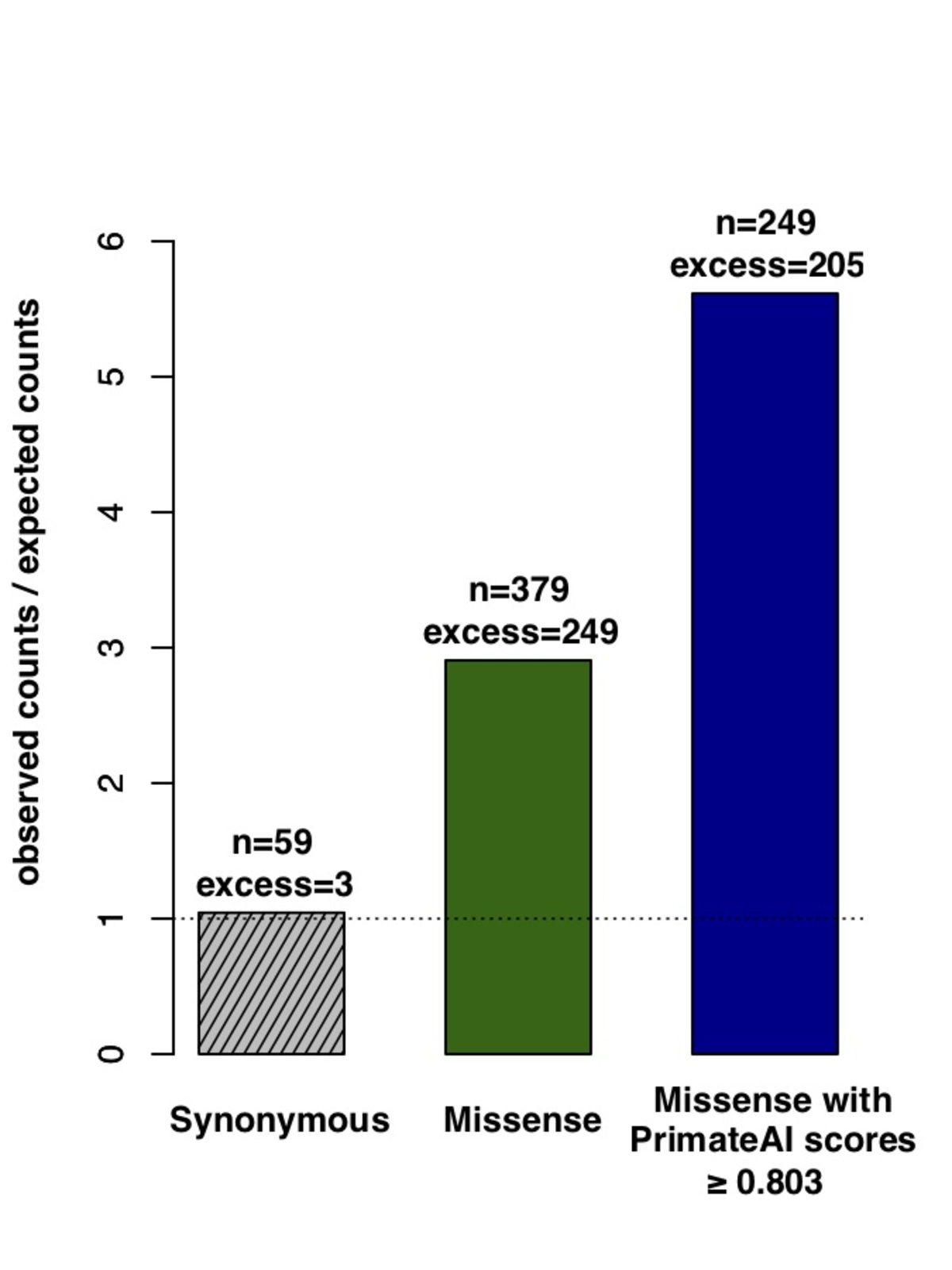
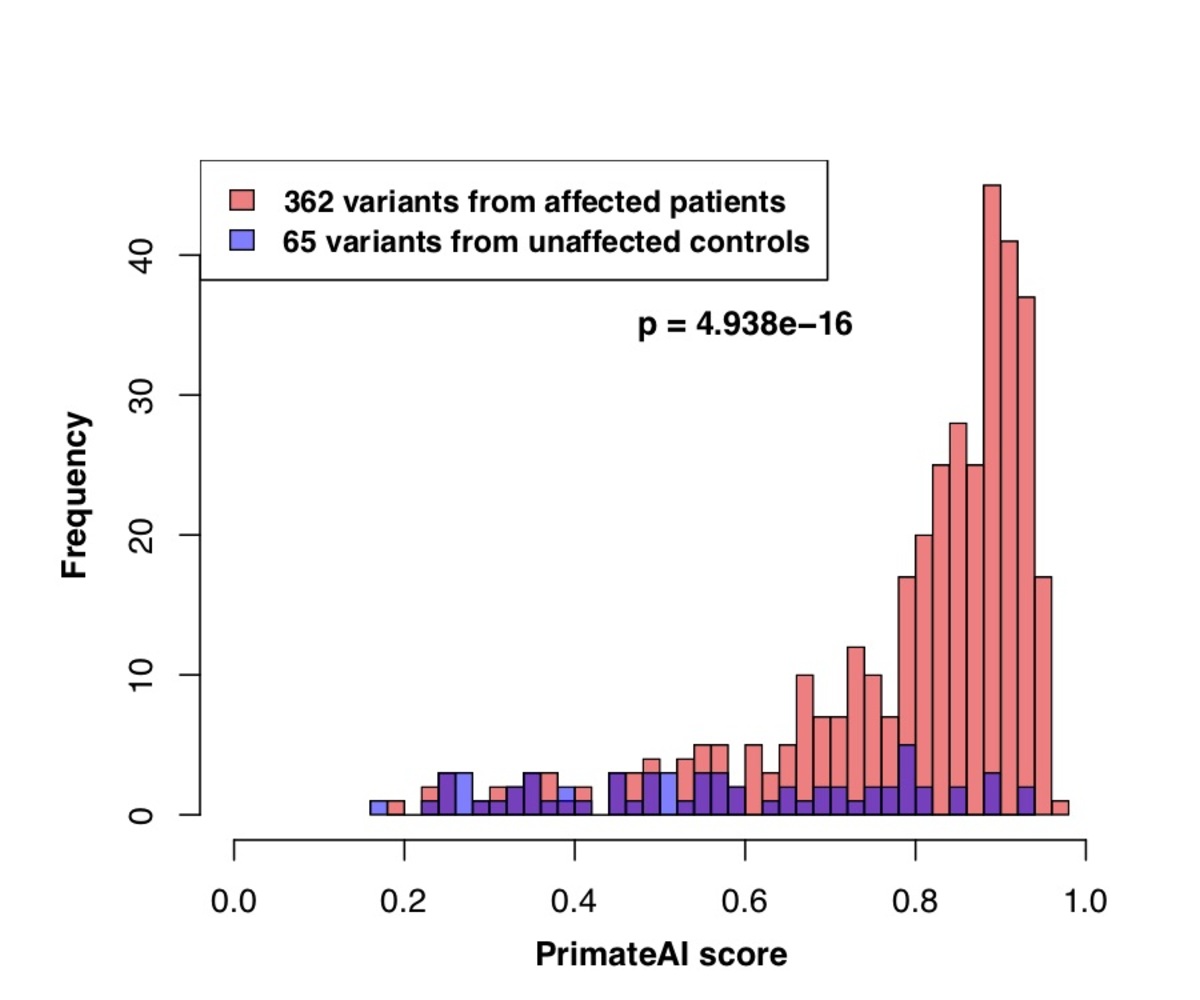
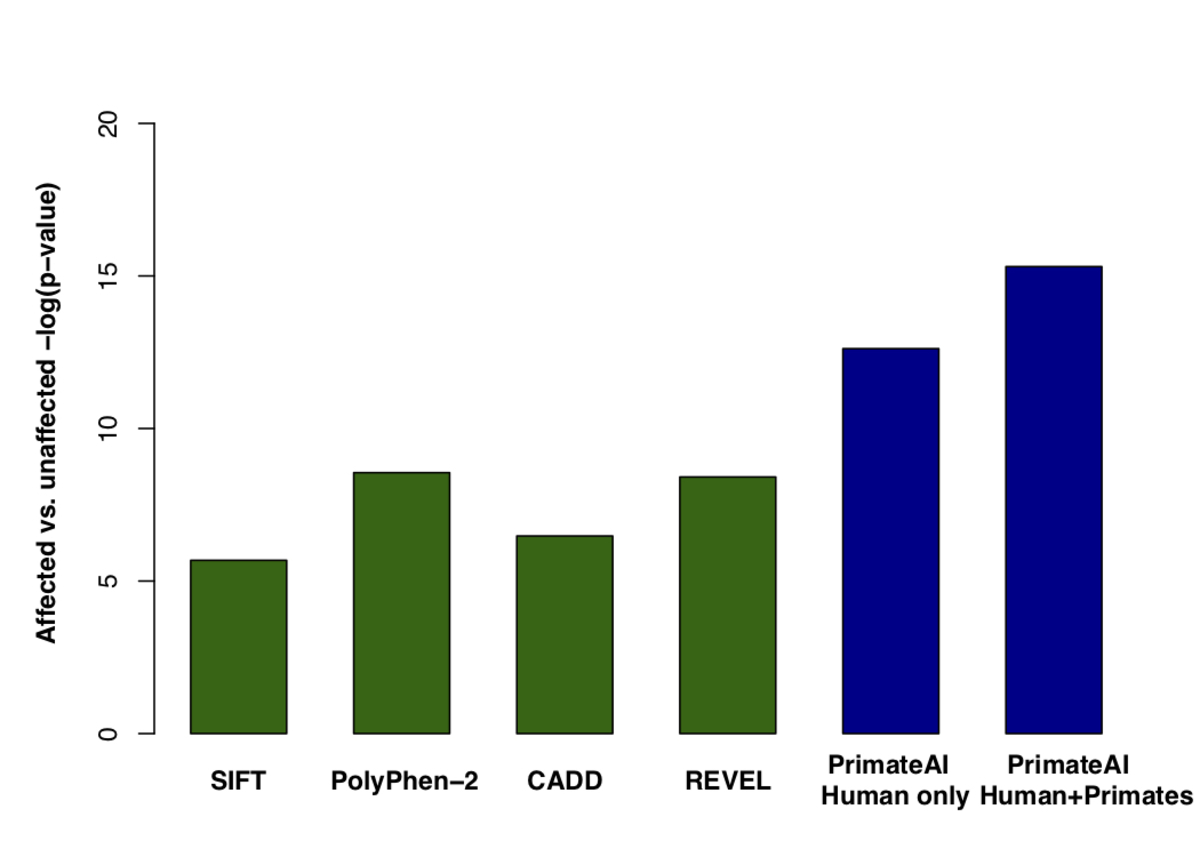
図8。P <0で605のDDD遺伝子内の分類精度。05. a、 DDDコホートの影響を受けた個体において、de novoタンパク質の短縮型変異に有意であった605の関連遺伝子内で、de novoミスセンス変異が期待を上回るよう濃縮されました(p <0.05)。 b 、 DDD患者で発生するde novoミスセンスバリアントと605の関連遺伝子内の影響を受けていない兄弟姉妹のPrimateAI予測スコアの分布 対応するWilcoxon順位和p値 c 、 605遺伝子内の症例と対照におけるde novoミスセンスバリアントの分離におけるさまざまな分類子の比較。y軸は、各分類子のWilcoxon順位和検定のp値を示します。
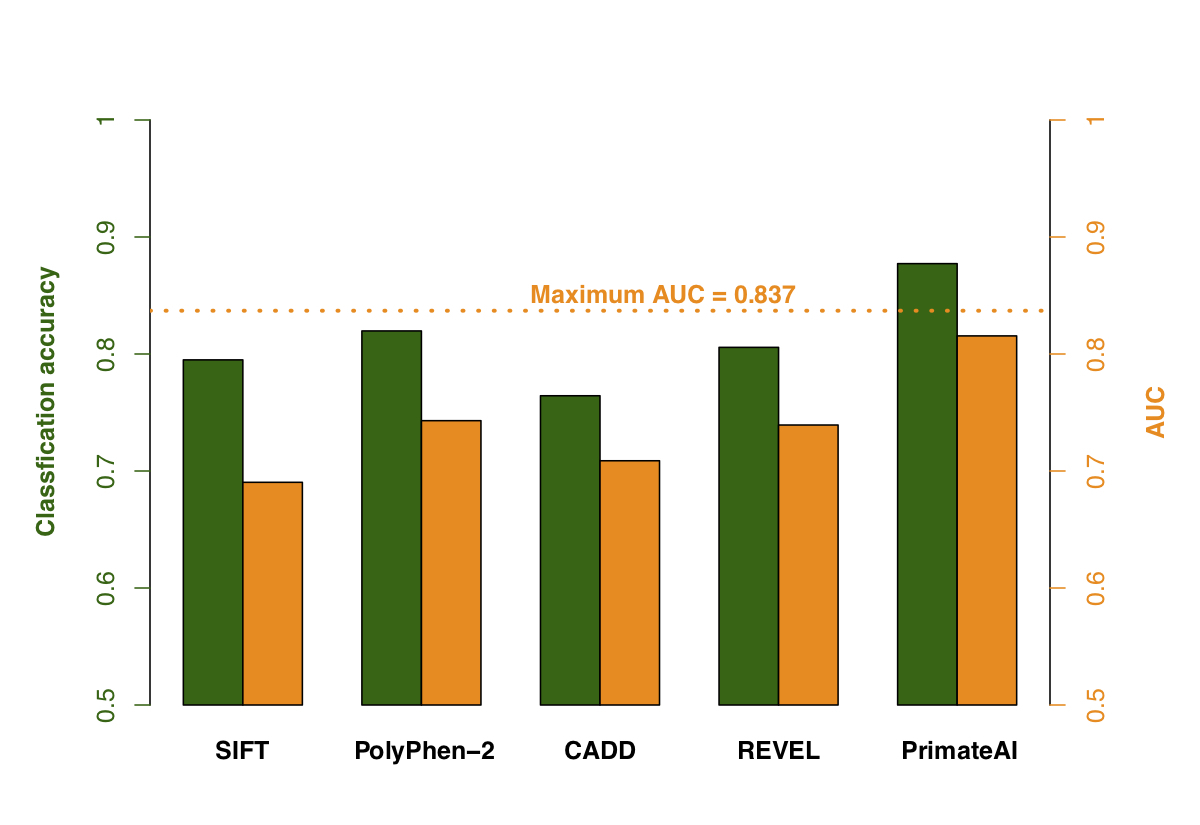
図9. さまざまな分類子の比較。 a , 受信者オペレーター特性(ROC)曲線に示されている性能、および各分類子の曲線下面積(AUC)。 b , 各分類子の分類精度とAUC。示される分類精度は、真の正および真の負のエラー率の平均であり、分類子が図8aの濃縮に基づいて予想されるのと同じ数の病原性および良性バリアントを予測する閾値を使用します。DDD de novoミスセンスバリアントの33%がバックグラウンドであることを考慮すると、完璧な分類子の達成可能な最大AUCは点線で示されます。
我々の結果は、系統的な霊長類集団シーケンスが、現在臨床ゲノムの解釈を制限する、何百万もの意義不明のヒトバリアントを分類するための効果的な戦略であることを示唆しています。保留された一般的な霊長類バリアントと臨床バリアントの両方における当社のディープラーニングネットワークの精度は、ネットワークのトレーニングに使用される良性バリアントの数とともに向上します。追加の霊長類種からの一般的なバリエーションをカタログ化することで、意義が不明な何百万ものバリアントの解釈が改善され、ヒトゲノムシーケンスの臨床的有用性がさらに向上します。
確認
J. K. Pritchard、M. E. Hurles、J. W. Belmont、およびR. E. Greenに、洞察に富んだ話し合いに感謝いたします。Genome Aggregation Database(gnomAD)と、エクソームとゲノムバリアントデータをこのリソースに提供したグループに感謝いたします。Yanjun LiとXiaolin Liは、国立総合医療科学研究所および国立科学財団(助成金CNS- 1747783、CNS- 1624782、およびOAC-1229576)のR01GM110240によって部分的にサポートされました。Laksshman Sundaram、Samskruthi Reddy Padigepati、Jeremy F. McRae、Yanjun Li、Jack A. Kosmicki、Nondas Fritzilas、Jorg Hakenberg、Anindita Dutta、John Shon、Jinbo Xu、Serafim Batzloglou、Xiaolin Liなどの原著作者をここに認めたいと思います。
外部リンク
出版物:https://pubmed.ncbi.nlm.nih.gov/30038395/
ソフトウェア:https://github.com/Illumina/PrimateAI
great apeゲノムプロジェクトから多型をプライミングします:
https://eichlerlab.gs.washington.edu/greatape/data.html
dbSNPデータベースから: https://www.ncbi.nlm.nih.gov/snp/
7,000万バリアントのPrimateAIスコア:https://basespace.illumina.com/s/cPgCSmecvhb4
参考文献
- MacArthur, D. G. et al. ヒト疾患におけるシーケンスバリアントの因果関係を調べるためのガイドライン。 Nature 508, 469-476, doi:10.1038/nature13127(2014)。
- Rehm, H. L., J. S. Berg, L. D. Brooks, C. D. Bustamante, J. P. Evans, M. J. Landrum, D. H. Ledbetter, D. R. Maglott, C. L. Martin, R. L. Nussbaum, S. E. Plon, E. M. Ramos, S. T. Sherry, M. S. Watson. ClinGen--臨床ゲノムリソース。 N. Engl. J. Med. 372, 2235-2242(2015)。
- Bamshad, M. J., S. B. Ng, A. W. Bigham, H. K. Tabor, M. J. Emond, D. A. Nickerson, J. Shendure. メンデル性疾患遺伝子発見のためのツールとしてのエクソームシーケンス。 ネイティブ。Rev. Genet. 12, 745–755(2011年)。
- Richards, S. et al. シーケンスバリアントの解釈に関する基準とガイドライン:American College of Medical Genetics and Genomics and the Association for Molecular Pathologyの共同合意勧告。 Genet Med 17, 405-424, doi:10.1038/gim.2015.30(2015)。
- Lek, M. et al. 60,706人のヒトにおけるタンパク質コーディング遺伝子変異の解析。 Nature 536, 285-291, doi:10.1038/nature19057(2016)。
- Liu, X., X. Jian, E. Boerwinkle. dbNSFP:ヒトの非同義SNPとその機能的予測の軽量データベース。 。 Human Mutation 32, 894–899(2011)。
- Chimpanzee Sequencing Analysis Consortium. チンパンジーゲノムの初期シーケンスおよびヒトゲノムとの比較。 Nature 437, 69-87, doi:10.1038/nature04072(2005)。
- Samocha, K. E. et al. ヒト疾患におけるde novo変異の解釈のためのフレームワーク。 Nat Genet 46, 944-950, doi:10.1038/ng.3050(2014)。
- Sherry, S. T. et al. dbSNP:遺伝的変異のNCBIデータベース。 Nucleic Acids Res 29, 308-311, doi:10.1093/nar/29.1.308(2001)。
- Prado-Martinez, J. et al. ゲノムの多様性と集団の歴史が素晴らしい。 Nature 499, 471-475(2013)。
- Kimura, M. 分子進化のニュートラルな理論。 ケンブリッジ大学プレス、1983年
- de Manuel, M. et al. チンパンジーのゲノム多様性は、ボノボとの古代の混合を明らかにします。 Science 354, 477-481, doi:10.1126/science.aag2602(2016)。
- Landrum, M. J. et al. ClinVar:臨床的に重要なバリアントの解釈の公開アーカイブ。 Nucleic Acids Res 44, D862-868, doi:10.1093/nar/gkv1222(2016)。
- Ng, P. C. & Henikoff, S. 有害なアミノ酸置換の予測。 Genome Res 11, 863-874, doi:10.1101/gr.176601(2001)。
- Adzhubei, I. A. et al. 損傷を与えるミスセンス変異を予測する方法とサーバー。 Nat Methods 7, 248-249, doi:10.1038/nmeth0410-248(2010)。
- Chun, S., J. C. Fay. 3つのヒトゲノム内の有害な変異の同定。 Genome Research 19, 1553-1561(2009)。
- Schwarz, J. M., C. Rödelsperger, M. Schuelke, D. Seelow. MutationTasterは、疾患の原因となる配列変化の可能性を評価します。 ネイティブ。Methods 7, 575–576(2010年)。
- Reva, B., Antipin, Y. & Sander, C. タンパク質変異の機能的影響の予測:がんゲノミクスへの応用。 Nucleic Acids Res 39, e118, doi:10.1093/nar/gkr407(2011)。
- Dong, C. et al. 全エクソームシーケンス研究における非同義SNVの有害性予測方法の比較と統合。 Hum Mol Genet 24, 2125-2137, doi:10.1093/hmg/ddu733(2015)。
- Carter, H., Douville, C., Stenson, P. D., Cooper, D. N. & Karchin, R. バリアント効果スコアリングツールによるメンデル性疾患遺伝子の同定。 BMC Genomics 14 Suppl 3, S3, doi:10.1186/1471-2164-14-S3-S3(2013)。
- Choi, Y., Sims, G. E., Murphy, S., Miller, J. R. & Chan, A. P. アミノ酸置換とインデルの機能効果の予測。 PLoS One 7, e46688, doi:10.1371/journal.pone.0046688(2012)。
- Gulko, B., Hubisz, M. J., Gronau, I. & Siepel, A. ヒトゲノム全体の点変異に対する適合性の結果の確率を計算する方法。 Nat Genet 47, 276-283, doi:10.1038/ng.3196(2015)。
- Shihab, H. A. et al. ノンコーディングおよびコーディングシーケンスのバリエーションによる機能的影響を予測するための統合的アプローチ。 Bioinformatics 31, 1536-1543, doi:10.1093/bioinformatics/btv009(2015)。
- Quang, D., Chen, Y. & Xie, X. DANN:遺伝的バリアントの病原性をアノテーションするためのディープラーニングアプローチ。 Bioinformatics 31, 761-763, doi:10.1093/bioinformatics/btu703(2015)。
- Bell, C. J., D. L. Dinwiddie, N. A. Miller, S. L. Hateley, E. E. Ganusova, J. Midge, R. J. Langley, L. Zhang, C. L. Lee, R. D. Schilkey, J. E. Woodward, H. E. Peckham, G. P. Schroth, R. W. Kim, S. F. Kingsmore. 次世代シーケンサーによる重度の小児劣性遺伝疾患の包括的なキャリア検査。 科学 翻訳 Med. 3、65ra64(2011)。
- Kircher, M., D. M. Witten, P. Jain, B. J. O’Roak, G. M. Cooper, J. Shendure. ヒト遺伝的バリアントの相対的な病原性を推定するための一般的なフレームワーク。 ネイティブ。遺伝子。 46, 310-315(2014年)。
- Smedley, D. et al. メンデル性疾患における病原性制御バリアントの効果的な同定のための全ゲノム解析フレームワーク。 Am J Hum Genet 99, 595-606, doi:10.1016/j.ajhg.2016.07.005(2016)。
- Ioannidis, N. M. et al. REVEL:希少ミスセンスバリアントの病原性を予測するアンサンブル法。 Am J Hum Genet 99, 877-885, doi:10.1016/j.ajhg.2016.08.016(2016)。
- Jagadeesh, K. A., A. M. Wenger, M. J. Berger, H. Guturu, P. D. Stenson, D. N. Cooper, J. A. Bernstein, G. Bejerano. M-CAPは、臨床エクソームにおける意義が不明なバリアントの大部分を高感度で除去します。 Nature Genetics 48, 1581-1586(2016)。
- Grimm, D. G. ミスセンスバリアントの影響を予測するために使用されるツールの評価は、2種類の循環性によって妨げられます。 Human Mutation 36, 513-523 (2015)。
- He, K., X. Zhang, S. Ren, J. Sun. コンピュータービジョンとパターン認識に関するIEEE会議の進行。 IEEE 770-778。
- Heffernan, R. et al. 反復的なディープラーニングにより、二次構造、局所的なバックボーン角度、およびタンパク質の溶媒でアクセス可能な表面積の予測を改善します。 Sci Rep 5, 11476, doi:10.1038/srep11476(2015)。
- Wang, S., J. Peng, J. Ma, J. Xu. 深層畳み込みニューラルフィールドを使用したタンパク質二次構造予測。 Scientific Reports 6, 18962-18962(2016)。
- Payandeh, J., Scheuer, T., Zheng, N. & Catterall, W. A. 電位依存性ナトリウムチャネルの結晶構造。 https://www.nature.com/articles/nature10238
- Shen, H. et al. ほぼ原子分解能での真核生物の電位依存性ナトリウムチャネルの構造。 https://science.sciencemag.org/content/355/6328/eaal4326
- Nakamura, K. et al. 大田原症候群に拡大するSCN2A変異の臨床スペクトル。 Neurology 81, 992-998, doi:10.1212/WNL.0b013e3182a43e57(2013)。
- Vissers, L. E., Gilissen, C. & Veltman, J. A. Genetic studies in intellectual disability and related disorders. Nat Rev Genet 17, 9-18, doi:10.1038/nrg3999(2016)。
- Neale, B. M. et al. 自閉症スペクトラム障害におけるエクソンde novo変異のパターンと割合。 Nature 485, 242-245, doi:10.1038/nature11011(2012)。
- Sanders, S. J. et al. 全エクソームシーケンスによって明らかになったde novo変異は自閉症と強く関連しています。 Nature 485, 237-241, doi:10.1038/nature10945(2012)。
- De Rubeis, S. et al. 自閉症で破壊されたシナプス遺伝子、転写遺伝子、クロマチン遺伝子。 Nature 515, 209-215, doi:10.1038/nature13772(2014)。
- Deciphering Developmental Disorders Study. 発達障害の新規遺伝的原因の大規模な発見。 Nature 519, 223-228, doi:10.1038/nature14135(2015)。
- Deciphering Developmental Disorders Study. 発達障害におけるde novo変異の有病率と構造。 Nature 542, 433-438, doi:10.1038/nature21062(2017)。
- Iossifov, I. et al. 自閉症スペクトラム障害に対するde novoコーディング変異の寄与。 Nature 515, 216-221, doi:10.1038/nature13908(2014)。
- Zhu, X., Need, A. C., Petrovski, S. & Goldstein, D. B. 1つの遺伝子、多くの神経精神疾患:メンデル性疾患からの教訓。 Nat Neurosci 17, 773-781, doi:10.1038/nn.3713(2014)。