はじめに
臨床で処方された多数の薬剤の薬物代謝率には個人間で大きなばらつきがあります。薬物代謝におけるこのばらつきの大きな要因は、薬物代謝酵素の遺伝子構成であり、したがってこれらの酵素をコードする遺伝子(すなわち、ファーマコゲネス)のジェノタイピングは個別化医療の重要なコンポーネントです1。チトクロームP450 2D6(CYP2D6)は、最も重要な薬物代謝酵素の1つをコードしており、臨床使用薬剤の約21%の代謝に関与しています2。CYP2D6遺伝子は多型性が高く、Pharmacogene Variation(PharmVar)Consortium3によって100を超えるスターアリルが定義されています。スターアリル4は、小規模バリアント(SNVとIndel)と構造多型(SV)の組み合わせによって定義される遺伝子ハプロタイプであり、異なるレベルの酵素活性、すなわち、不良、中間、正常、超高速の代謝因子に対応します5–7。
CYP2D6のジェノタイピングは、CYP2D6の一般的な欠失と重複、およびCYP2D6とその偽遺伝子パラログであるCYP2D7の間のハイブリッド4,8,9によって課題に直面しています。これは、ほぼ同一に近い領域を含む94%のシーケンス類似性を共有しています8,10。従来、CYP2D6ジェノタイピングは、PharmacoScanなどのアレイベースのプラットフォーム、またはTaqManアッセイ、ddPCR、ロングレンジPCRなどのポリメラーゼ連鎖反応(PCR)ベースの方法で、低~中スループットで行われます。これらのアッセイは、精査するスターアリル(バリアント)の数が異なるため、アッセイ間でジェノタイピング結果にばらつきが生じます8,11,12。SVを検出するには、これらのアッセイまたはテストプラットフォームを、CNV(コピー数バリエーション)アッセイで補完する必要が生じることがあり、このアッセイも既知のCNVのサブセットのみの検出に限定される可能性があります4,9。
近年の次世代シーケンサーの進歩により、ハイスループットかつ臨床的に意義のある時間枠でゲノム全体のプロファイリングが可能になりました。これらの進歩により、多くの国が大規模な集団シーケンスプロジェクトを実施しています13–15。ファーマコゲノミクス(PGx)検査は、これらの取り組みの臨床的有用性を大幅に向上させます。これらの研究でPGxを可能にするため、当社はCYP2D6コーラー、Cyrius16を開発しました。CYP2D6とCYP2D7の相同性の課題を克服する新しいWGS固有のCYP2D6ジェノタイピングツールです。ここでは、Cyriusの概要とWGSによって実現されるPGxのアプリケーションの今後の方向性について説明します。
Cyriusが使用するCYP2D6ジェノタイピング法
CYP2D6ではCYP2D7との相同性のためにリードアライメントの精度が低下し(図1)、バリアントコーリングが困難になり、エラーが発生しやすくなります。Cyriusは、この課題を克服するために新しいアプローチを使用しています。図2ではCoriellサンプルNA12878(*3/*68+*4)を例として使用して、詳細なワークフローを示します。まず、Cyriusは、CYP2D6およびCYP2D7の総コピー数を特定します。このプロセスでは、CYP2D6またはCYP2D7にマッピングされたすべてのリードを使用して、WGSにアラインされたBAMファイルのリード深度を計算します。次にCyriusは完全型のCYP2D6遺伝子の数を同定し、集団の中でCYP2D6とCYP2D7の間で異なっていることが証明されている117の塩基に基づいてハイブリッド遺伝子を同定します。ハイブリッドは、これらの塩基で測定されたCYP2D6のコピー数(CN)が遺伝子内で変化したときに同定されます。例えば、図2Bに示すサンプルNA12878には、CYP2D6の完全なコピーが2つと、エクソン1がCYP2D6から、エクソン2~9がCYP2D7からのハイブリッド(すなわち*68)が1つあります。次に、Cyriusは、リードアライメントを解析して、スターアリルを定義するタンパク質を変化させる小規模バリアントを同定します(図2C)。最後に、CyriusはSVと小規模バリアントをスターアリルの定義と照合し、コールされたバリアントと一致する遺伝型を生成します。
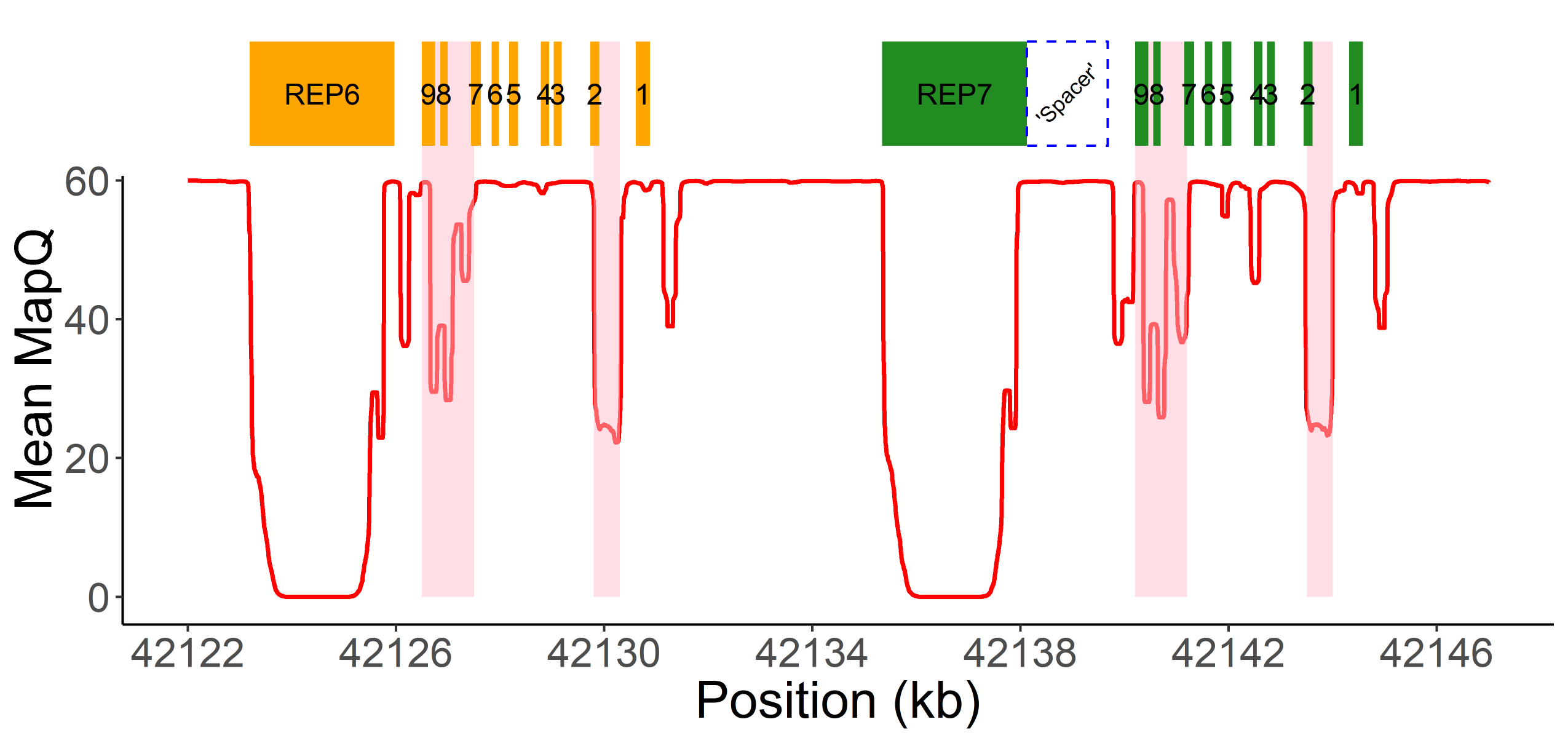
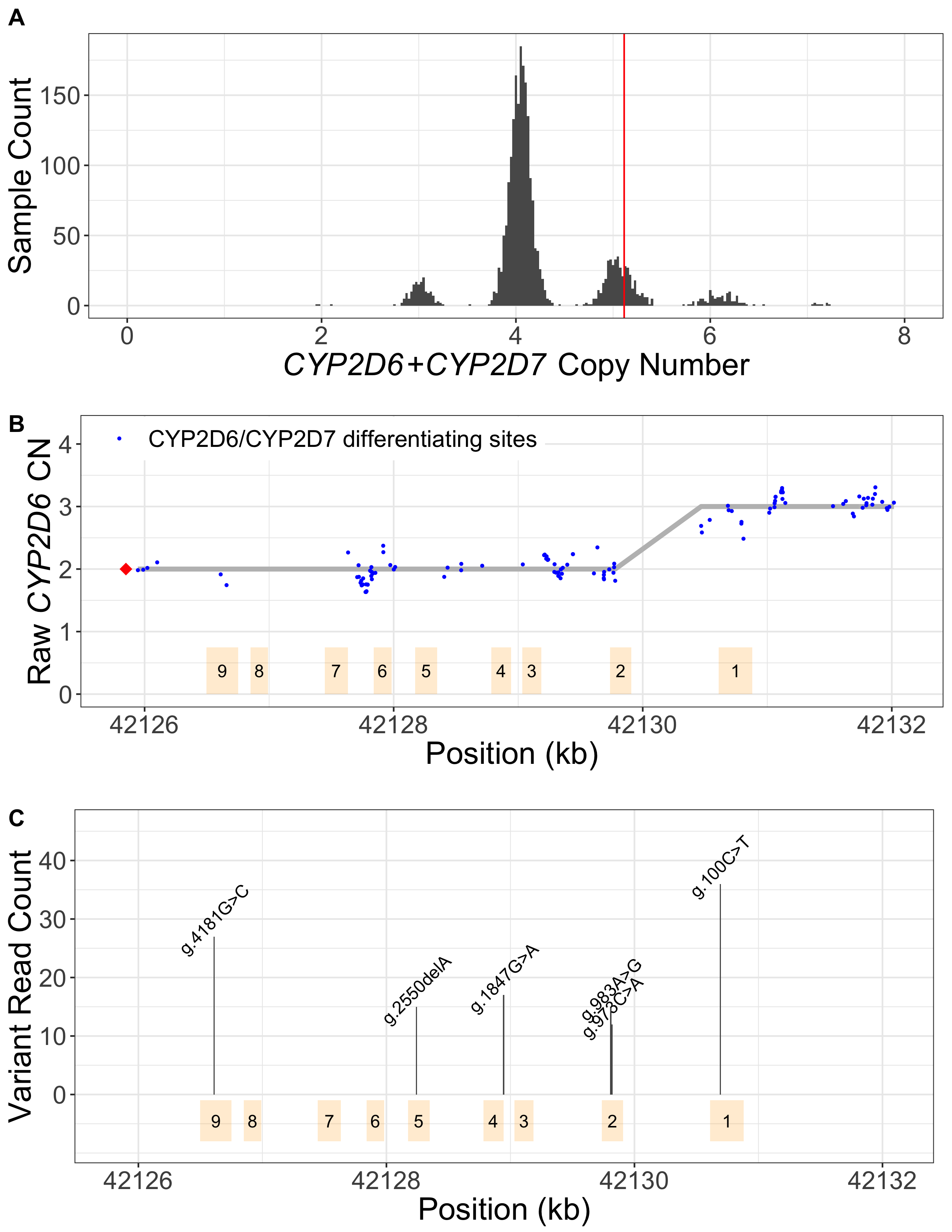
A. CN(CYP2D6+CYP2D7)は、CYP2D6またはCYP2D7のいずれかにアラインするすべてのリードをカウントし、モデル化することで得られます。ヒストグラムは、1kGPサンプルにおけるノーマライズされたCYP2D6+CYP2D7深度の分布を示し、CN2、3、4、5、6および7のピークを示しています。赤色の縦線はNA12878の値を表し、CN5に対応しており、追加のコピー(CYP2D6またはハイブリッドの可能性も)を示します。B. SVは、CYP2D6/CYP2D7を区別する塩基のCNを調べることでコールされます。エクソンは黄色のボックスで示されます。青色の点は、未処理のCYP2D6のコピー数(CN)を示し、これはCN(CYP2D6+CYP2D7)にCYP2D6をサポートするリード数がCYP2D6とCYP2D7をサポートするリード数の合計に占める割合を掛けて算出します。赤色のひし形は、3’末端でCYP2D6由来の遺伝子(完全なCYP2D6またはCYP2D7-CYP2D6ハイブリッド)のCNを示し、CN(CYP2D6+CYP2D7)からCN(スペーサー)を引いて算出されます。CYP2D6のCNは、CYP2D6/CYP2D7の分化部位ごとにコールされます。また、遺伝子内でCYP2D6のCNが変化する場合は、ハイブリッドの存在が示唆されます。NA12878では、CYP2D6 CNはエクソン2とエクソン1の間で2から3に変化し、CYP2D6-CYP2D7ハイブリッド(*68)を示します。C. タンパク質を変化させる小規模バリアントを定義するスターアリルのリード数のサポートは、各バリアントのコピー数をコールするために使用されます。y軸は、照会されたすべての小規模バリアント位置のリード数を示します。NA12878では6つのバリアントがコールされ、そのうちの1つはg.100C>Tで2つのコピーとしてコールされています(スターアリル定義により1つのコピーは*4に属し、もう1つのコピーは*68に属します)。最後に、検出されたSVと小規模バリアントに基づいてスターアリルがコールされます。
性能検証
CDC Genetic Testing Reference Material Program(GeT-RM)11,17による標準サンプルパネルを利用でき、そこでは、主要なファーマコゲネスのコンセンサス遺伝型が複数のジェノタイピングプラットフォームを使用して導出されているので、新たに開発された手法のジェノタイピング精度の評価が可能になりました。Cyriusが行ったCYP2D6コールを評価するため、144の真理値セットサンプルと比較しました。これには、138のGeT-RMサンプル、およびPacBio HiFiシーケンスリードを使用して真理値を取得した8つのサンプル(2つのサンプルがGeT-RMとPacBio間で重複)が含まれます。Cyriusは当初、144の正解サンプル中5件の不一致コールを示し、一致率は96.5%でした。その後、原因を同定し、Cyriusがこれらの5つのサンプルのうち4つを正しくコールすることができ、99.3%(144サンプル中143サンプル)の「トレーニング済み」一致率を達成しました。Cyriusの精度は、既存の2つのCYP2D6コーラーであるAldy18(86.8%)およびStargazer19(84.0%)よりも高くなっています16。
合わせて、この研究で使用した検証サンプルにより、40のスターアリルを含む47種の異なるハプロタイプ、および*13+*2、*68+*4、*36+*10、*36+*36+*10を含む重複やタンデム配列などの複数のSV構造におけるCYP2D6コールの精度を確認しました。これらの40のスターアリルは、PharmVarの131スターアリルの30.5%(2020年7月時点)、既知の機能を持つスターアリルの51.7%(60のうち31)を占めています。5つの異なる集団(ヨーロッパ人、アフリカ人、東アジア人、南アジア人、混合アメリカ人)を表す1000 Genomes Project(1kGP)20からのサンプルにCyriusを適用することで、これらの検証済みの40のスターアリルが、パンゲノム集団に存在するスターアリルの96%を占めることが示されています。
次に、597の1kGPトリオからのシーケンスデータにおけるCyriusコールのメンデルの法則との一貫性を評価しました。上記の正解の遺伝型との比較では異なるハプロタイプフェージングが可能ですが、メンデルの法則との一貫性チェックは、CYP2D6のコピーが3つ以上存在する場合のスターアリルのフェージングをより厳密にチェックするものです。家族3人全員をコールした572のトリオのうち、561(98.1%)がメンデルの法則に一致しました。法則に一致しないすべてのトリオは、フェージングを変更することで解決できます。つまり、発端者のコールされたスターアリルが、両親のどちらにも存在しないというケースはありませんでした。不一致ケースの大半(8/11件)は、トリオ解析により2つの同一コピーのCYP2D6が同じハプロタイプ上に存在し、もう一方のハプロタイプにはCYP2D6のコピーがないと特定されたケースでした。具体的には、Cyriusのコール(*1/*1)と、トリオベースのフェージング結果(*5/*1×2)の間にずれが生じていました。このメンデルの法則との一貫性チェックは、家系全体での遺伝型の一貫性を確認しますが、コールされたスターアリルの精度は確認しません。トリオ一致テストと、正解の遺伝型に対して上記で実施された精度テストを組み合わせることで、Cyriusから得られる遺伝型全体の精度に信頼が持てます。
5つの民族集団にわたる実用的な薬理遺伝学バリアント
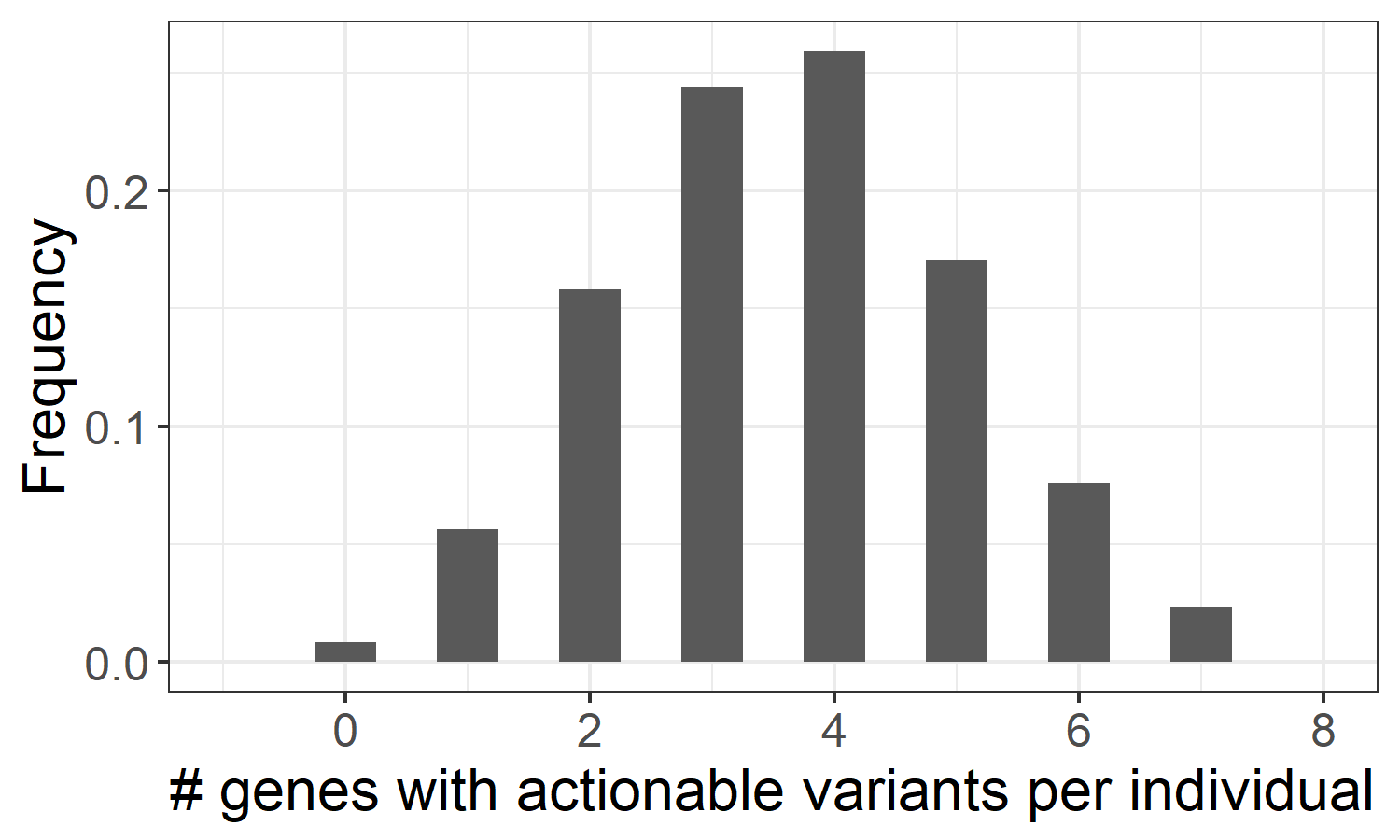
Cyriusで採用されている方法は、別のファーマコジーンCYP2B6を含む、同じ相同性の問題を抱えるその他のパラログを解決するために適用できます。より完全なファーマコゲノミクスの全体像を提供するために、CPIC21レベルAガイドライン(https://cpicpgx.org/genes-drugs/)に沿って遺伝型19のファーマコジーンへの追加パイプラインを開発しました。このガイドラインでは、遺伝情報を使用して影響を受けた薬剤の処方を変更する必要があります。これらの19の遺伝子には、CACNA1S、CFTR、CYP2B6、CYP2C19、CYP2C9、CYP2D6、CYP3A5、CYP4F2、DPYD、G6PD、IFNL3、NUDT15、RYR1、SLCO1B1、TPMT、UGT1A1、VKORC1、HLA-A、HLA-Bが含まれます。このうち当社は、CYP2D6/CYP2B6(Cyrius)およびHLA-A/HLA-B(DRAGEN HLAコーラー)のジェノタイピングに専門コーラーを使用しています。小規模バリアントやCNVをスターアリルや活性スコアに変換することで、1kGPサンプルにおけるさまざまな代謝能ステータスの分布を評価し、修正された薬剤処方を必要とする遺伝型を持つ遺伝子を同定できます。この解析に基づき、当社はパンゲノム集団の99.2%の個人が、対処可能なバリアントを持つファーマコジーンを少なくとも1つ持っていると推定しており(図3)、WGSは、ほぼすべての健康な個人に貴重なファーマコジェネティクス情報を提供できることを示しています。
まとめ
非常に複雑なCYP2D6領域を精確にジェノタイピングする新しいソフトウェアツール、Cyriusを紹介しました。144の検証サンプル全体で、パンゲノム集団のスターアリルの約96%を占める40の異なるスターアリル全体でCyriusの精度を確認することができます。Cyriusの詳細についてご関心があれば、この手法を説明する当社の原稿を確認することをお勧めします16。従来のジェノタイピングアッセイと比較すると、WGSはCNVを含むすべてのバリアントを検出し、適切なソフトウェアと組み合わせることで、既知のスターアリルをすべて精確に解析できるため、集団全体でより精確なアリル頻度データベースを構築するための有望な選択肢となります。WGSベースのPGxツールや、Cyriusのようなよりターゲットを絞った手法の開発を継続していくことで、当社はファーマコゲノミクスを加速し、個別化医療に一歩近づくことに貢献できます。
学術用途向けの詳細情報またはDRAGEN試用版ライセンスについては、dragen-info@illumina.comまでお問い合わせください。
謝辞
CDC Genetic Testing Reference Material Program(GeT-RM)によるコンセンサス遺伝型の生成に感謝いたします。1kGP WGSデータの生成とリリースについて、New York Genome CenterとCoriell Institute for Medical Researchに感謝いたします。共同著者のFei Shen氏、Nina Gonzaludo氏、Alka Malhotra氏、Cande Rogert氏、Ryan Taft氏、David Bentley氏に感謝いたします。DRAGEN HLAコーラーの提供についてBochao Zhang氏に感謝いたします。
外部リンク
論文:https://www.nature.com/articles/s41397-020-00205-5
ソフトウェア:https://github.com/Illumina/Cyrius
関連コンテンツ
注釈
- Evans WE, Relling MV. Moving towards individualized medicine with pharmacogenomics. Nature.2004;429(6990):464-468. doi:10.1038/nature02626
- Zhou S-F. Polymorphism of human cytochrome P450 2D6 and its clinical significance: Part I. Clin Pharmacokinet.2009;48(11):689-723. doi:10.2165/11318030-000000000-00000
- Gaedigk A, Ingelman-Sundberg M, Miller NA, et al. The Pharmacogene Variation (PharmVar) Consortium: Incorporation of the Human Cytochrome P450 (CYP) Allele Nomenclature Database. Clin Pharmacol Ther.2018;103(3):399-401. doi:10.1002/cpt.910
- Nofziger C, Turner AJ, Sangkuhl K, et al. PharmVar GeneFocus: CYP2D6. Clin Pharmacol Ther.2020;107(1):154-170. doi:10.1002/cpt.1643
- Gaedigk A, Simon SD, Pearce RE, Bradford LD, Kennedy MJ, Leeder JS. The CYP2D6 activity score: translating genotype information into a qualitative measure of phenotype. Clin Pharmacol Ther.2008;83(2):234-242. doi:10.1038/sj.clpt.6100406
- Gaedigk A, Sangkuhl K, Whirl-Carrillo M, Klein T, Leeder JS. Prediction of CYP2D6 phenotype from genotype across world populations. Genet Med.2017;19(1):69-76. doi:10.1038/gim.2016.80
- Caudle KE, Sangkuhl K, Whirl-Carrillo M, et al. Standardizing CYP2D6 Genotype to Phenotype Translation: Consensus Recommendations from the Clinical Pharmacogenetics Implementation Consortium and Dutch Pharmacogenetics Working Group. Clin Transl Sci.2020;13(1):116-124. doi:10.1111/cts.12692
- Nofziger C, Paulmichl M. Accurately genotyping CYP2D6: not for the faint of heart. Pharmacogenomics.2018;19(13):999-1002. doi:10.2217/pgs-2018-0105
- Yang Y, Botton MR, Scott ER, Scott SA. Sequencing the CYP2D6 gene: from variant allele discovery to clinical pharmacogenetic testing. Pharmacogenomics.2017;18(7):673-685. doi:10.2217/pgs-2017-0033
- Gaedigk A. Complexities of CYP2D6 gene analysis and interpretation. Int Rev Psychiatry Abingdon Engl.2013;25(5):534-553. doi:10.3109/09540261.2013.825581
- Pratt VM, Everts RE, Aggarwal P, et al. Characterization of 137 Genomic DNA Reference Materials for 28 Pharmacogenetic Genes: A GeT-RM Collaborative Project. J Mol Diagn JMD.2016;18(1):109-123. doi:10.1016/j.jmoldx.2015.08.005
- Bousman CA, Jaksa P, Pantelis C. Systematic evaluation of commercial pharmacogenetic testing in psychiatry: a focus on CYP2D6 and CYP2C19 allele coverage and results reporting. Pharmacogenet Genomics.2017;27(11):387-393. doi:10.1097/FPC.0000000000000303
- Ashley EA. The Precision Medicine Initiative: A New National Effort. JAMA.2015;313(21):2119-2120. doi:10.1001/jama.2015.3595
- The Genome of the Netherlands Consortium, Francioli LC, Menelaou A, et al. Whole-genome sequence variation, population structure and demographic history of the Dutch population. Nat Genet.2014;46(8):818-825. doi:10.1038/ng.3021
- Turnbull C, Scott RH, Thomas E, et al. The 100 000 Genomes Project: bringing whole genome sequencing to the NHS. BMJ. 2018;361:k1687. doi:10.1136/bmj.k1687
- Chen X, Shen F, Gonzaludo N, et al. Cyrius: accurate CYP2D6 genotyping using whole-genome sequencing data. Pharmacogenomics J.Published online January 18, 2021:1-11. doi:10.1038/s41397-020-00205-5
- Gaedigk A, Turner A, Everts RE, et al. Characterization of Reference Materials for Genetic Testing of CYP2D6 Alleles: A GeT-RM Collaborative Project. J Mol Diagn JMD.Published online August 9, 2019. doi:10.1016/j.jmoldx.2019.06.007
- Numanagić I, Malikić S, Ford M, et al. Allelic decomposition and exact genotyping of highly polymorphic and structurally variant genes. Nat Commun.2018;9(1):1-11. doi:10.1038/s41467-018-03273-1
- Lee S, Wheeler MM, Patterson K, et al. Stargazer: a software tool for calling star alleles from next-generation sequencing data using CYP2D6 as a model. Genet Med.2019;21(2):361. doi:10.1038/s41436-018-0054-0
- The 1000 Genomes Project Consortium. A global reference for human genetic variation. Nature.2015;526(7571):68-74. doi:10.1038/nature15393
- Relling MV, Klein TE. CPIC: Clinical Pharmacogenetics Implementation Consortium of the Pharmacogenomics Research Network. Clin Pharmacol Ther.2011;89(3):464-467. doi:10.1038/clpt.2010.279