エクソームシーケンスは、希少な遺伝性疾患を持つ患者や家族の臨床診断を変革し、ファーストライン検査として採用された場合、診断の時間とコストを大幅に削減しました。しかし、エクソームシーケンスの診断率は、希少遺伝性疾患コホート1では約25~30%であり、多くの患者は診断を受けません。機能的なノンコーディングバリアントを同定できる方法があれば、全ゲノムシーケンスにより診断率が大幅に上昇する可能性があります。
クリプティックスプライスバリアントは、必須のGTおよびAGスプライスジヌクレオチドの外部に横たわっているにもかかわらず、mRNAスプライシングの正常なパターンを破壊するバリアントであり、希少な遺伝性疾患において重要な役割を果たすことが長い間認識されてきたノンコーディングバリアントのクラスです。しかし、これらの変異は、スプライシングコードの理解が不完全であり、その結果、スプライス変異バリアントを正確に特定することが困難であるため、臨床現場では見落とされがちです。
近年、RNA-seqはこのようなバリアントを検出するための有望なアッセイとして登場しました2。しかし、これまで、臨床現場でのRNA-seqの有用性は、関連する細胞型が生検に利用しやすい少数の症例に限定されています。任意のプレmRNAシーケンスからのスプライシングの一般的な予測は、全ゲノムシーケンスを介して特定されたノンコーディングバリアントのスプライスの変化による結果の正確な予測を可能にする可能性があり、遺伝性疾患患者の診断を大幅に改善する可能性があります。
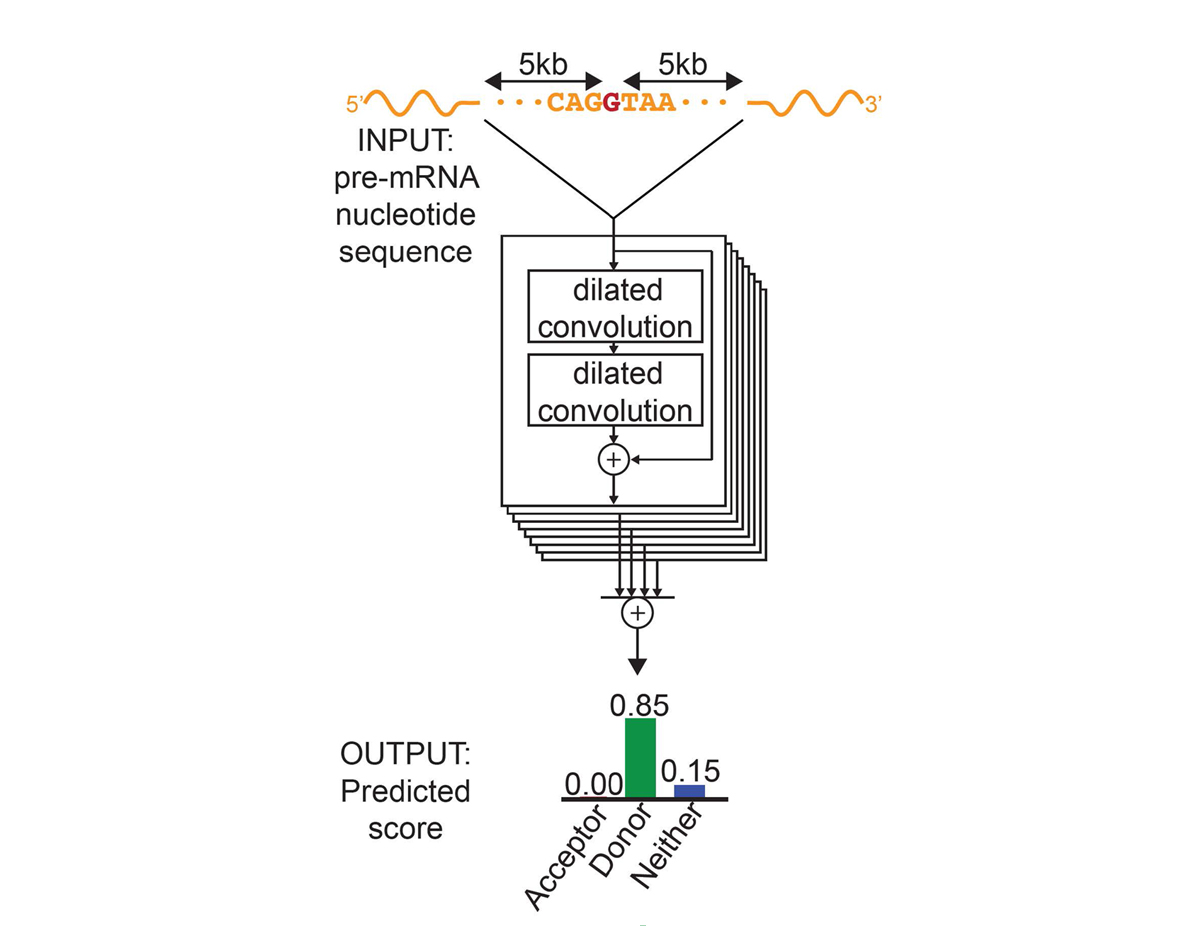
プレmRNA転写産物の各位置について、SpliceAI-10k は、10,000ヌクレオチドの隣接配列を入力として使用し、その位置がスプライスアクセプター、スプライスドナー、またはどちらでもないかどうかを予測します。
SpliceAIを構築しました。これは、プレmRNA転写産物の各位置が、プレmRNA転写産物のゲノムシーケンスに基づいてスプライスドナーか、またはスプライスアクセプターかを予測するディープ残留ニューラルネットワークです(図1)。スプライスドナーとスプライスアクセプターは数千のヌクレオチドで分離される可能性があるため、非常に大きなゲノム距離にわたるシーケンス決定因子を認識できるニューラルネットワークアーキテクチャを採用しました。エクソン-イントロン境界3に隣接する短いヌクレオチドウィンドウのみを考慮した、またはヒトが設計した特徴4に依拠した以前の方法とは対照的に、当社のニューラルネットワークは、mRNA前駆体の各位置のスプライス機能を予測するために、隣接コンテキストシーケンスの10,000ヌクレオチドを評価することで、一次シーケンスから直接スプライシング決定因子を学習します。ヒト染色体のサブセット上のGENCODEアノテーションされたmRNA前転写配列を使用して、ニューラルネットワークのパラメーターをトレーニングし、残りの染色体上の転写産物をパラログを除外してネットワークの予測をテストしました。
テストデータセット内のプレmRNA転写産物の場合、ネットワークは、95%のtop-k精度でスプライスジャンクションを予測します。これは、予測されたスプライス部位の数がテストデータセットに存在する実際のスプライス部位の数と等しい閾値における正しく予測されたスプライス部位の割合です(図2)。CFTRなどの100 kbを超える遺伝子でも、ヌクレオチド精度に完全に再構成されることがよくあります(図3)。
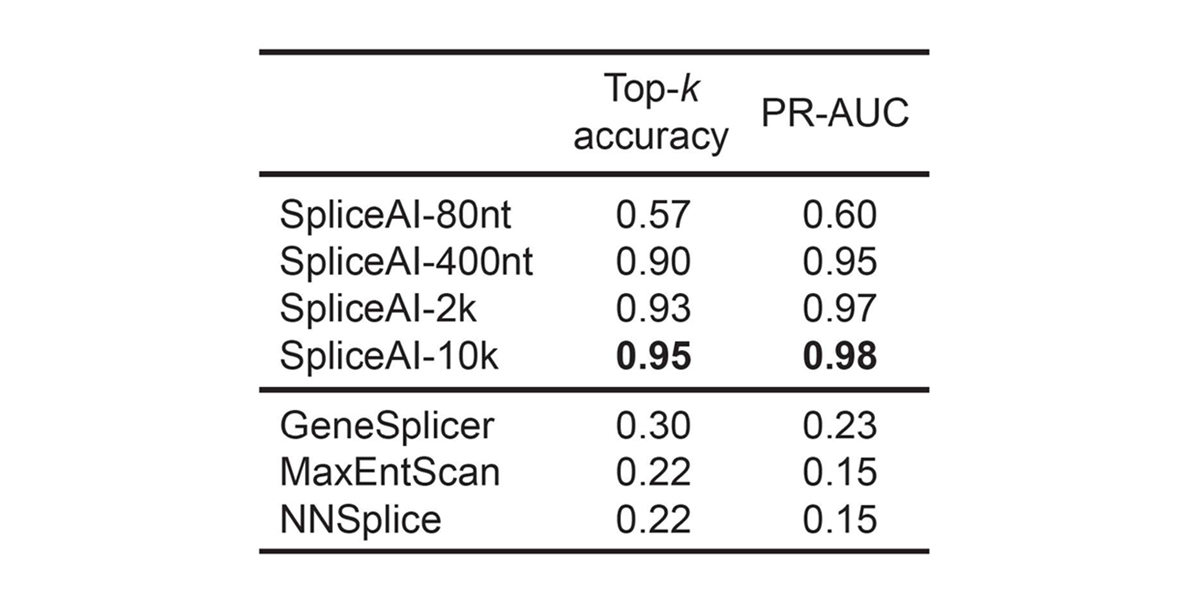
テストデータセットのmRNA前駆体転写産物の場合、ネットワークは95%のtop-k精度でスプライスジャンクションを予測します。これは、予測されたスプライス部位の数がテストデータセットに存在する実際のスプライス部位の数と等しい閾値における正しく予測されたスプライス部位の割合です。
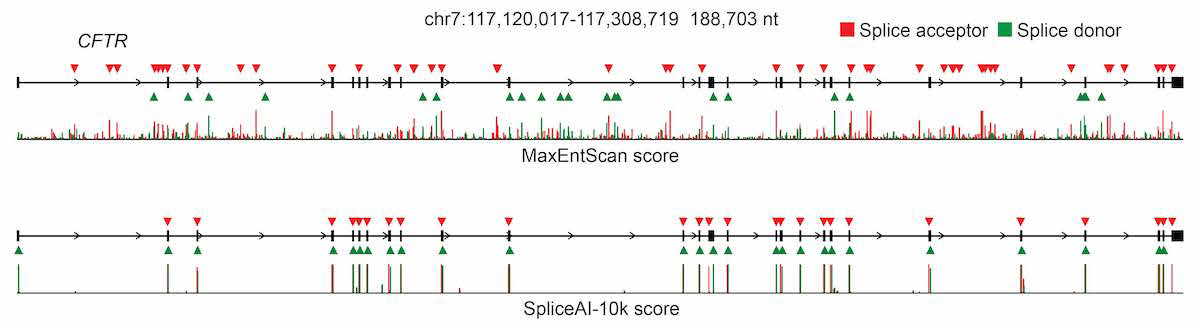
MaxEntScan(上)およびSpliceAI-10k(下)を使用してスコアリングしたCFTR遺伝子の完全なmRNA前転写産物が、予測されるアクセプター(赤色の矢印)およびドナー(緑色の矢印)の部位とエクソンの実際の位置(黒色のボックス)とともに表示されます。各手法について、予測サイト数を実際のサイト数の合計に等しくする閾値を適用しました。
ネットワークが単にエクソンシーケンスバイアスに依存していないことを確認するために、長いノンコーディングRNAについてもネットワークをテストしました。ノンコーディング転写産物のアノテーションが不完全であるにもかかわらず、ネットワークは、84%のtop-k精度でlincRNAの既知のスプライスジャンクションを予測し、タンパク質コーディング選択圧のない任意のシーケンス上のスプライセオソームの挙動に近似できることを示唆しています。
このネットワークは、リファレンス転写シーケンスとスプライスジャンクションアノテーションについてのみトレーニングされ、 バリアントデータはトレーニング中に使用されず スプライシングのシーケンス決定因子を正確にモデル化するネットワークの能力の、バリアント効果の予測を困難なテストにしています。 我々は、リファレンスプレmRNA転写産物シーケンスとバリアントを含む代替転写産物シーケンスの両方についてエクソン-イントロン境界を予測することで、スプライス改変機能のための遺伝子バリアントの評価にディープラーニングネットワークを拡張しました。 スコアの差を測ります(∆スコア、 図4)。
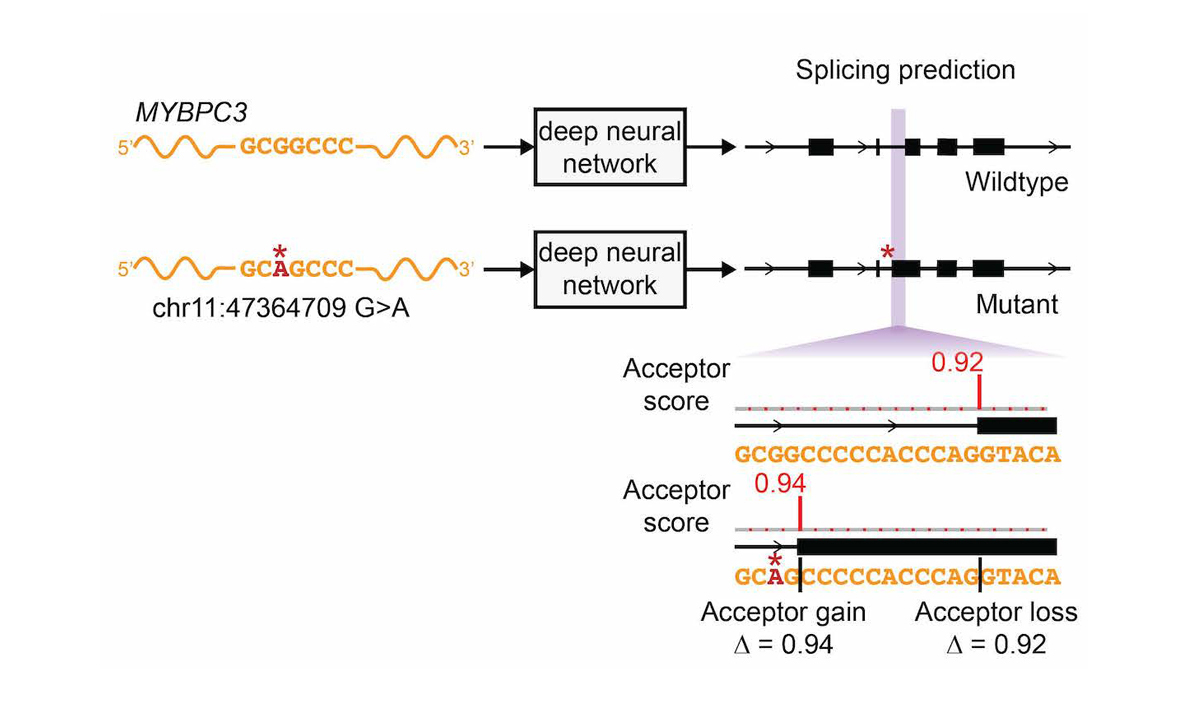
変異のスプライスの変化の影響を評価するため、SpliceAI-10kは、変異の有無にかかわらず、遺伝子のプレmRNAシーケンスの各位置におけるアクセプターとドナーのスコアを予測します。rs397515893は、心筋症に関連するMYBPC3イントロンの病原性クリプティックスプライスバリアントです。変異の∆スコア値は、バリアントから50 nt以内のスプライス予測スコアの最大の変化です。
GTExコホート5のRNA-seqデータにおける暗号スプライスバリアントの効果を調べました。このデータには、全ゲノムシーケンスと複数の組織からのRNA-seqの両方を持つ149人の個人が含まれています。自信を持って予測されたクリプティックスプライスバリアント(∆ Score > 0.5)は、必須のGTまたはAGスプライス破壊率の4分の3でRNA-seqを検証します(SpliceAI論文の図2D)。クリプティックスプライスバリアントの検定率と効果サイズはどちらも、その∆スコアを綿密に追跡し、モデルの予測スコアがバリアントのスプライス変更の可能性の優れた代用であることを示しました。検証済みのバリアント、特にスコアが低いバリアント(∆スコア< 0.5), are often incompletely penetrant, and result in alternative splicing with production of a mixture of both aberrant and normal transcripts in the RNA-seq data. For cryptic splice variants that produce aberrant splice isoforms in at least three-tenths of the observed copies of the mRNA transcript, the network has a sensitivity of 71% when the variant is near exons, and 41% when the variant is in deep intronic sequence (∆ Score >0.5、SpliceAI論文の図2F)。これらの結果は、ディープイントロンバリアントは予測がより困難であることを示しており、これはおそらくディープイントロン領域にはエクソンの近くに存在するように選択された特異性決定因子がより少ないためであると考えられます。
予測されるクリプティックスプライスバリアントの自然選択の特徴を探索するために、Exome Aggregation Consortium(ExAC)データベース6のヒトエクソーム60,706個 、Genome Aggregation Database(gnomAD)コホート7のヒト15,496個に存在する各バリアントをスコアリングし、エクソン-イントロン境界を変えることが予測されるバリアントを特定しました。予測されたスプライス改変バリアントに作用するネガティブな選択の程度を測定するために、共通のアリル頻度で見つかった予測されたスプライス改変バリアントの数をカウントし(>コホートでは0.1%)、シングルトンアリル頻度で予測されたスプライス改変バリアントの数と比較しました。ヒト集団の規模が最近急激に拡大したため、シングルトンバリアントは、最近作成された変異を表し、選択を精製することで最小限のフィルターでろ過されています。対照的に、一般的なバリアントは、精製選択のふるいを通過した中性変異のサブセットを表しています。 高スコアのクリプティックスプライスバリアントは、一般的なバリアントで大幅に枯渇しており、これらのバリアントが強力なネガティブ選択下にあることを示しています(SpliceAI論文の図4Bおよび4D)。これは、自信を持って予測されたクリプティックスプライス変異の大部分が機能的であることを示しています。
自閉症スペクトラム障害と重度の知的障害を持つ患者の大規模なシーケンス研究では、神経発達パスウェイの遺伝子を破壊するde novoタンパク質コーディング変異(ミスセンス、ナンセンス、フレームシフト、および必須スプライスジヌクレオチド)の中心的な役割が示されています。スプライシングの変化によって作用するノンコーディング変異の臨床的影響を評価するために、Simons Simplex CollectionおよびAutism Sequencing Consortium 9-11のDeciphering Developmental Disordersコホート(DDD)8の知的障害を持つ4,293人、自閉症スペクトラム障害(ASD)を持つ3,953人、Simons Simplex Collectionの影響を受けていない兄弟姉妹対照2,073人を対象に、ニューラルネットワークを適用してde novo変異の影響を予測しました。スプライシングの中断が予測されるde novo変異は、健常対照と比較して、知的障害および自閉症スペクトラム障害のコホートで有意に濃縮されていることを示しています(SpliceAI論文の図5A)。罹患者と非罹患者のde novo変異の過剰量に基づくと、各研究でシーケンスカバレッジまたはバリアント確認が欠如している領域で予想される変異の割合を調整した後、クリプティックスプライス変異は自閉症スペクトラム障害の病原性変異の約11%、知的障害の9%を占めると推定されます。
次に、クリプティックスプライスサイト予測の精度を実験的に検証しました。そのため、Simons Simplex Collectionから36名の個人から末梢血由来のリンパ芽球様細胞株(LCL)を入手しました。LCLは、LCLの発現レベルが少なくとも最小限である遺伝子におけるde novoのクリプティックスプライス変異を予測していました。各個人が近親者の中で自閉症の唯一の症例でした。ほとんどの希少な遺伝性疾患の場合と同様に、関連性のある組織や細胞の種類(脳の発達が疑われる)にはアクセスできませんでした。そのため、LCLにおけるこれらの転写産物の多くの発現の弱さを補うために、詳細なmRNAシーケンスを実施しました。関心遺伝子のRNA-seqカバレッジが不十分な8名を除外した後、28名中21名で、予測されたde novoのクリプティックスプライス変異に関連する固有の異常なスプライシングイベントを特定しました。これらの異常なスプライシング事象は、深いLCL RNA-seqが得られた他の35名、およびGTExコホートの149名には見られませんでした。転写産物が十分に発現しているにもかかわらず、7例はLCLで異常なスプライシングを示しませんでした。これらのサブセットは偽陽性の予測を示している可能性がありますが、一部のクリプティックスプライス変異は、これらの実験条件下でLCLでは観察できない組織特異的な代替スプライシングをもたらす可能性があります。
ディープラーニングは生物学における比較的新しい手法であり、潜在的なトレードオフがないわけではありません。ディープラーニングモデルでは、ヒトの専門家が十分に説明していないシーケンス決定要因を利用することができますが、スプライセオソームの真の挙動を反映していない特徴がモデルに組み込まれているリスクもあります。これらの交絡特性は、アノテーションされたエクソン-イントロン境界を予測する見かけ上の精度を高める可能性がありますが、遺伝的変異によって誘発される任意の配列変化のスプライス変化効果を予測する精度を低下させる可能性があります。バリアントの正確な予測は、モデルが真の生物学に一般化できるという最も強力なエビデンスを提供するため、3つの完全に直交する手法を使用して、予測されたスプライス改変バリアントの裏付けとなるエビデンスを提供します。RNA-seq、ヒト集団における自然選択、症例コホートと対照コホートにおけるde novoバリアントの濃縮。これは、無関係な特徴のモデルへの組み込みを完全に排除するものではありませんが、結果として得られるモデルは、遺伝性疾患患者におけるクリプティックスプライス変異の同定などの実用的なアプリケーションに重要な価値があるスプライシングの真の生物学に十分に忠実であるように見えます。
ノンコーディングゲノムの変異がヒトの疾患にどのようにつながるかについての理解は、まだまだ十分ではありません。小児神経発達障害における浸透性デノボクリプティックスプライス変異の可能性の発見は、全ゲノムシーケンスとノンコーディングゲノムの解釈の改善が、重度の遺伝性疾患を持つ患者に利益をもたらす可能性があることを実証しています。クリプティックスプライス変異はがんでも重要な役割を果たしていることが示されており、スプライス因子の反復性体細胞変異はスプライシング特異性に広範な変化をもたらすことが示されています12。特にスプライソソーム内のタンパク質に直接影響を与える変異の場合、異なる組織や細胞環境におけるスプライシングの制御を理解するために多くの作業が未だに行われています。配列特異的な方法でスプライシングの欠陥をターゲットにする可能性があるオリゴヌクレオチド療法の最近の進歩13を踏まえると、この顕著なプロセスを制御する制御メカニズムをより深く理解することで、治療介入のための新しい候補への道を開くことができます。
確認
J. K. Pritchardの洞察に富んだ議論とサポート、Genome Aggregation Database(gnomAD)、およびエクソームとゲノムバリアントデータをこのリソースに提供したグループに感謝いたします。Stephan J. Sandersは、Simons Foundationからの助成金(SFARI #402281および#574598)によって支援されました。
参考文献
- Lee H, Deignan JL, Dorrani N, et al. 希少なメンデル性疾患の遺伝子同定のための臨床エクソームシーケンス。 ジャマ。2014;312(18):1880-1887。
- Cummings BB, Marshall JL, Tukiainen T, et al. トランスクリプトームシーケンスによるメンデル性疾患の遺伝子診断の改善。 Sci Transl Med. 2017;9(386):eaal5209.
- Yeo G, Burge CB. RNAスプライシングシグナルへのアプリケーションによるショートシーケンスモチーフの最大エントロピーモデリング。 J Comput Biol. 2004;11(2-3):377-394。
- Xiong HY, Alipanahi B, Lee LJ, et al. RNAスプライシング。ヒトのスプライシングコードは、疾患の遺伝的決定要因に関する新しい洞察を明らかにします。 科学。2015;347(6218):1254806。
- The GTEx Consortium. 遺伝子型-組織発現(GTEx)パイロット解析:ヒトにおける多組織遺伝子制御。 Science 2015;348:648–660。
- Lek M, Karczewski KJ, Minikel EV, et al. 60,706人のヒトにおけるタンパク質コーディング遺伝子変異の解析。 自然。2016;536(7616):285-291。
- Karczewski KJ, Francioli LC, Tiao G, et al. 141,456人のヒトにおける変動から定量化された変異制約スペクトル。 自然。2020;581(7809):434-443。
- Deciphering Developmental Disorders Study. 発達障害におけるde novo変異の有病率と構造。 自然。2017;542(7642):433-438。
- De Rubeis S, He X, Goldberg AP, et al. 自閉症で破壊されたシナプス遺伝子、転写遺伝子、クロマチン遺伝子。 自然。2014;515(7526):209-215。
- Sanders SJ, He X, Willsey AJ, et al. 71のリスク遺伝子座から、自閉症スペクトラム障害のゲノムアーキテクチャと生物学に関する洞察が得られます。 ニューロン。2015;87(6):1215-1233。
- Turner TN, Hormozdiari F, Duyzend MH, et al. 自閉症に罹患した家族のゲノムシーケンスにより、推定ノンコーディング規制DNAの破壊が明らかになりました。 Am J Hum Genet. 2016;98(1):58-74。
- Jung H, Lee D, Lee J, et al. イントロンの保持は、腫瘍抑制因子の不活性化の広範なメカニズムです。 Nat Genet. 2015;47(11):1242-1248。
- Finkel RS, Mercuri E, Darras BT, et al. 乳児期発症型脊髄性筋萎縮症におけるヌシネルセンと偽対照の比較。 N Engl J Med. 2017;377(18):1723-1732。