はじめに
遺伝性疾患は、特に新生児集中治療室(NICU)に入院した乳児において、小児死亡の主な原因となっています。1,2 新生児死亡の約40%は希少な遺伝性疾患と関連しています。1-5 遺伝性疾患を持つ小児患者の非常に急速な疾患進行のため、原因となる遺伝的変異を迅速に特定することが非常に重要です。迅速な診断により、治療的介入をタイムリーに適用することができ、この脆弱な患者集団における重度の罹患率と死亡率を防ぐことができます。6 標準的なゲノムシーケンス技術を使用した診断までの時間は、最大数週間かかる場合があり、医療従事者による解釈と管理の決定が必要です。これらの診断の遅れや治療の開始は、希少な遺伝性疾患を持つ一部の急性疾患患者の転帰不良につながる可能性があります。
ラピッド全ゲノムシーケンス(rWGS)は、標準的なゲノムシーケンスに代わるものです。 50時間未満で重症乳児の希少な遺伝性疾患の迅速かつ正確な診断を提供できる可能性があります。7 臨床試験では、 rWGSは、最大57%の症例で陽性診断率を示しており、ICUに入院した小児患者の症例の30%~72%で、8名および十分な情報を得た上での医療管理。7-13 しかし rWGSの臨床的有用性は、スケーラビリティの欠如と、ICUにおける小児の遺伝子診断の20%に寄与する複雑な構造的変動を解決できないため、制限されています。14
この記事では、速度、再現性、スケーラビリティを向上させるために、新しい再設計されたゲノムシーケンスワークフローの研究と開発における当社の関与について説明します。rWGSは13.5時間です。このパイプラインはRady Children’s Institute for Genomic Medicineと共同で開発され、イルミナの次世代シーケンサー(NGS)テクノロジーによって支えられており、ライブラリー調製、シーケンス、バリアントコーリングを最適化することで、従来のrWGSワークフローにおける重要なボトルネックをなくし、複雑な構造変化を含む遺伝的バリアントの迅速かつ正確な同定を実現します。
最適化された13.5時間のゲノムシーケンスワークフロー
これまでの研究から、20時間以内に原因となる遺伝的変異を正しく特定するrWGSパイプラインを報告しました。9 有望ではありますが、この手法にはスケーラビリティがなく、構造的変異を適切に捉えることができませんでした。このレポートでは、rWGSワークフローを最適化し、コピー数バリアント(CNV)および構造バリアント(SV)のスケーラビリティ、ターンアラウンドタイム、および解析性能を向上させるためのさらなるイノベーションについて説明します(図1)。

VC:バリアントコーリング、SV:構造バリアント、TSS:TruSight Software Suite、GEM:Fabric GEMゲノム解釈プラットフォーム、MOON:Invitaeバリアント解釈ソフトウェア。* 660分間は、カスタマイズされたランレシピを使用し、サイクルタイムを最大限短縮しました。
Illumina DNA PCR-Free Prepによるライブラリー調製の迅速化
rWGSのサンプル調製における重要な課題は、少量のサンプルから高品質のゲノムDNAを精製することです。エチレンジアミン四酢酸(EDTA)血液サンプルから直接シーケンスライブラリーを調製したか、または新生児の必須スクリーニングで頻繁に使用されるサンプルタイプであるNucleic Card Matrix乾燥血液スポット(Thermo Fisher、カタログ:4473977)から3 mmパンチを5つ調製しました。イルミナDNA PCR-Free Prep, Tagmentationは、ビーズ結合トランスポソームを使用して、ハンズオンタイムを短縮しながら一貫したインサートサイズを提供するもので、ライブラリー調製に使用されました。オンビーズタグメンテーションは、CNVおよびSVを同定する能力を維持しながら、中間DNA精製ステップの必要性を排除します。インキュベーション時間を最大限短縮し、ライブラリー出力をノーマライズしました。これにより、ライブラリー定量キット(KAPA Biosystems、カタログ KK4824-079601666001)。全体として、ライブラリー調製は精製されたゲノムDNAから平均45分、血液から平均72分で完了しました。
NovaSeq−(* 6000システムを使用した高性能シーケンス
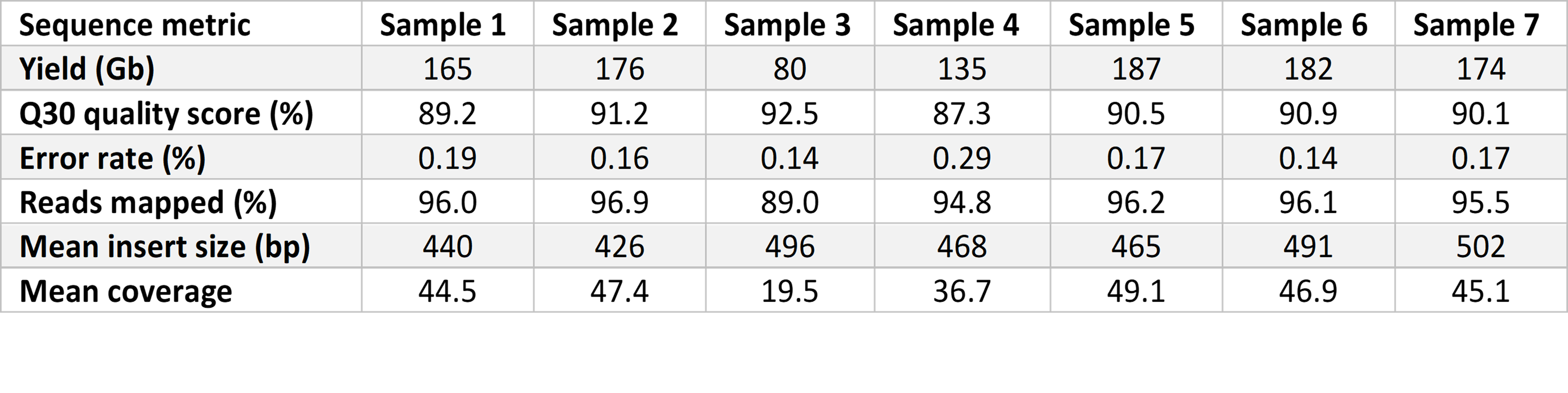
調製済みライブラリーは、高品質のシーケンス出力のためにバージョン1.5試薬のSPフローセルを使用して、NovaSeq 6000システムで2 × 101 bpサイクルでシーケンスしました。フローセルを、2つのレーンのそれぞれについて単一表面でイメージングしました。サイクルタイムを最大限短縮し、シーケンス品質を維持できるカスタマイズされたランレシピを使用しました。シーケンスの収量は、40倍以上のカバレッジで約150 Gb、30を超えるPhred品質スコア(Q30)で87%のリードでした(表1)。シーケンスランの平均時間は11時間12分でした。
Dynamic Read Analysis for GENomics(DRAGEN−(* v.3.7)による迅速なシーケンスアライメントとバリアントコーリング
スピード、感度、および精度に高度に最適化されたイルミナDRAGENプラットフォームは、シーケンスアライメントとバリアントコーリングに使用されました。NovaSeq 6000シーケンスシステムからDRAGENプラットフォームへのファイルの転送は自動化されました。シーケンスをヒトゲノムアセンブリGRCh37(hg19)にアライメントし、バリアントをDRAGENプラットフォームv.3.7.5で同定し、ジェノタイピングしました。バリアントコールファイルは、イルミナTruSight−(* Software SuiteやサードパーティソフトウェアMOON(Invitae)、GEM(Fabric Genomics)などのバリアント解釈ソフトウェアに自動的にルーティングされました。三次解析は3つのソフトウェアプラットフォームすべてで並行して実行されました。デシジョンツリー、ベイズモデル、ニューラルネットワーク、自然言語処理を使用したシーケンスフィルタリングとランク付けバリアントを適用し、バリアント同定のための出力を生成しました。
この改善されたrWGSパイプラインの分析性能は、2つのリファレンスDNAサンプル(NA12878およびNA24385)を使用して評価されました。これらのリファレンスサンプルには、SNVおよび挿入欠失(indel)(NISTv4.1)、SVおよびCNV(NISTv0.6)について、米国国立標準技術研究所(NIST)によって確立されたゴールドスタンダードバリアントセットが含まれています。Witty.Er。DNAサンプル調製からバリアントコーリング完了までの平均時間は12時間42分で、DRAGEN v3.5.3を使用した以前のベンチマークと比較して35%改善しました(図2)。
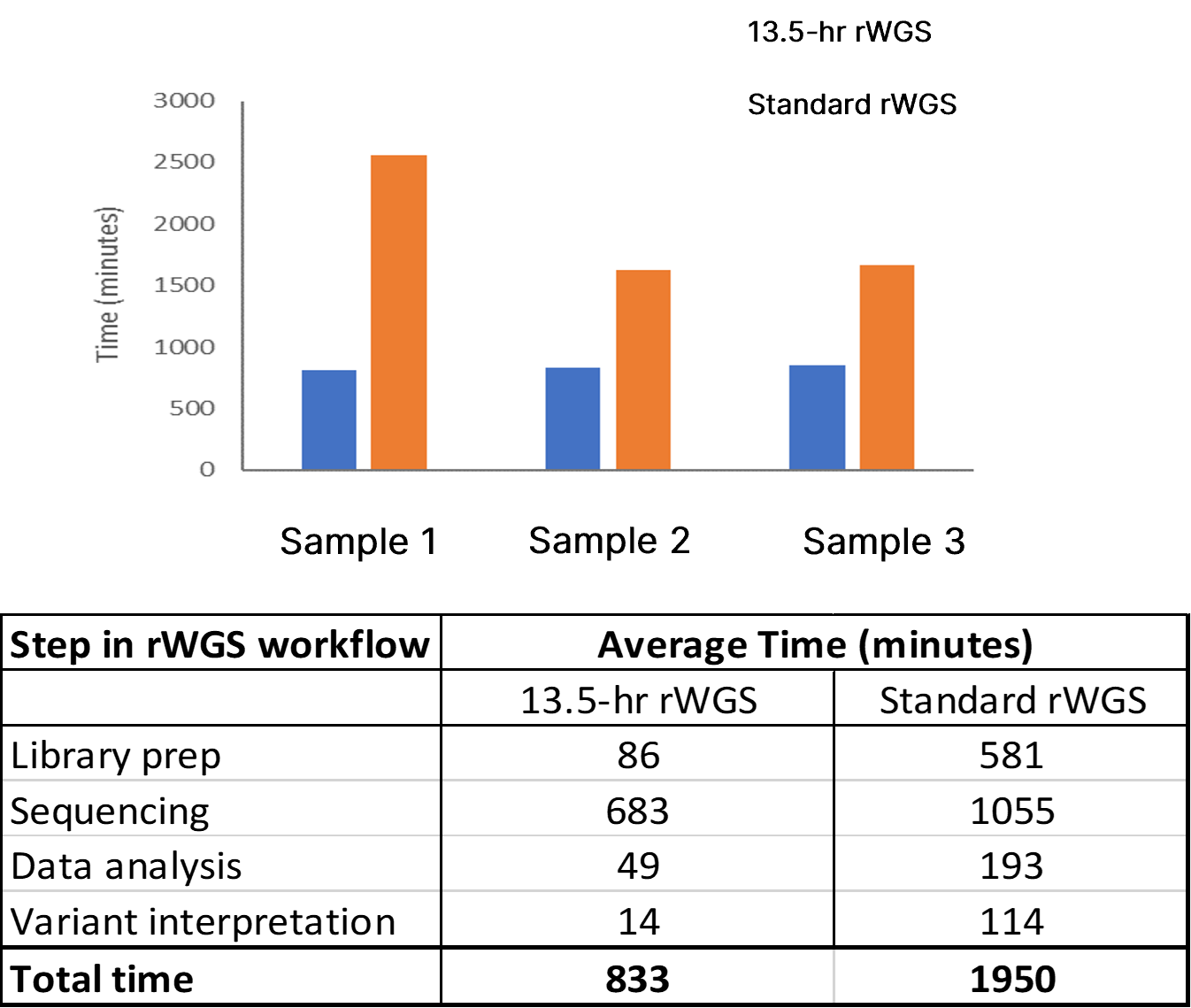
トップパネル:13時間rWGS(青)法と標準rWGS(オレンジ)法を使用した3つのテストサンプルにおける完了までの合計時間。下部パネル:13.5時間および標準rWGSパイプラインを使用したrWGSワークフローの各ステップでの平均所要時間。
構造バリアント検出のためのDRAGEN v.3.7の優れた解析性能
DRAGEN v.3.7の性能を、SV(サイズ50 nt)およびCNV(サイズ>10 > kb)の検出に一般的に使用されるツールであるMantaおよびCNVnatorと比較しました。DRAGEN v.3.7は、挿入SV(DRAGENでは49%、Mantaでは27%)および欠失CNV(DRAGENでは88%、CNVnatorでは9%)に対して優れた感度を示しました(表2)。ベースコールからFASTQ形式への生データの変換、リファレンスゲノムへのアライメント、バリアントコーリングを含む一次解析と二次解析の平均時間は、MantaとCNVnatorを使用した3時間9分と比較して、DRAGEN v.3.7を使用した場合の47分で有意に改善されました。

リファレンスサンプルNA24385でDRAGEN v.3.7およびManta/CNVnatorを使用した場合、NISTベンチマークを使用した場合、CNV欠失の検出において精度とリコールが有意に改善されました。SNV:一塩基変異、indel:挿入/欠失、SV:構造バリアント(サイズ>50 bp)、CNV:コピー数変異(サイズ>10 kb)。
13.5時間rWGSパイプラインの潜在的な有用性
13.5時間のrWGSパイプラインでは、7つの検査サンプルのうち6つのサンプルで遺伝的変異の正確な同定ができました。原因となる遺伝的変異は、標準的なrWGSでは平均42時間であったのに対し、当社のrWGSワークフローを用いて14時間以内にすべてのサンプルで同定されました。完了までの最短時間は13時間13分でした。当社のrWGSワークフローは、速度と再現性、スケーラビリティを兼ね備えているため、一般化可能なハイスループットアプリケーションに最適です。
まとめ
この記事では、テストサンプルにおける原因となる遺伝的変異を迅速かつ正確に同定するために最適化された、リエンジニアリングされたrWGSパイプラインについて概説します。NovaSeq 6000システムによるライブラリー調製の高速化や高性能シーケンスなど、rWGSワークフローにおける重要なステップを、DRAGEN v.3.7バリアントコーリングの精度と速度と組み合わせることで、rWGSは13.5時間で完了できました。このrWGSパイプラインでは、シーケンス速度の向上とサードパーティのバリアント解釈ツールとの互換性に加え、構造的バリエーションを含むバリアントを高精度で迅速に検出できます。
参考文献
1. Gunne E, McGarvey C, Hamilton K, Treacy E, Lambert DM, Lynch SA。アイルランドにおける小児死亡率に対する希少疾患の寄与に関する後ろ向きレビュー。Orphanet J Rare Dis . 2020;15(1):311. doi:10.1186/s13023-020-01574-7Weiner
2. Weiner J, Sharma J, Lantos J, Kilbride H. 新生児集中治療室での乳児の死亡例:1999年から2008年までの傾向。Arch Pediatr Adolesc Med . 2011;165(7):630-634. doi:10.1001/archpediatrics.2011.102
3. Murphy SL, Xu J, Kochanek KD, Arias E. Mortality in the United States, 2017。NCHSデータ概要。2018年(328):1-8.
4. Arth AC、Tinker SC、Simeone RM、Ailes EC、Cragan JD、Grosse SD。あらゆる年齢の人の出生異常に関連する入院費用 - 米国、2013年。MMWR Morb Mortal Wkly Rep . 2017;66(2):41-46。
5. Berry MA, Shah PS, Brouillette RT, Hellmann J. 小児病院新生児集中治療室に入院した新生児の死亡率と入院期間の予測。Jペリナトール。2008;28(4):297-302。
6. Petrikin JE, Cakici JA, Clark MM, et al. NSIGHT1-randomized比較対照試験:重症乳児における迅速な病因診断のための迅速な全ゲノムシーケンス。NPJ Genom Med . 2018;3:6. doi:10.1038/s41525-018-0045-8
7. Saunders CJ, Miller NA, Soden SE, et al. 新生児集中治療室における遺伝性疾患診断のための迅速な全ゲノムシーケンス。Sci Transl Med . 2012;4(154):154ra135. doi:10.1126/scitranslmed.3004041
8. Petrikin JE, Willig LK, Smith LD, Kingsmore SF. 迅速な全ゲノムシーケンスと精密な新生児学。Semin Perinatol . 2015;39(8):623-631. doi:10.1053/j.semperi.2015.09.009Farnaes
9. Clark MM, Hildreth A, Batalov S, et al. 迅速な全ゲノムシーケンスと自動表現型解析と解釈による、重症児の遺伝性疾患の診断。Sci Transl Med . 2019;11(489):eaat6177. doi:10.1126/scitranslmed.aat6177
10. Kingsmore SF, Cakici JA, Clark MM, et al. シングルトン、トリオ、ラピッドゲノム、エクソームシーケンスの解析および診断性能を疾患児を対象にした無作為化比較対照試験。Am J Hum Genet . 2019;105(4):719-733. doi:10.1016/j.ajhg.2019.08.009
11. Farnaes L, Hildreth A, Sweeney NM, et al. 迅速な全ゲノムシーケンスは、乳児の罹患率と入院費用を減らします。NPJ Genom Med . 2018;3:10. doi:10.1038/s41525-018-0049-4
12. Willig LK, Petrikin JE, Smith LD, et al. 重症乳児におけるメンデル性疾患の同定のための全ゲノムシーケンス:診断および臨床所見のレトロスペクティブ解析。Lancet Respir Med . 2015;3(5):377-387. doi:10.1016/S2213-2600(15)00139-3
13. Sanford EF, Clark MM, Farnaes L, et al. 迅速な全ゲノムシーケンスは、PICUの小児において臨床的有用性があります。Pediatr Crit Care Med . 2019;20(11):1007-1020. doi:10.1097/PCC.0000000000002056
14. Maron JL, Kingsmore SF, Wigby K, et al. ゲノム検査の臨床統合に関連する新たなバリアントの発見と課題: 新生児および乳児疾患のためのゲノム医学の中間報告書(GEMINI)研究。JAMA Pediatr . 2021;175(5):e205906. doi:10.1001/jamapediatrics.2020.5906