はじめに
血液中を循環するセルフリーDNA(cfDNA)は、体内に存在するさまざまな組織、腫瘍、または微生物から生じます。血液中のcfDNAのリキッドバイオプシー解析は、がんを含む特定の疾患の評価とモニタリングのための比較的非侵襲的な手法です。1 腫瘍はDNAを排出します。血中循環腫瘍DNA(ctDNA)と呼ばれていますが、これは血液中に存在する全cfDNAのうちのごく一部です。そのため、血流中に存在する低濃度のctDNAの変異を検出するには、ロバストなアッセイが必要になります。
次世代シーケンサー(NGS)法は、ctDNAのバリアントを高感度で特異的に検出してシーケンスを行うことを可能にします。ctDNAのシーケンスにおける大きな課題は、その希少性から生じます。微量のctDNAから低頻度のバリアントを同定する能力は、ライブラリー調製、PCR増幅、シーケンス中に生じるさまざまなエラーやアーティファクトによって交絡し、すべてのノイズから真のシグナル(遺伝子バリアント)を区別することが困難になります。この記事では、分子バーコード(UMI)とDRAGENプラットフォームのパワーを利用してノイズとエラーを削減し、ctDNAにおいて精確な体細胞バリアントコールを可能にする方法を紹介します。
手法
核酸の調製
cfDNAは、StreckまたはEDTA血液チューブ中の血漿2.0~6.0 mlからQIAamp Circulating Nucleic Acid Kit(QIAGEN、カタログ番号 55114)を使用して抽出されました。抽出されたcfDNAは、75~250 bpの領域をターゲットとするFragment Analyzer System(Agilent Technologies, Inc.)を使用してキャピラリー電気泳動を行い、定量化しました。
ライブラリー調製
シーケンスライブラリーは、TruSight™ Oncology 500 ctDNA Kit(48サンプル、カタログ番号 20039252)を使用して作成し、サンプルごとにcfDNA物質を30 ngインプットしました。DNA断片の突出末端を平滑化/末端平滑化し、Aテールを付加し、PCRを介したサンプルのバーコード付けの前にアダプターにライゲーションしました。 分子バーコード(UMI)を含むアダプターをDNA断片にライゲーションし、エラー補正のためにPCR増幅中にデュプレックスバーコードを付加しました。すべてのサンプルのターゲット領域を濃縮するには、TruSight OncologyインデックスPCR製品を使用し、57°Cで2つのハイブリダイゼーションステップを必要としました。AccuClear Ultra High Sensitivity dsDNA Quantitation Kit(Biotium、カタログ番号31028-T)を使用して、濃縮後のライブラリーの収量が十分になるようにしました。濃縮後のライブラリーは、ビーズベースのノーマライゼーションを用いてノーマライズし、等量でプールし、変性させ、さらに適切なローディング濃度に希釈しました。
シーケンス
NovaSeq 6000 S4 Reagent Kit v1.5(300 cycles、カタログ番号 20028312)を使用し、各レーンローディング(レーンあたり6プレックス、フローセルあたり24プレックス)のXPワークフローに従って、151 bpのペアエンドリードを用いてNovaSeq 6000システム上で調製済みライブラリーのシーケンスを実行しました。平均して、各サンプルからライブラリーあたり約10億リードが得られました。
データ解析
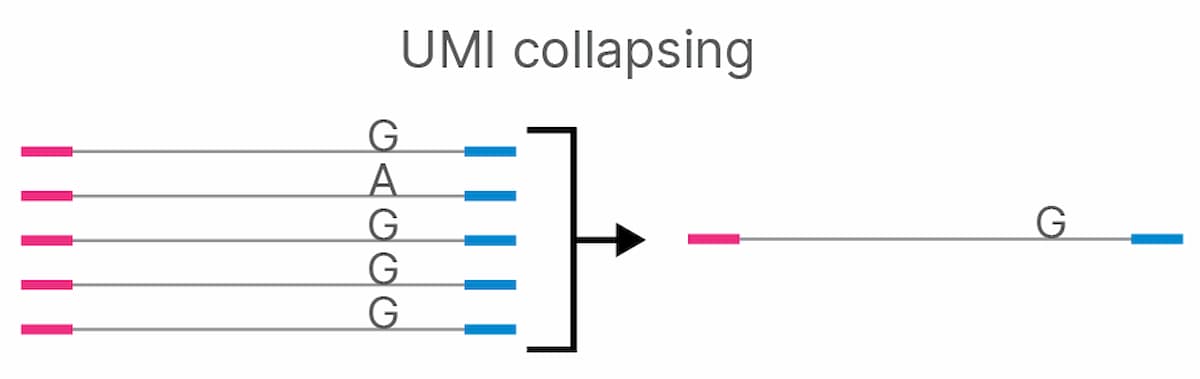
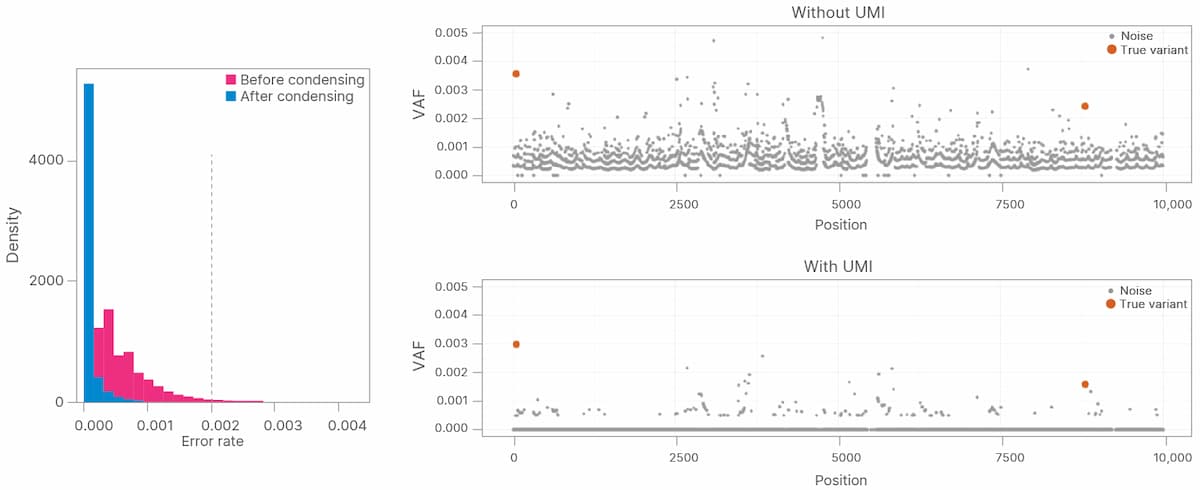
低頻度バリアントを維持しながら、重複リード(同一のUMIを持つシーケンスリード)をPCRの重複とシーケンスエラーを除去した単一のコンセンサス配列にコラプシングするために、UMIバーコードが使用されました(図1)。1本のストランドから取得したリードによってサポートされるコラプスされたシーケンスはシンプレックスシーケンスと呼ばれ、順鎖と逆鎖の両方からのリードによってサポートされるシーケンスは、デュプレックスシーケンスと呼ばれます。デュプレックスシーケンスでは、両ストランドで同一のエラーを持つ確度が低いため、エラー率[偽陽性/偽陰性(FP+FN)]がさらに低下します。
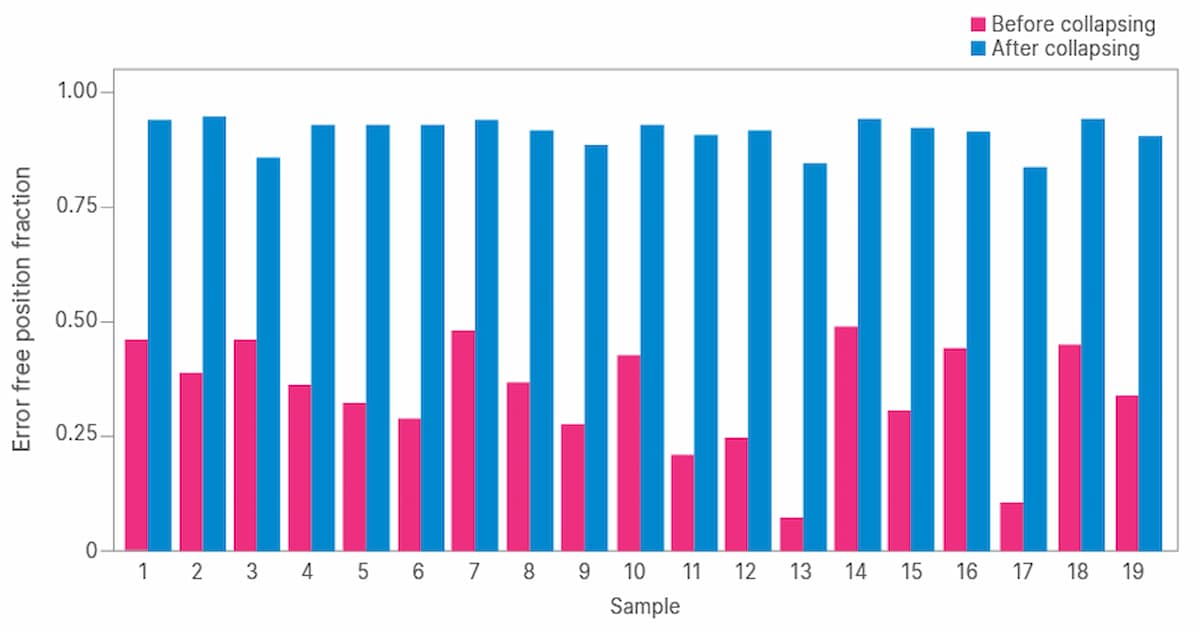
DRAGENプラットフォームのUMIによるエラー補正の性能を評価するために、2つのメトリクスが決定されました。エラーフリー位置比率は、リファレンスシーケンスと一致する各ゲノム位置のすべてのシーケンスリードにおけるベースコールの割合を示します。平均エラー率は類似していますが、すべてのゲノム位置にわたって平均化されています。
結果
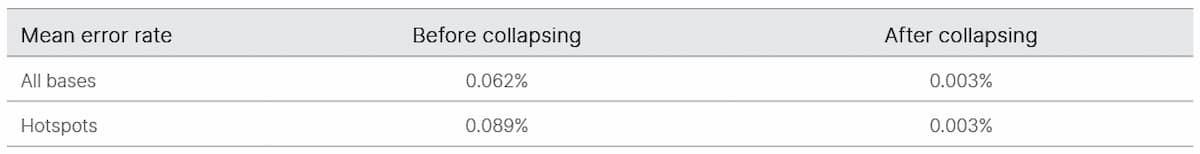
ctDNAシーケンスのノイズ低減のために、TruSight Oncology UMI試薬を評価することから始めました。19サンプルのすべてについて、UMIがコラプシングする前後のエラーフリー位置比率の測定を行いました(図2)。エラーフリー位置平均パーセントは、コラプシング前では36.3%、コラプシング後では92.9%に増加しました(表1)。

まとめ
リキッドバイオプシーによるNGSベースのctDNA解析により、さまざまな腫瘍学アプリケーションを使用して腫瘍の特性を評価し、がんの発生と進行をより良く理解することが可能になります。しかし、ctDNAの希少性のために、真のシーケンスバリアントからノイズやエラーを識別することは困難です。本稿では、UMIとDRAGENプラットフォームを組み合わせることでエラーを大幅に削減できる方法をご紹介しました。しかし、当社は系統ノイズのフィルター、加齢に伴うアーティファクトの低減など、精度を高めるためにその他の戦略も採用しています。これらの手法を組み合わせることで、ctDNAの精確な体細胞バリアントコールが可能になります。
詳細はこちら
学術用途向けの詳細情報またはDRAGEN試用版ライセンスについては、dragen-info@illumina.comまでお問い合わせください。
注釈
- 1. Bettegowda C, Sausen M, Leary RJ, et al. Detection of circulating tumor DNA in early- and late-stage human malignancies Sci Transl Med. 2014;6(224):224ra24.