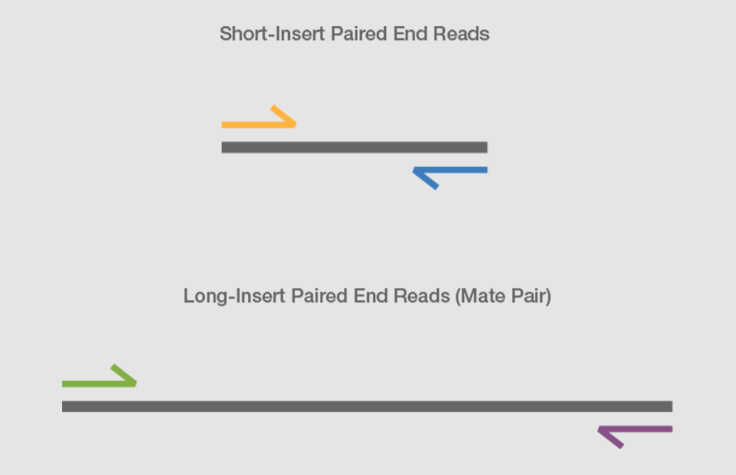
メイトペアシーケンス
メイトペアライブラリー調製プロセス
DNA断片化の後、DNA断片は標識dNTPで最終修復されます。DNA断片は環状化され、非環状化DNAは消化によって除去されます。円形DNAは断片化され、標識された断片(結合した元のDNAの末端に対応)はアフィニティー精製されます。精製された断片は末端修復され、イルミナのペアエンドシーケンスアダプターにライゲーションされます。
フローセルオリゴヌクレオチドに相補的な追加の配列が、テールPCRプライマーを用いてアダプター配列に追加されます。最終的に調製されたライブラリーは、元々数キロベースで分離されていた2つのDNAセグメントで構成される短い断片で構成されています。これらのライブラリーは、ペアエンドクラスター形成、続いてイルミナ次世代シーケンス(NGS)システムを使用したシーケンスの準備が整いました。

メイトペアシーケンスのハイライト
高いゲノム多様性
効率的なプロトコールにより、あらゆる次世代プラットフォームの中で最高のゲノム多様性を実現します。
ユーザーフレンドリーなワークフロー
ハンズオンタイムが限定され、複数の停止ポイントがあるシンプルなワークフロー。
低DNAインプット要件
わずか1 μgの出発物質が必要です。
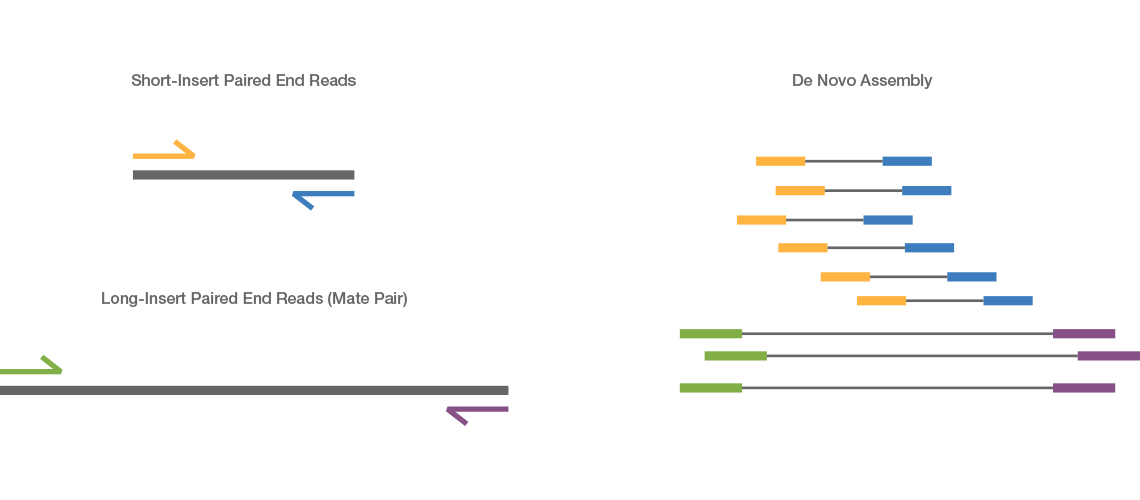
Kiwiゲノムアセンブリのメイトペアシーケンス
研究者が、キウイ鳥ゲノムのde novoアセンブリにメイトペアシーケンスをどのように使用したかをご覧ください。
イルミナからご研究分野に応じたニュースレター、研究事例や情報などをお送りいたします。ご登録はこちらから
Nextera Mateペアライブラリーキットは製造中止となりました。交換用キットはありません。