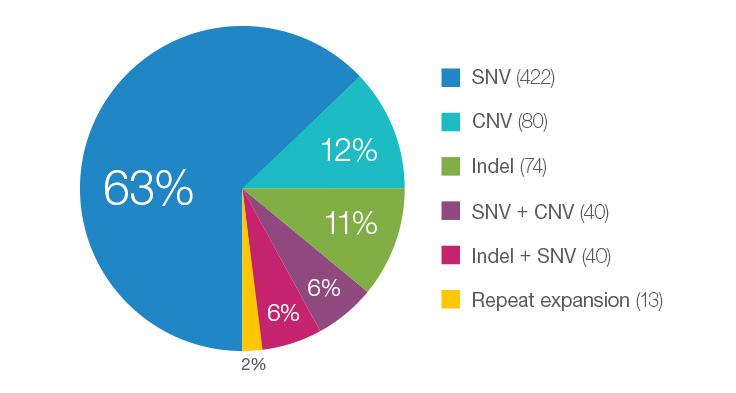
希少疾患の全ゲノムシーケンス
希少疾患に対する最も包括的な検査1-8
全ゲノムシーケンスは、希少な遺伝性疾患の診断がつく可能性が最も高い10
希少疾患におけるWGSの利点
希少疾患のための全ゲノムシーケンス(WGS)は、他の遺伝子検査方法と比較して、3つの重要な利点があります。
- 診断率を向上させる可能性10
- 業務効率の向上
- コストの削減3,6
特に希少疾患の症例では、全ゲノムシーケンスは、1回のアッセイで複数のバリアントタイプを検出できる最も包括的な検査です。1-8大規模ランダム化比較試験において、新生児集中治療および小児集中治療患者の診断までの時間の中央値は、標準検査では107日であるのに対し、WGSでは13日でした。9
WGSは希少疾患の診断を求める終わりなき旅に終止符を打つことができる
希少疾患の全ゲノムシーケンスは、医師が遺伝性疾患を迅速に診断するのに役立ち、家族の診断を求める終わりなき旅を避けることができます。すべてのゲノム検査法の中で、WGSは診断がつく可能性が最も高い検査法です。10他の検査法でカバーされていない領域だけでなく、他の検査法が対象とする領域内でも、ヒトゲノムのカバー率が最も高くなります。11,12ファーストラインでのこの高いカバー率は、不必要な反復検査の必要性を減らし、NICUでの入院期間を短縮できることを示しています。13,14
WGSは患者さんの治療にも影響を与えることができます。WGSによる診断を受けた小児科外来患者の49~75%において、管理の変更が報告されています。15,16
希少疾患のためのWGSへのアクセス拡大
Medical Genome Initiativeは、ベストプラクティスを公開することで、遺伝性疾患診断のための高品質なWGSへのアクセスを拡大することを目的としています。
記事を読む希少疾患WGSの専門家に聞く

希少疾患に対する臨床WGSの標準化
The Hospital for Sick ChildrenのChristian Marshall博士が、実験室および臨床のベストプラクティスにより、遺伝性疾患診断のための全ゲノムシーケンスがどのように可能になるかについて解説しています。
WGSと診断を求める終わりなき旅の終わり
Duke UniversityのVandana Shashi博士とUndiagnosed Diseases NetworkのKimberly LeBlanc氏が、WGSが希少疾患患者の診断を求める終わりなき旅をいかに短縮するかについて語っています。

重症児の迅速な全ゲノムシーケンス
Shimul Chowdhury博士が、迅速なWGSが子供の希少疾患の原因を突き止めるのに役立つことを説明しています。
主な希少疾患WGSの発表論文
希少疾患診断に関する10万人ゲノムパイロット試験
国民医療システムにおけるWGSのパイロットスタディでは、さまざまな希少疾患における診断率の向上が示されました。
遺伝性疾患が疑われる乳児の臨床管理に対するWGSの効果
WGSへのアクセスにより、精密診断を受け、臨床管理が変更された患者の割合が2倍となりました。
WGSの医療現場への導入
WGSによって解析された3,219人の希少疾患患者における、複数の臨床疾患にわたる高い診断率。

臨床現場でのWGSのパフォーマンス、有用性、および実装
この一連の出版物は、WGSが単一のアッセイで複数のバリアントタイプを検出するための最も包括的なテストであり、急性疾患の乳児および診断されていない小児外来患者における現在の遺伝子検査アプローチよりも迅速に遺伝子診断を提供できることを示しています。
概要小冊子を見る参考文献
- Lionel AC, Costain G, Monfared N, et al. Improved diagnostic yield compared with targeted gene sequencing panels suggests a role for whole-genome sequencing as a first-tier genetic test.Genet Med 2017;8月3日 doi: 10.1038/gim.2017.119.
- Sanghvi RV, Buhay CJ, Powell BC, et al. Characterizing reduced coverage regions through comparison of exome and genome sequencing data across 10 centers.Genet Med. 2018;20(8):855-866. doi:10.1038/gim.2017.192
- Dolzhenko E, van Vugt JJFA, Shaw RJ, et al. Detection of long repeat expansions from PCR-free whole-genome sequence data.Genome Res. 2017;27(11):1895-1903. doi:10.1101/gr.225672.117
- Gross A, Ajay SS, Rajan V, et al. Copy number variants in clinical WGS: deployment and interpretation for rare and undiagnosed disease.Genetic Med. 2019;21(5):1121-1130.
- Alfares A, Aloraini T, Subaie LA, et al. Whole-genome sequencing offers additional but limited clinical utility compared with reanalysis of whole-exome sequencing.Genet Med. 2018;20(11):1328-1333. doi:10.1038/gim.2018.41
- Lindstrand A, Eisfeldt J, Pettersson M, et al. From cytogenetics to cytogenomics: whole genomes sequencing as a first-line test comprehensively captures the diverse spectrum of disease-causing genetic variation underlying intellectual disability.Genome Med. 2019;11(1):68.
- Chen, X, Sanchis-Juan, A., French, CE et al. Spinal muscular atrophy diagnosis and carrier screening from genome sequencing data.Genet Med. 2020; 22:945–953. https://doi.org/10.1038/s41436-020-0754-0
- Chen X, Schulz-Trieglaff O, Shaw R, et al. Manta: rapid detection of structural variants and indels for germline and cancer sequencing applications.Bioinformatics. 2016;32(8):1220–1222. http://doi.org/10.1093/bioinformatics/btv710.
- Petrikin JE, Cakici JA, Clark MM, et al. The NSIGHT1-randomized controlled trial: rapid whole-genome sequencing for accelerated etiologic diagnosis in critically ill infants.NPJ Genom Med. 2018;3:6. 2018年2月9日発表。doi:10.1038/s41525-018-0045-8
- Clark, M.M., Stark, Z., Farnaes, L. et al. Meta-analysis of the diagnostic and clinical utility of genome and exome sequencing and chromosomal microarray in children with suspected genetic diseases. npj Genomic Med. 2018;3:16. https://doi.org/10.1038/s41525-018-0053-8
- Meienberg J, Bruggmann R, Oexle K, Matyas G. Clinical sequencing: is WGS the better WES?Hum Genet. 2016;135(3):359-362. doi:10.1007/s00439-015-1631-9
- Belkadi A, Bolze A, Itan Y, et al. Whole-genome sequencing is more powerful than whole-exome sequencing for detecting exome variants.Proc Natl Acad Sci U S A. 2015 Apr 28;112(17):5473-8. doi: 10.1073/pnas.1418631112
- Farnaes L, Hildreth A, Sweeney NM, et al. Rapid whole-genome sequencing decreases infant morbidity and cost of hospitalization.NPJ Genom Med. 2018;3:10. 2018年4月4日発表 doi:10.1038/s41525-018-0049-4
- Stark Z, Lunke S, Brett GR, et al. Meeting the challenges of implementing rapid genomic testing in acute pediatric care.Genet Med. 2018;20(12):1554-1563. doi:10.1038/gim.2018.37
- Scocchia A, Wigby KM, Masser-Frye D, et al. Clinical whole genome sequencing as a first-tier test at a resource-limited dysmorphology clinic in Mexico.NPJ Genom Med. 2019;4:5. 2019年2月14日発表。doi:10.1038/s41525-018-0076-1
- Bick D, Fraser PC, Gutzeit MF, et al. Successful application of whole genome sequencing in a medical genetics clinic.J Pediatr Genet. 2016;6(2):61-76.