がん免疫研究
腫瘍を特性付けるゲノムツール
免疫腫瘍学は、腫瘍が自然免疫反応を回避する方法の理解を深めることで、がんとの闘いにおいて大きな進歩を遂げた新興分野です。がん免疫研究を牽引する研究者たちは、次世代シーケンサー(NGS)を活用し、免疫療法反応因子、バイオマーカー、ゲノムを研究することで、個別化免疫療法の研究を向上させています。
腫瘍が免疫反応を回避するために用いるメカニズムに関するがん免疫研究は、有望な治療ターゲットにつながっています。これらの療法は、免疫システムのがんを標的とする能力を高めたり、腫瘍が免疫反応を回避する能力を制限したりします。さらに、腫瘍環境においてどのパスウェイが活性化され、それががん細胞の増殖、生存、浸潤、転移などのプロセスにどのように関わっているかを同定するのに、NGSは役立ちます。

免疫療法:次世代のがん治療
このアプリケーションノートでは、進化する傾向、研究者のニーズ、この急速に進化する分野で利用できるゲノムテクノロジーなど、免疫腫瘍学の最近の進歩を強調しています。
NGSを利用したがん免疫研究のソリューション
腫瘍微小環境と免疫組成
次世代シーケンサーは、がんゲノムの詳細な解析を提供できる強力なツールです。また、腫瘍増殖または治療に反応する免疫マーカー発現をモニタリングするために、高感度で腫瘍微小環境をリアルタイムで効率的に評価することもできます。NGSは、免疫細胞のレパートリーを特徴付け、微小環境内のさまざまな細胞集団を同定し、同時に数千のターゲットにおける遺伝子発現を包括的に定量することができます。
腫瘍変異負荷とネオアンチゲン予測
次世代シーケンサーは、研究者らがネオアンチゲンの同定、免疫反応を高める革新的な治療法の研究、および遺伝的変異がその効果に与える影響を解明するのにも役立ちます。NGSは、腫瘍特異的な反応を誘発するために適用できる新規の腫瘍抗原の予測的な選択も可能にします。
腫瘍微小環境解析のための空間トランスクリプトミクス
空間トランスクリプトミクスは、組織切片にマッピングされた遺伝子発現パターンのトポグラフィー配置を提供し、構造と活性を関連付けます。この能力により、研究者は細胞レベルで生物学的相互作用を明確にし、腫瘍微小環境などの複雑な組織に対する新しい洞察を得ることができます。
がんにおける腸内細菌の組成
宿主微生物相互作用ががんの発生、進行、治療効果に及ぼす影響を研究するために、NGSが使用できます。NGSによってさまざまな状況で微生物群をプロファイルでき、このプロファイリングは新たながん治療法の開発でターゲットとなりうる種や症状を同定する上で非常に重要です。
腫瘍微小環境におけるシングルセル解析
シングルセルRNAシーケンス(scRNA-Seq)は、シングルセル解像度で、がんや腫瘍微小環境のトランスクリプトームプロファイルを調べるためにますます使用されています。免疫細胞研究は、シングルセルトランスクリプトミクスと組み合わせて、がんの免疫サーベイランスと免疫療法に対する耐性における主要な要因を理解するために適用されています。
NGSベースのがん研究アプリケーション
| ネオアンチゲン、 変異負荷 |
発現 プロファイリング |
マイクロバイオーム(16S) シーケンス |
TCR/BCRa プロファイリング |
エピジェネティクス プロファイリング |
|
|---|---|---|---|---|---|
| 治療への応用 | |||||
| チェックポイント阻害剤 | |||||
| ワクチン | |||||
| 養子細胞療法 | |||||
| 予後 | |||||
| 微生物 | |||||
| 免疫レパートリー | |||||
| 監視 |
- 主なアプリケーション
a TCR:T細胞受容体、BCR:B細胞受容体
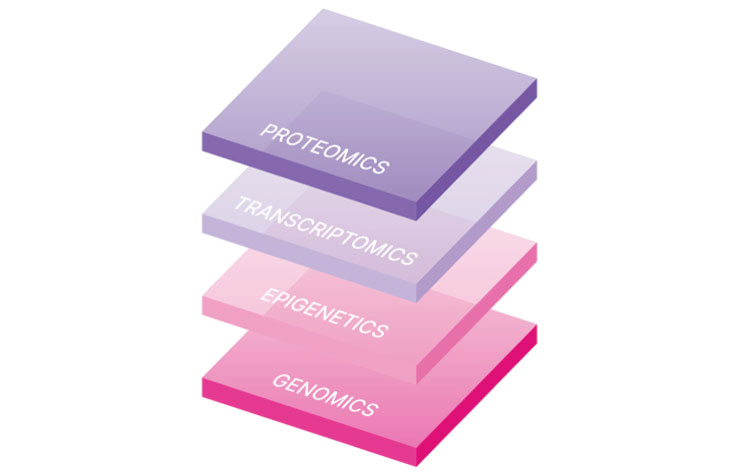
腫瘍学研究におけるマルチオミクス:複数の情報レイヤーにより、単一オミクス研究だけでは解決できない腫瘍生物学に対する新しい洞察が得られます。
がん免疫研究におけるマルチオミクスアプローチ
マルチオミクス研究は、ゲノム、エピゲノム、トランスクリプトーム、メタゲノム、プロテオミクスのアプローチからの高次元データセットを統合し、多くの場合、計算およびネットワーク生物学を使用して、これらの技術によって生成される膨大な量のデータを解釈します。臨床研究データにマルチオミクスを適用すると、既存のオミクス手法の探索力が強化され、新しいバイオマーカーや免疫療法のターゲットが明らかになります。
さらに、マルチオミクスアプローチは、腫瘍および微小環境の分子プロファイルの包括的な見方を提供することができます。
免疫療法研究のためのNGSワークフロー
イルミナでは、がん免疫研究に対応したデータ解析オプションが利用できるライブラリー調製およびシーケンスオプションを複数ご用意しています。効率化されたワークフローとフレキシブルなキット構成で、さまざまな研究デザインに対応できます。
ライブラリー調製
AmpliSeq for Illumina Immune Repertoire Plus, TCR beta Panel
T細胞受容体β鎖の再配列のシーケンスにより、T細胞多様性とクローン増殖を調査するターゲットRNA研究パネル。
Illumina Stranded Total RNA Prep w/ RiboZero Plus
コーディングと複数形態の非コーディングRNAの解析にすぐれたパフォーマンスを発揮します。代替転写産物、遺伝子融合、およびアレル特異的発現を高い信頼性で発見できます。
Illumina DNA Prep
ヒト全ゲノムシーケンスからアンプリコン、プラスミド、微生物種まで、広範囲の用途に対して迅速な統合ワークフロー。
シーケンス
NovaSeq Xシリーズ
NovaSeq Xシリーズは、膨大なアプリケーションを幅広く提供することで、生産規模でのデータ集約型の手法を可能にします。
NovaSeq 6000システム
拡張性のあるスループットと柔軟性により、ほぼすべてのゲノム、シーケンス手法、プロジェクト規模に対応します。
NextSeq 1000および2000システム
コスト効率に優れ、使いやすいミッドスループットのベンチトップシーケンサーは、新たなアプリケーションをサポートする極めて高い柔軟性を備えています。
データ解析
BaseSpace Sequence Hub
NGSデータ解析と管理を行うイルミナのゲノミクスコンピューティング環境。
BaseSpace Sequence Hub RNA Ampliconアプリ
RNA Ampliconアプリは、イルミナプラットフォームでシーケンスされたアンプリコンパネルを使用した遺伝子発現プロファイリングを可能にします。
Local Run Manager RNAアンプリコンモジュール
シーケンスランの作成、ランステータスのモニタリング、シーケンスデータ解析、および結果の表示を行うために設計された統合ソリューションです。

がん研究手法に関するガイド
がん研究手法に関するガイドは、幅広いがん研究アプリケーションのためのシンプルで包括的なワークフローを紹介した40ページ以上にわたる包括的なリソースです。このガイドには、シングルセルシーケンス、空間シーケンス、メチル化プロファイリング、マルチオミクス、セルフリーRNAシーケンスなどが含まれます。
無料ガイドをダウンロード免疫腫瘍学リソース
ゲノミクスによる腫瘍学の変革
オンタリオがん研究所とUnited Health Networkの研究者がゲノム研究を通じてがん生物学についてより深く知ることで、大学や医療システムが、がんとの闘いを前進させる方法を紹介します。
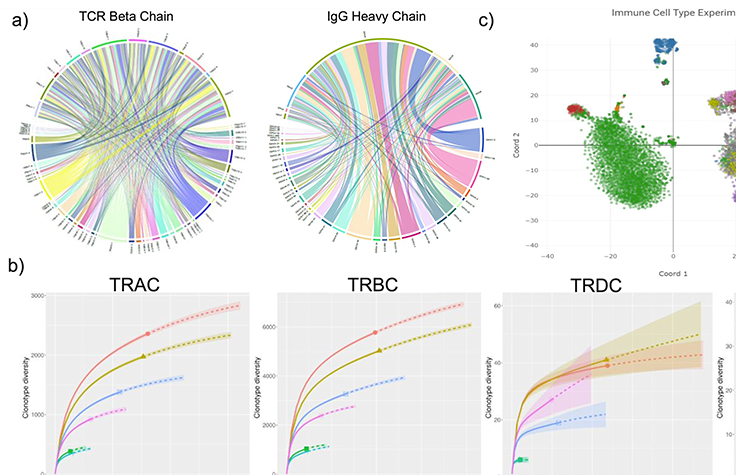
がん研究のためのIR-Seqの実現
このダウンロード可能なポスターは、がん研究用の新しいスケーラブルなツールの概要を示しており、完全長V(D)J免疫レパートリーシーケンス(IR-Seq)を可能にします。
TruSight Oncology製品
組織から液体生検サンプルまで、包括的なゲノムプロファイリングを可能にする当社のソリューションをご覧ください。
関連リソース
がん研究の概要
イルミナのシーケンステクノロジーとマイクロアレイテクノロジーは、DNAからRNA解析、エピジェネティクスなど、がんゲノム研究の応用分野を幅広くサポートしています。
がん研究アプリケーション
リキッドバイオプシー研究、シングルセル解析、エピジェネティクスなど、がん研究におけるさまざまなアプリケーションを探ります。
がん研究のためのゲノミクス製品
研究者ががんのゲノム変化を同定するのに役立つキットと試薬。
参考文献
- Caushi JX, Zhang J, Ji Z, et al. Transcriptional programs of neoantigen-specific TIL in anti-PD-1-treated lung cancers. Nature. 2021;596(7870):126-132. doi:10.1038/s41586-021-03752-4