NGS用語ガイド
シーケンスプロジェクトを計画する際に、重要な用語の定義を確認し重要な概念を明確にするのに役立つ次世代シーケンサー用語集です。
サンガーシーケンスとNGSの重要な違いはシーケンス量です。サンガー法では一度に1つのDNA断片のみをシーケンスしますが、NGSは極めて並行です。つまり、ランごとに数百万の断片を同時にシーケンスします。このプロセスにより、一度に数百から数千の遺伝子をシーケンスします。また、NGSには、ディープシーケンスで新規または希少なバリアントを検出するより高い検出力もあります。
一般的なNGS用語の用語集
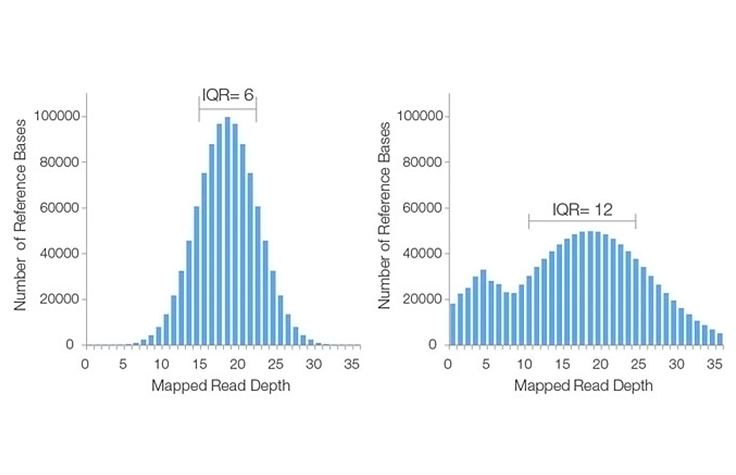
既知のリファレンス塩基にアラインする、または「カバー」するシーケンスされた塩基の平均数。例えば、30×カバレッジでシーケンスされた全ゲノムは、ゲノムの各塩基が平均30回シーケンスされたことを意味します。より高いカバレッジレベルでは、ベースコールはより高い信頼度で行うことができます。
詳細はこちら
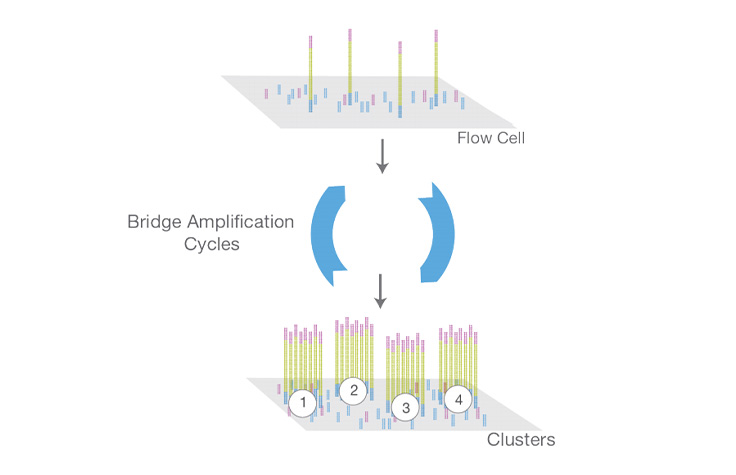
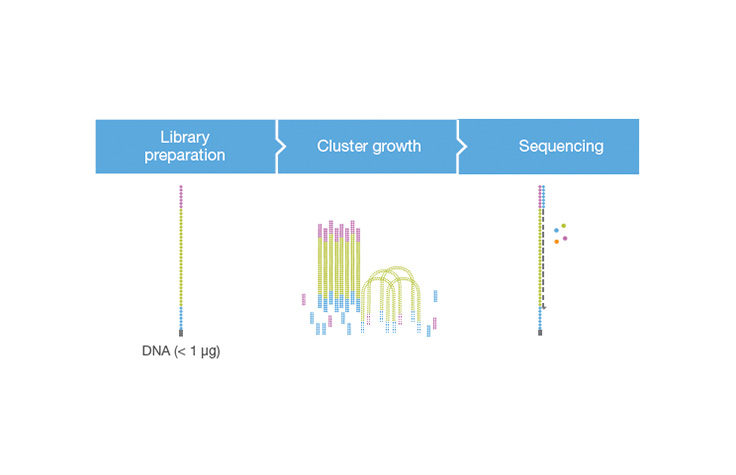
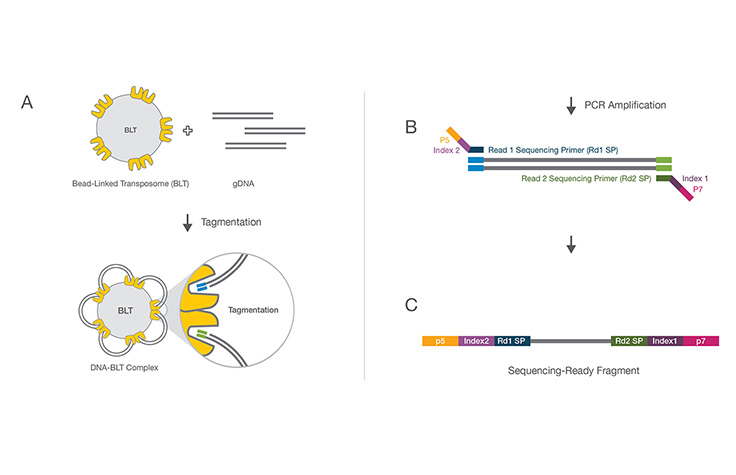
ゲノムDNAサンプル(またはcDNAサンプル)をシーケンスライブラリーに変換する分子生物学プロトコールで、NGS装置でシーケンスすることができます。ライブラリー調製の最初のステップは、DNAサンプルのランダムな断片化です。その後、各DNA断片に5’アダプターと3’アダプターをライゲーションします。あるいは、「タグメンテーション」により断片化反応とライゲーション反応を1つのステップにまとめ、ライブラリー調製プロセスの効率を大幅に向上させます。
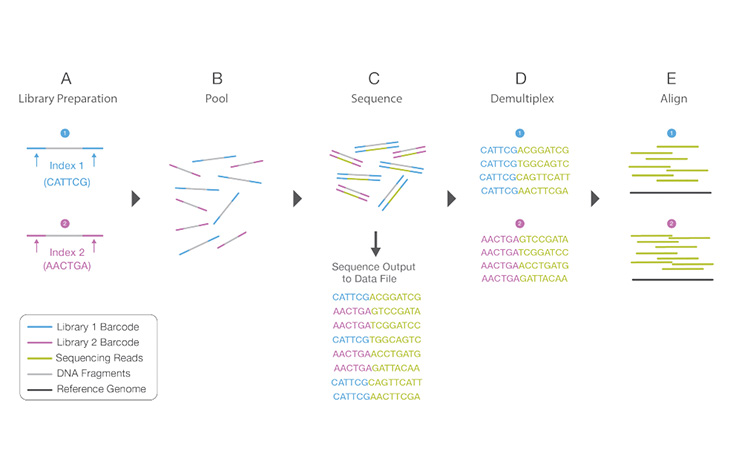
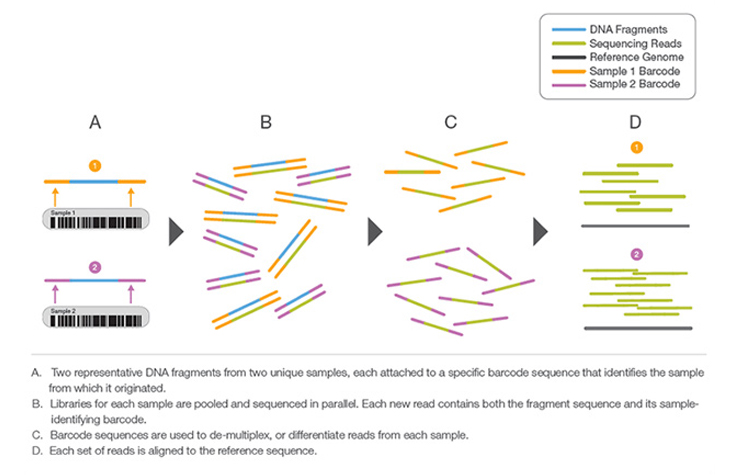
詳細はこちらライブラリー調製中に、固有の短いDNAシーケンス、すなわち「インデックス」を各DNA断片に添加するプロセス。ユニークなシーケンスにより、多くのライブラリーを一緒にプールし、同時にシーケンスすることができます。プールされたライブラリーからのシーケンスリードは、最終データ解析の前に同定され、計算的にソートされます。ライブラリーマルチプレックスは、小さなゲノムや関心のあるゲノム領域をターゲットにする場合に有用な手法です。マルチプレックスにより、コストやランタイムの大幅な増加なしに、1回のランで分析できるサンプル数を飛躍的に増やせます。
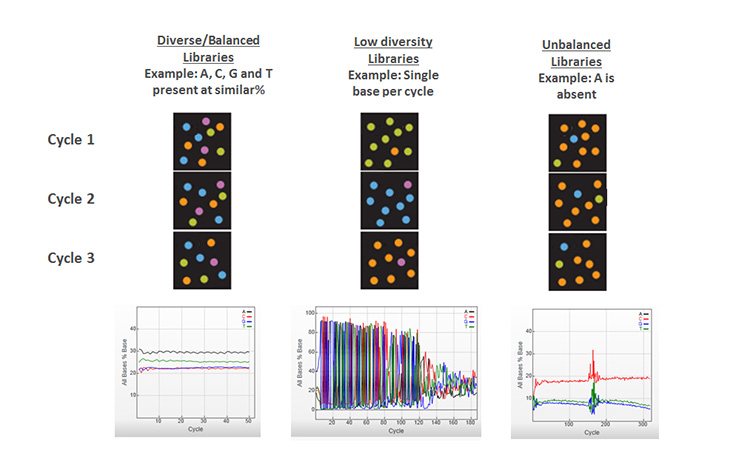
詳細はこちらシーケンスライブラリー内のすべてのDNA断片にわたる各塩基位置に、A、C、G、Tヌクレオチドの等比率を有すること。イルミナのシーケンスシステムで効果的なイメージ解析を行うには、カラーバランスが必要です。したがって、ほとんどのイルミナライブラリー調製ワークフローにはランダムな断片化ステップが含まれており、ライブラリーの各塩基位置で必要なシーケンスの多様性を生成します。
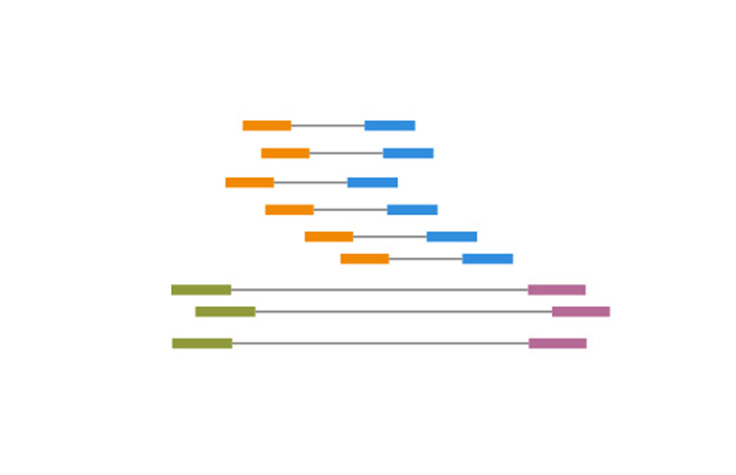
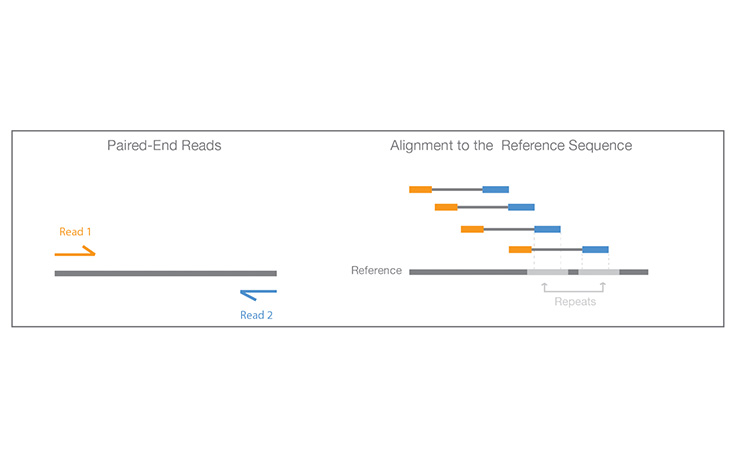
イルミナの知識同じランでDNA断片の両端からシーケンスするプロセス。
詳細はこちらNGSのメトリクスで、ベースコーリングにおけるエラーの確度を予測または推定します。クオリティスコア(Qスコア)は、非常に小さなエラー確率を伝達するコンパクトな方法です。高いQスコアは、ベースコールの信頼性が高く、不正確である可能性が低いことを意味します。
詳細はこちら1メガ塩基=1,000,000塩基。
1ギガ塩基=1,000,000,000塩基。
すべてのi5インデックスとすべてのi7インデックスが1回のみ使用されるようなインデックスのペア。ユニークデュアルインデックスにより、インデックスホッピングのリードを同定してフィルタリングできるため、マルチプレックスサンプルの信頼性が向上します。
記事を読むゲノムのタンパク質コード領域(エクソーム)のみをターゲットとする広く使用されているシーケンス法。
詳細はこちら
NGSの導入eBook
ご研究へのNGSの導入をお考えですか? 新しいテクノロジーの採用には不安がつきまとうものです。NGSをラボに導入するための包括的でわかりやすいガイドを作成しました。NGS手法、ワークフロー、データ解析ソリューション、追加の用語集用語について学び、NGSを始めるためのステップバイステップガイドを見つけることができます。
eBookをダウンロードする新着NGS eBookをダウンロードする
補足資料
初心者向けNGS
NGSについての詳細にご関心をお持ちですか? このページでは、次世代シーケンサーとその従来の手法に勝る多くの利点について、簡潔で明確にご説明します。
NGSチュートリアル
NGSの主な概念を理解するのに役立つチュートリアルを多数ご用意しています。ライブラリー調製、シーケンス、およびデータ解析のベストプラクティスについて、順を追って説明します。
オンライントレーニング
装置、ライブラリー調製キット、ソフトウェアなどのオンデマンドトレーニングにアクセスできます。