Karyomapping
Karyomappingでより迅速な結果を得る
Karyomappingは、in vitro胚の遺伝子変異を検査するために使用される技術です。これは、ゲノム内の遺伝子マーカーを使用して、遺伝子突然変異を担持するin vitro胚の可能性を評価するものです。
単一遺伝子疾患を同定する代替法では、疾患に特異的な遺伝子座に隣接する短いタンデムリピート(STR)の遺伝を調べます。STR法は、各テストラン用に開発されたため、費用と時間がかかります。
当社のkaryomappingソリューションは、STR解析のより迅速な代替手段です。1 単一細胞からのゲノムワイドな連鎖ベースの解析を使用して、変異を含む遺伝子領域が胚に伝播されたかどうかを検出します。

Karyomappingの利点
Karyomapping法は、in vitro胚染色体の完全なマップを作成することができ、単一遺伝子欠損の遺伝におけるゲノミクスの役割に関する貴重な洞察を研究者に提供します。
Karyomapping解析では、ゲノムワイドな連鎖を使用して、片方または両方の親に存在する遺伝性疾患遺伝子座の遺伝を明らかにします。マーカーとして関心遺伝子に隣接するSTRの代わりに、karyomappingは一塩基多型(SNP)を使用します。SNPは、ゲノム内の一塩基が個人間で異なる場合に発生するDNAシーケンスのバリエーションです。既存のゲノムワイドSNPテクノロジーを使用して容易に同定できます。karyomappingでは、in vitro胚のSNPとリファレンスのSNPを比較し、胚が変異遺伝子を遺伝する可能性を確立します。
Karyomappingは、STR解析よりも以下のような多くの利点があります。
- SNPはゲノム全体にかなり均等に分布しています。
- HumanKaryomap-12 BeadChipは多数のデータポイントを提供し、関心のあるゲノム領域で組み換えイベントを見逃すリスクを低減します。
- Karyomappingでは、遺伝性単一遺伝子障害ごとにプローブをカスタムデザインする必要がないため、時間を大幅に節約できます。
- Karyomappingは、一貫した結果を得るためにソフトウェアを使用した自動解析に適しています。
STR解析とKaryomappingの比較
| STR解析 | Karyomapping | |
|---|---|---|
| 説明 | 特定の遺伝子座に隣接する2~6塩基対のDNAリピートを使用して、欠陥遺伝子を同定します |
SNPとゲノムワイドな連鎖データを使用して、欠陥遺伝子の有無を知らせます |
| マーカー | マルチアレル、リピート長の変動を測定 |
両アレル、単一塩基で変動を測定 |
| カバレッジ | STRマーカーの各セットの単一遺伝子座に限定 |
複数の遺伝子座を並行してスクリーニングできる |
| 場所 | 影響を受けた遺伝子の位置に関する知識が必要 |
影響を受けた遺伝子の位置に関する知識が必要 |
| 調製時間 | 通常、複数のSTRマーカーの精密検査と検証に3~6か月かかります |
なし、市販のソリューション |
| ワークフロー | 症例ごとにカスタマイズされたプライマーセット |
すべての研究の標準ワークフロー |
| リンケージ解析 | 手動で実施 |
BlueFuseMulti解析ソフトウェアを使用した自動データ解釈 |
| 異数性 | 利用不可 |
現在提供されていない |
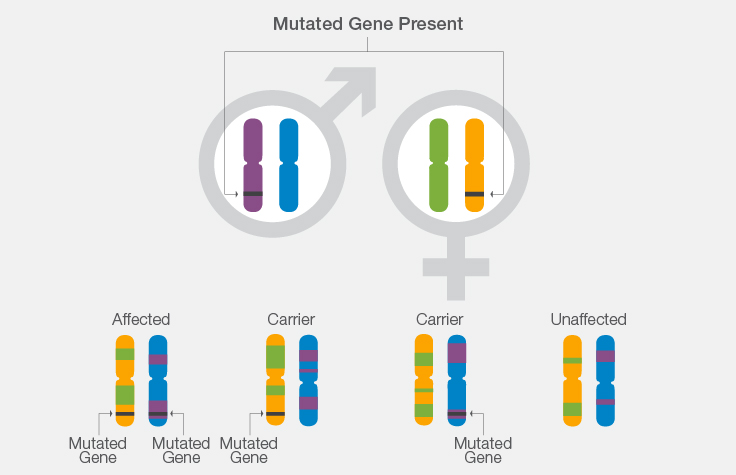
Karyomappingの仕組み:嚢胞性線維症の例
- 両親が嚢胞性線維症の変異を持っている場合、その子が病気になる確率は4分の1です。
- 親のCFキャリアや疾患状態に関する知識を用いて、遺伝子が属する染色体セグメントを調べ、どの親染色体から遺伝子が由来しているかを判断することができます。
- サイエンティストはその後、各in vitro胚によって受け継がれた染色体セグメントに、CF遺伝子の正常コピーまたは変異コピーが含まれているかどうかを判断できます。
- これは、in vitro胚が親のいずれかから単一の変異を遺伝し、影響を受けない保因者になるかどうか、in vitro胚が各親から変異を遺伝し、CFに影響を受けるかどうか、in vitro胚が変異を遺伝しなかったかどうかを決定するのに役立ちます。
Karyomapping製品
イルミナは、以下を含む包括的なkaryomappingソリューションを提供します。
Infinium HumanKaryomap-12 DNA Analysis Kit
このBeadChipアレイは、ゲノム全体のカバレッジを効率化するために、ゲノム内の最も情報の多い約300,000のマーカーをターゲットとしています。Karyomappingは、ゲノム内のバイオマーカーを使用して、単一遺伝子障害に関与する遺伝子の変異バージョンを持つin vitro胚の可能性を評価します。
NextSeq 550システム
全ゲノム、エクソーム、トランスクリプトームの解析に必要な柔軟性とシンプルさを提供するデスクトップシーケンスシステム。最適化されたシーケンス試薬とクラスター化ケミストリーにより、イルミナに期待されるデータ品質が得られます。
BlueFuse Multi Software
明確で直感的なユーザーインターフェース、共通のワークフロー、ラボのニーズに合わせた拡張性を提供します。この広く実装されているソフトウェアは、PGSを実施するほとんどのラボで使用されています。
参考文献
- American Society for Reproductive Medicine. Frequently asked questions about infertility. ReproductiveFacts.Org Website Accessed April 4, 2016.