mRNAシーケンス
mRNAシーケンスの概要
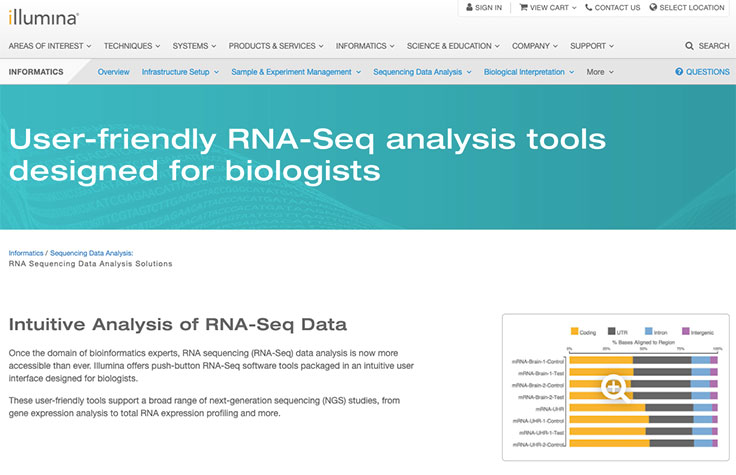
mRNAシーケンス(mRNA-Seq)は、疾患状態や生物学的プロセスのトランスクリプトームの解析メソッドとして、また広範な研究デザインにおいて、急速に注目を集めています。mRNA-Seqは、遺伝子発現を定量化する高感度で正確な手段であることに加え、既知および新規の転写物アイソフォーム、融合遺伝子、その他の特徴や、アリル特異的発現を同定することが可能です。mRNA-Seqは既知情報のみのフィルターによって狭められない、コーディングトランスクリプトームの詳細な情報を提供します。
シングルセルmRNA-Seq
Norma Neff博士が、スタンフォード大学の研究者が、どのようにシングルセルmRNA-Seqを使用して早期開発を理解しているかについて語ります。
インタビューはこちらmRNAシーケンスの利点
mRNA-Seqには、トランスクリプトームの解析において、遺伝子発現アレイと比較して多くの利点があります
- より広いダイナミックレンジを提供し、遺伝子発現の倍率変化をより高感度かつ高精度に測定
- 既知の特徴だけでなく、新規の特徴もキャプチャー
- 幅広い生物種に適用可能
トランスクリプトームの正確で高解像度な情報
比率圧縮は遺伝子発現アレイのよく知られた技術的制限であり、ダイナミックレンジを減少させ、測定された転写変化をマスキングまたは変化させることがあります1–3。一方、mRNA-Seqはこのバイアスに左右されず、より包括的で正確な遺伝子発現変化の測定が可能です。
さらに、mRNA-Seqではストランドの情報を得ることができるため、アンチセンス発現の検出が可能になり、オーバーラップする転写物をより正確に定量化でき、アライメント可能なリードの割合が増加します。
自閉症とmRNA-Seq
SynapDx社の社長、CEO、創設者であるStanley Lapidus氏が、同社がどのようにmRNA-Seqを利用して自閉症を研究しているかについて論じています。
動画を視聴標準サンプルに対する推奨mRNA-Seqワークフロー
ライブラリー調製
Illumina Stranded mRNA Prep
インプット量わずか25 ngの標準(未分解)RNAを利用してコーディングトランスクリプトームを解析できるシンプルで拡張性が高くコスト効率の良い、1日で完了できる迅速ソリューション
シーケンス
NextSeq 1000および2000システム
これらの費用対効果が高く、ユーザーフレンドリーなミッドスループットのベンチトップシーケンサーは、mRNA-Seqに加えて、現在および将来のさまざまなアプリケーションにも対応しています。
MiSeq i100 Series
mRNAシーケンスのための最速で最もシンプルなベンチトップシステム。
データ解析
DRAGEN RNAパイプライン
アライメント、定量化、および融合遺伝子検出を行います。
RNA-Seq Differential Expression
差次的な遺伝子発現解析が可能です。

RNAシーケンスメソッドガイド
このガイドでは、ターゲットパネルからmRNA、全トランスクリプトームまで、RNAプロファイリングのためのイルミナのソリューションを提供しています。イルミナのRNAシーケンスワークフローは、ライブラリー調製、シーケンシング、データ解析をシームレスに統合し、トランスクリプトーム研究をサポートします。
ガイドをダウンロードRNAライブラリー調製
RNA-Seqライブラリー調製の進歩は、トランスクリプトーム研究に革命をもたらしています。当社の強化されたRNAシーケンスライブラリー調製ポートフォリオは、mRNA-SeqからトータルRNA-Seqまで、複数のタイプのシーケンス研究に及びます。これらのソリューションは、迅速なターンアラウンドタイム、幅広い研究の柔軟性、およびシーケンスのスケーラビリティを可能にします。
RNAライブラリー調製の詳細はこちら包括的なmRNA-Seqのワークフロー
Sequence by Synthesis(SBS)ケミストリーは、最も広く採用されているNGSテクノロジーであり、世界のシーケンスデータの約90%を作成しています。*
イルミナは、業界をリードするデータ品質に加えて、ライブラリー調製からデータ解析、生物学的解釈までのプロセス全体を簡素化する統合型mRNA-Seqワークフローを提供します。
mRNAシーケンス特集記事

発生生物学におけるシングルセル解析
Dr. Colin Trapnellが、自身のラボにおけるシングルセルmRNA-Seqの実績と、バイオインフォマティクスツールを誰もが利用できるようにするための取り組みについて論じます。
インタビューを読む関連ソリューション
がん研究におけるRNA-Seq
mRNA-Seqで遺伝子発現の変化をモニタリングすることにより、研究者は疾患の予後や治療への反応性を予測するバイオマーカーの同定が可能です。がんRNA-Seqについてもっと知る。

疾患研究のための遺伝子発現解析
RNA-Seqを用いた遺伝子発現プロファイリング研究により、遺伝的および環境的要因がどのように広範な疾患に関与するかを視覚化することができます。遺伝子発現プロファイルについてもっと知る。

RNA薬物反応バイオマーカーの探索
RNA-Seqを使って、新規のRNAに基づく薬剤応答性バイオマーカーを同定する方法をご紹介します。新規ユーザーがこのアプリケーションを使用するためのリソースにアクセスできます。薬剤応答性RNAバイオマーカー分析についてもっと知る。
シーケンス手法に関する最新のニュース、研究事例などの情報を電子メールにてご提供します。ぜひご登録ください。
電子メールアドレスをご入力の上、ご登録ください。
Additional Resources
参考文献
- Shi L, Tong W, Su Z, et al. Microarray scanner calibration curves: characteristics and implications. BMC Bioinformatics. 2005;6 Suppl 2:S11.
- Naef F, Socci ND, Magnasco M. A study of accuracy and precisions in oligonucleotide arrays: extracting more signal at large concentrations. Bioinformatics. 2003;19:178-184.
- Yuen T, Wurmbach E, Pfeffer RL, Ebersole BJ, Sealfon SC. Accuracy and calibration of commercial oligonucleotide and custom cDNA microarrays. Nucleic Acids Res. 2002;30:e48.
*社内計算データ Illumina, Inc., 2015