CRISPRゲノム編集とNGS
CRISPRゲノム編集の応用
CRISPR-Cas9技術は、基礎研究、臨床研究、薬物療法、創薬、農業、および環境の分野で応用されていることが分かっています。臨床研究では、がん、AIDS、ハンチントン病、デュシェンヌ型筋ジストロフィーなどの疾患においてCRISPRを利用できる可能性が示されています。
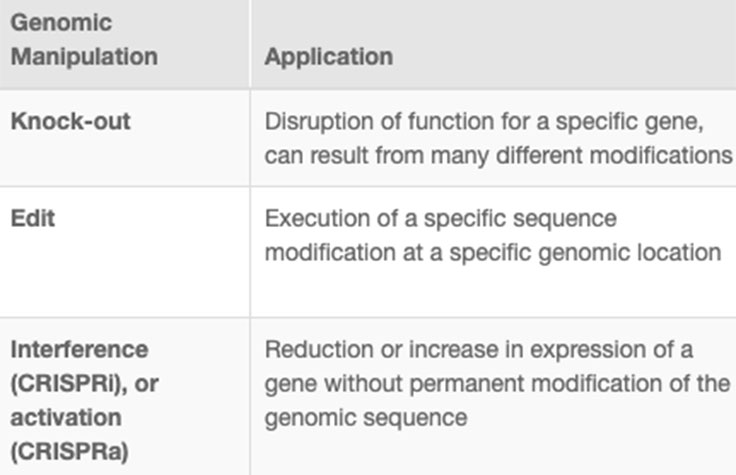
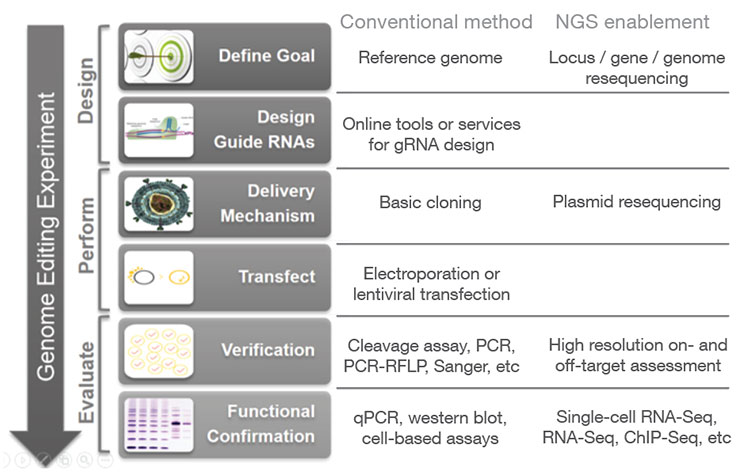
CRISPRゲノム編集により、研究者は遺伝子改変細胞株や動物モデルを迅速かつ精確に作製することができます。遺伝子ノックアウトの作製や特異性の高い改変の他にも、研究者はCRISPR技術によりゲノム配列を変化させずに阻害(CRISPRi)や活性化(CRISPRa)を介して遺伝子発現を調節することができます(表)。
CRISPRノックアウトとその他の編集の確認
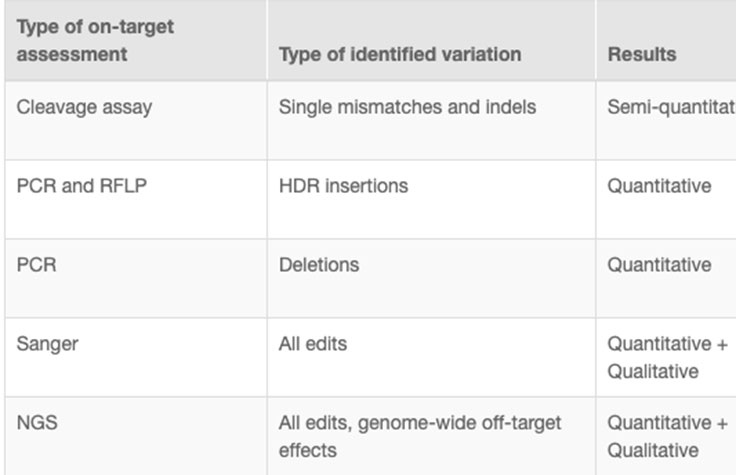
CRISPRゲノム編集実験からは細胞集団が混じり合ったものが得られ、その中の小さなサブセットのみで目的の編集がされています。研究者は、どの細胞に目的のCRISPRノックアウトや標的変異があるのかを判断する必要があります。編集を評価する現行の手法には、切断アッセイ、PCR、サンガー法、およびNGSなどがあります(表)。
NGSは、全ての種類の改変にわたり高解像度で定性的情報も定量的情報も得ることができ、あらゆるスループットのニーズに対応でき、オフターゲット効果のモニタリングにも使用できる唯一の方法です。7 NGSを用いるターゲットシーケンスは、改変の標的領域に注目することでCRISPRによる編集を確認できるコスト効率の高いソリューションです。
ターゲットシーケンスの詳細はこちらNGSを用いたCRISPRオフターゲット解析
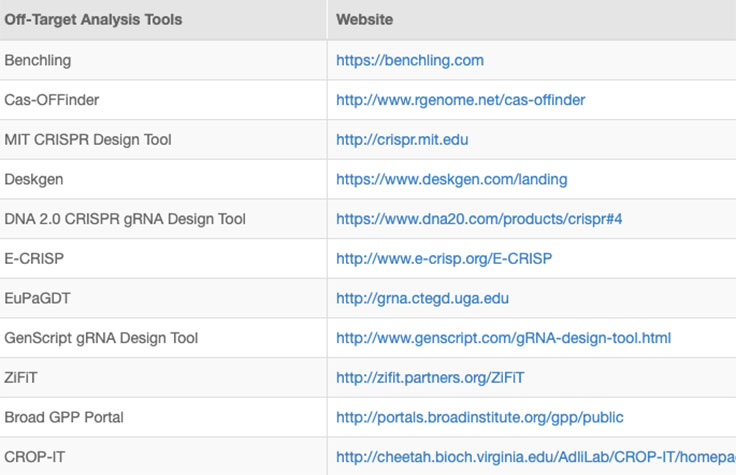
CRISP/Cas9技術の実施を成功させるには、目的のターゲット以外の部位における意図しない改変であるオフターゲット効果を特定し、低減するストラテジーなどが必要となります。ゲノム編集実験では、RNAの特異性を評価し、オフターゲット部位を予測する計算法が広く使用されています。
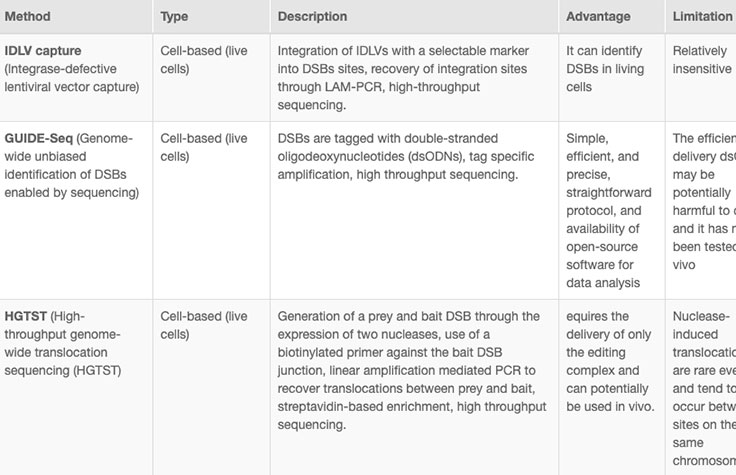
オンラインツールやウェブベースのアルゴリズムが公開されています(表)。ただし、予測アルゴリズムでは見逃されるおそれのあるオフターゲット部位を発見するには、NGSによる全ゲノムシーケンス(WGS)などの全ゲノム解析が必要です。8
WGSの詳細はこちらCRISPRゲノム編集による機能的影響
シングルセルRNAシーケンス
CRISPR改変後に細胞集団をスクリーニングして、何千もの個々の細胞において同時に多数の遺伝子の遺伝子制御の影響を判定
RNAシーケンス
トランスクリプトーム全体または遺伝子/遺伝子ファミリーの発現に対する変異の影響を評価
ChIP-Seq
DNAとタンパク質の結合に対する変異の影響を判定
メチル化シーケンス
メチル化状態およびクロマチンリモデリングに対する変異の下流影響を調査
主な製品
Francis deSouzaによるゲノミクス、CRISPR、未来
Illumina, Inc.の社長兼CEOであるFrancis deSouzaが、Aspen Ideas: コロラド州の健康は、医療全体におけるゲノミクスの影響と、より多くの人々にその利益をより迅速に提供することの緊急性について聴衆を教育します。
記事を読む
参考文献
- Cong L, Ran F A, Cox D, et al.Multiplex genome engineering using CRISPR/Cas systems.Science.2013;339:819-823.
- Mali P, Yang L, Esvelt KM, et al.RNA-guided human genome engineering via Cas9.Science.2013;339:823-826.
- Maruyama T, Dougan SK, Truttmann MC, Bilate AM, Ingram JR, Ploegh HL.Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining.Nat Biotechnol.2015;33:538-542.
- Chu VT, Weber T, Wefers B, et al.Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells.Nat Biotechnol.2015;33:543-548.
- Qi LS, Larson MH, Gilbert LA, et al.Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell.2013;152:1173-1183.
- Cheng AW, Wang H, Yang H, et al.Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system.Cell Res.2013;23:1163-1171.
- Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F. Genome engineering using the CRISPR-Cas9 system.Nat Protoc.2013;8:2281-2308.
- Tsai SQ, Joung JK.Defining and improving the genome-wide specificities of CRISPR-Cas9 nucleases.Nat Rev Genet.2016;17:300-312.
- Gabriel R, Lombardo A, Arens A, et al.An unbiased genome-wide analysis of zinc-finger nuclease specificity.Nat Biotechnol.2011;29:816-823.
- Tsai SQ, Zheng Z, Nguyen NT, et al.GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases.Nat Biotechnol.2015;33:187-197.
- Chiarle R, Zhang Y, Frock RL, et al.Genome-wide translocation sequencing reveals mechanisms of chromosome breaks and rearrangements in B cells.Cell.2011;147:107-119.
- Crosetto N, Mitra A, Silva MJ, et al.Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing.Nat Methods.2013;10:361-365.
- Kim D, Kim S, Kim S, Park J, Kim JS.Genome-wide target specificities of CRISPR-Cas9 nucleases revealed by multiplex Digenome-seq. Genome Res.2016;26:406-415.
- Tsai SQ, Nguyen NT, Malagon-Lopez J, Topkar VV, Aryee MJ, Joung JK.CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets.Nat Methods.2017;14:607-614.