
DNAメチル化についてのeBook
DNAメチル化の理解は、ヒト疾患やその他の表現型の研究に不可欠です。このeBookでは、DNAメチル化の解析や、研究デザイン、サンプルタイプ、データ解析などのエピゲノムワイド関連解析の実施に関する考慮事項について説明します。
DNAメチル化についてのeBook
ご提供いただいた個人情報は、お客様へのサポート、サービス、および販売活動の目的にのみ使用させていただきます。
DNAメチル化シーケンス解析にNGSテクノロジーを使用したバイサルファイトシーケンシング
バイサルファイトシーケンシング(BS-Seq)または全ゲノムバイサルファイトシーケンシング(WGBS)は、ゲノムDNA中のメチル化シトシンを検出するための定評のあるプロトコールです。この方法では、ゲノムDNAを亜硫酸水素ナトリウムで処理し、シーケンスすることで、ゲノム内のメチル化シトシンの単一塩基分解能が得られます。DNAを亜硫酸水素ナトリウムで処理すると、非メチル化シトシンはウラシルに脱アミノ化され、シーケンス後、チミンに変換されます。同時に、メチル化シトシンは脱アミノ化に抵抗し、シトシンとして読み取られます。メチル化シトシンの位置は、処理されたシーケンスと未処理のシーケンスを比較することで決定されます。1,2


DNAメチル化についてのeBook
DNAメチル化の理解は、ヒト疾患やその他の表現型の研究に不可欠です。このeBookでは、DNAメチル化の解析や、研究デザイン、サンプルタイプ、データ解析などのエピゲノムワイド関連解析の実施に関する考慮事項について説明します。
ご提供いただいた個人情報は、お客様へのサポート、サービス、および販売活動の目的にのみ使用させていただきます。

遺伝子発現や制御についてのバイオインフォマティクス(eBook)
遺伝子制御は、mRNAやノンコーディングRNA転写産物などの遺伝子産物の時間的および空間的発現を制御する生物学的プロセスです。BS-SeqやNGSテクノロジーなどの最新のトランスクリプトミクスやエピジェネティクスの手法が、この生物学的プロセスに関する発見や理解を深めるためにどのように使用されているかをご覧ください。
ご提供いただいた個人情報は、お客様へのサポート、サービス、および販売活動の目的にのみ使用させていただきます。

eBook『マルチオミクスのパワー』
DNAは本質的にマルチオミクス的であり、遺伝的塩基およびエピジェネティック塩基で分子情報が保存されています。RNA-Seqからのトランスクリプトーム情報をメチル化シーケンスデータと組み合わせることで、細胞の分化、メカニズム、および挙動をより良く理解することができます。さらに詳しく学び、マルチオミクスアプローチの力を解き放ちましょう。
ご提供いただいた個人情報は、お客様へのサポート、サービス、および販売活動の目的にのみ使用させていただきます。
低発現バイサルファイトシーケンシング(RRBS-Seq)またはシングルセル低発現バイサルファイトシーケンシング(scRRBS)は、ゲノムDNA上の1つまたは複数の制限酵素を使用してシーケンス特異的な断片化を生成するプロトコールです。次に、断片化されたゲノムDNAを亜硫酸水素塩で処理し、シーケンスします。RRBS-Seqは、特定の関心領域を研究するための理想的な方法です。プロモーターやリピート領域など、メチル化が多い場所では特に効果的です。3,4
酸化的バイサルファイトシーケンシング(oxBS-Seq)は、5mCと5hmCを区別します。oxBSでは、5hmCは酸化剤で5-ホルミルシトシン(5fC)に酸化されますが、5mCは変化しません。酸化した5hmCの亜硫酸水素ナトリウム処理により、ウラシルへの脱アミノ化が生じ、シーケンス時にチミンとして読み取られます。oxBSで処理したDNAのディープシーケンスと、処理したDNAと未処理DNAのシーケンス比較により、塩基分解能で5mCの位置を特定できます。5,6
タグメンテーションベースの全ゲノムバイサルファイトシーケンシング(T-WGBS)は、Tn5トランスポゼースとバイサルファイト変換を使用して5mCを研究するプロトコールです。標準的なWGBS法と比較して、T-WGBSは最小限のDNA(約20 ng)で材料をシーケンスできます。さらに、Tn5トランスポゼースは、DNA断片化とシーケンスアダプターの装着をワンステップで実現します。その後、DNAは亜硫酸水素ナトリウムで処理され、PCR増幅され、シーケンスされます。7
シングルセルバイサルファイトシーケンシング(scBS-Seq)は、確立されたバイサルファイトシーケンシング(BS-Seq)とポストバイサルファイトアダプタータグ(PBAT)プロトコルのバージョンで、シングルセルからゲノムDNAのメチル化シトシンを検出するために修正されています。この方法では、単一細胞を単離した後、ゲノムDNAを亜硫酸水素ナトリウムで処理し、DNAを断片化します。その後、変換されたDNAはランダムプライミングを数回受け、シーケンスのためにPCR増幅されます。ディープシーケンスは、単一細胞からのメチル化シトシンについて単一ヌクレオチド単位の高分解能を提供します。8
| 方法 | バイサルファイトシーケンシングの利点 |
バイサルファイトシーケンシングのデメリット |
|---|---|---|
| BS-Seq |
|
|
| RRBS-Seq / scRRBS |
|
|
| oxBS-Seq |
|
|
| T-WGBS |
|
|
バイサルファイトシーケンシングは、シーケンスデータで非メチル化CをTに変換するためにバイサルファイトを使用するDNAメチル化を検出するために一般的に使用される方法です。ただし、この方法にはいくつかの制限があります。バイサルファイト変換後、ゲノム内のCpG部位の約10%はアライメントが困難であると推定され、90%のレベルに達する可能性のあるDNA分解があります。9
バイサルファイトシーケンシングに代わるイルミナ5塩基ソリューションは、新しい化学と解析を活用して、遺伝子バリアントとメチル化の同時検出を可能にします。イルミナ5塩基ケミストリーは、DNAを損傷しないシンプルなシングルステップで5mCのみをTに直接変換するため、ライブラリーの複雑さが保持されます。イルミナ5塩基ソリューションは、非修飾塩基(A、T、G、C)と5mCを単一アッセイで読み取ることができます。
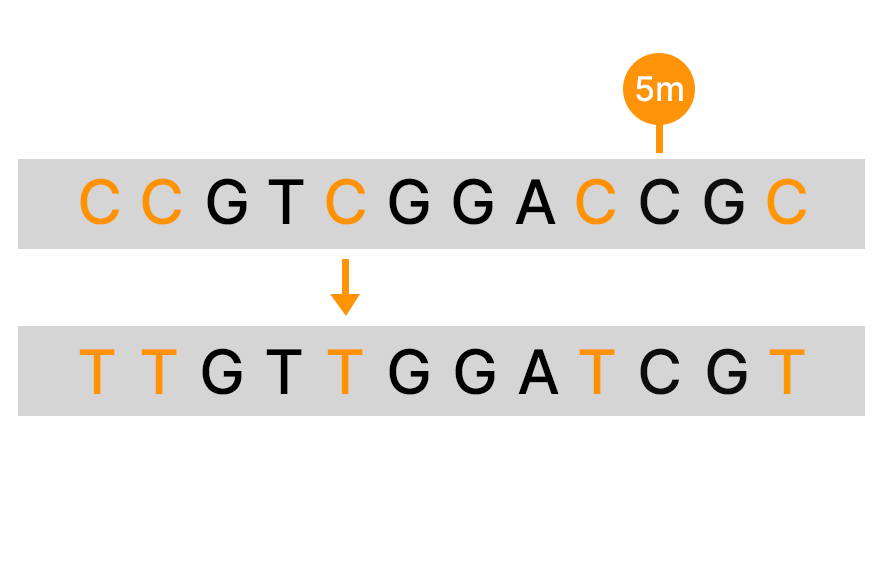
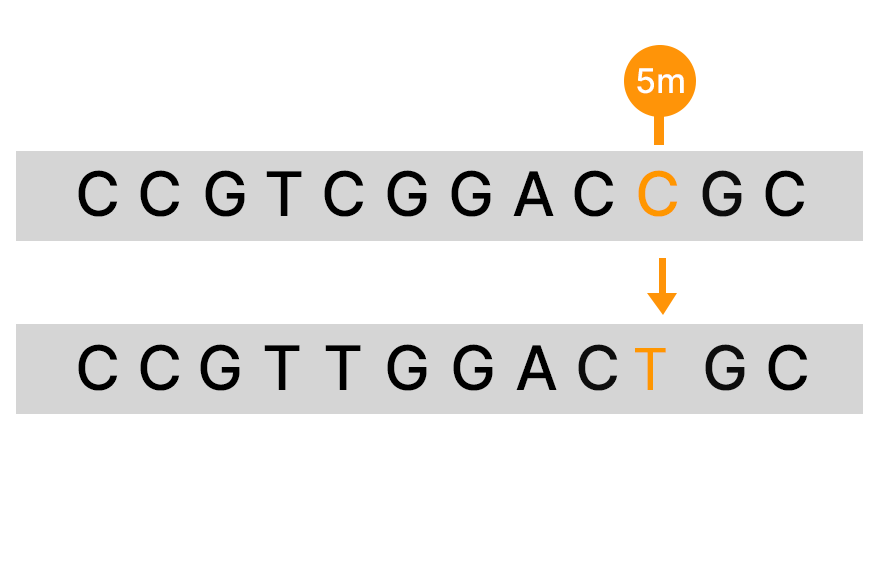
バイサルファイト変換は、メチル化シトシンが変換されないまま、亜硫酸水素ナトリウムが非メチル化シトシンをウラシルに選択的に変換するプロセスです。バイサルファイト処理は、ゲノムワイドなDNAメチル化プロファイリングを可能にするために、NGSと並行して一般的に使用されています。NGSの基礎についてさらに詳しく知る。
バイサルファイトシーケンシングは、DNAメチル化の研究に一般的に使用される手法です。シーケンスの前に、非メチル化シトシンは亜硫酸水素ナトリウムで処理することでウラシルに変換されます。次に、亜硫酸水素塩処理サンプルと未処理サンプルからのシーケンスDNAを比較して、メチル化シトシンと非メチル化シトシンの位置を特定します。
DNAを亜硫酸水素ナトリウムで処理すると、非メチル化シトシンが化学脱アミノ化によってウラシルに変換されますが、メチル化シトシンはこのプロセスから保護されます。
バイサルファイト変換プロセスとその後のシーケンスには、DNAの分解、ゲノムの複雑性の低下、アライメントプロセスに伴うさらなる複雑性など、いくつかの限界があります。10
ターゲットBS-Seqは、バイオマーカー探索での使用が研究されており、ビオチン化RNAプローブなどのコンポーネントを使用して、目的の特定のゲノム断片を捕捉します。一方で、WGBSはゲノム全体のシーケンスと解析を行います。11
参考文献
メチル化研究のソリューションの詳細については、専門家にご相談ください。
ご提供いただいた個人情報は、お客様へのサポート、サービス、および販売活動の目的にのみ使用させていただきます。